利用深度学习预测药物测试的多类细胞核表型
引言
量化蛋白质的动态运动(如核受体对药物的反应)需要使用染料和染色剂的稳定细胞核分割方法。但这一过程具有明显的局限性:活细胞染色程序耗时、成本高昂,并可能导致光敏化合物(例如 R18811)的光毒性、光漂白和失活。
为了克服这些挑战,我们利用采用 TruAI 深度学习技术的 cellSens 成像软件开发了一个神经网络(NN),无需荧光标记即可识别细胞核。然后我们训练这个 NN 来区分不同的细胞表型对药物的反应。在这方面,我们应用 NN 工作流程来预测使用 AR 靶向药物治疗的活前列腺癌细胞内雄激素受体 (AR) 动态变化过程。
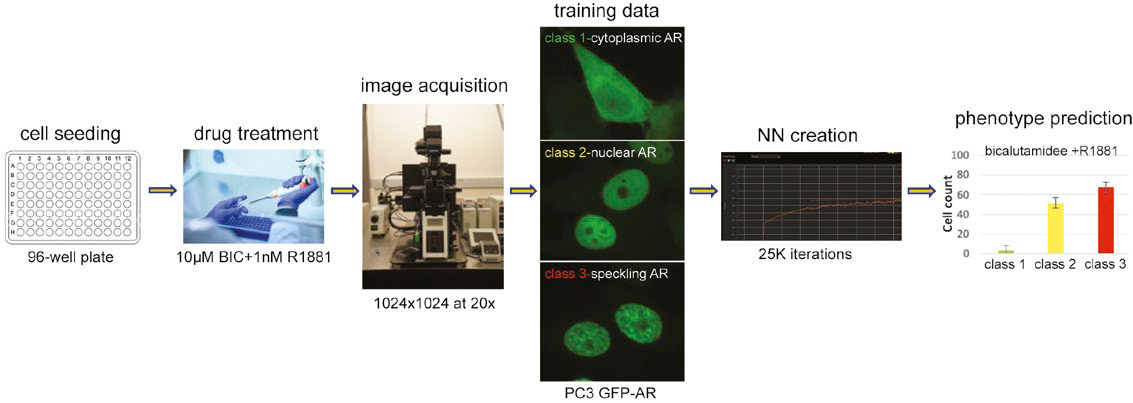
图 1:使用经过训练的 NN 预测多类细胞核表型的工作流程。
激活后,细胞质 AR 易位到细胞核,并定位到转录活性位点,这可以利用共聚焦显微术可视化为独特的散斑图案。为了预测 AR 动态变化过程,我们首先生成了一个神经网络,它可以在不使用核染剂的情况下探测细胞核。然后,我们根据 GFP-AR 核易位和反式激活(散斑)状态对细胞进行分类,这两种状态都是用于测试化合物靶向 AR 能力的必要读数。最后,我们证明了我们的 NN 工作流程能够在不进行核染色的情况下成功预测这些关键的 AR 表型。
好处
- 不利用荧光标记识别细胞核表型
- 节省花费在核染料(染色和成像)上的时间和成本
- 减少了光毒性和光漂白
- 保持光敏化合物的活性
方法
我们生成了一个训练数据集,由三种通常可以在药物反应中观察到的主要 AR 表型组成。我们使用了一半的图像生成神经网络,另一半用于评估其性能。最后,我们将我们的多类神经网络应用于一个未进行核染色的独立数据集。
来自三种 AR 表型的训练数据准备
作为构建神经网络的第一步,我们生成了由三种主要 AR 表型组成的训练数据集:
- 无活性细胞质 AR(空白对照,简称 NTC)
- 配体结合但无活性的核 AR(10 μM 比卡鲁胺)
- 活性核 AR 散斑 (1 nM R1881)
为了生成实际数据,我们使用 SiR-DNA 对细胞核进行了染色。我们使用奥林巴斯 FV3000 激光共聚焦显微镜的振镜扫描器,通过 UPLSAPO 20 倍物镜采集了 1024x1024 像素比例图像。在这里,我们将整个数据集(60 幅图像)分成两个相等的部分,分别代表训练数据集和验证数据集(各 30 幅图像)。为了训练神经网络,我们使用了各 10 幅分别来自三种 AR 表型的核染色最大强度投影 GFP-AR 图像(50% 的数据,总共 30 幅图像)。
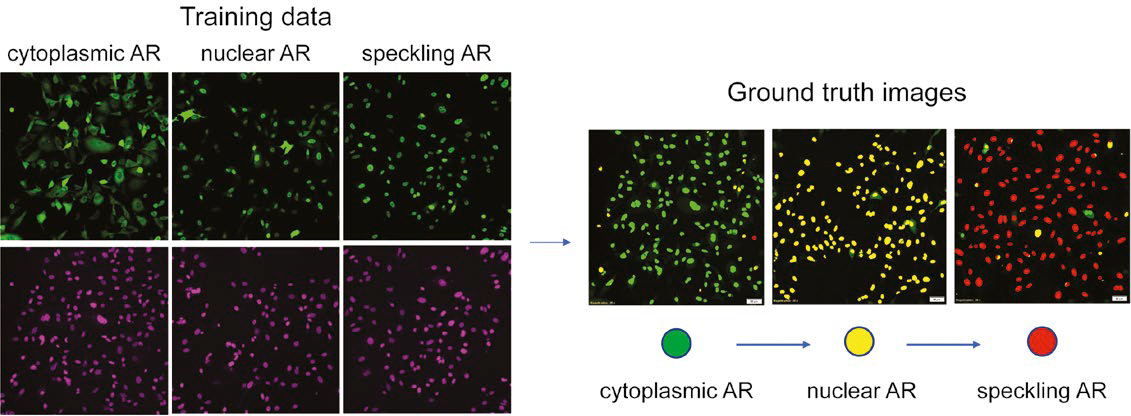
图 2:基于细胞核染色的多类 AR 表型训练数据的准备。(左)绿色:AR-GFP,紫色:SiR-DNA。(右)实际数据集的评估。
实际数据优化
图 2 显示了神经网络评估的实际数据,细胞质 AR 为绿色,核 AR 为黄色,散斑 AR 为红色。我们使用这种以色彩区分的分割图,通过手动校正核探测和分割中的错误以及重新分类归类错误的细胞核来微调实际数据。
使用 TruAI 深度学习技术创建神经网络
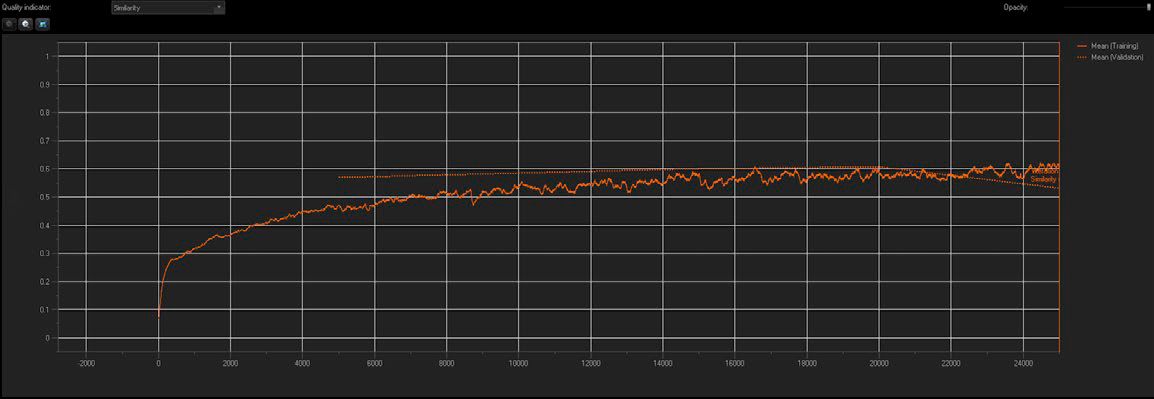
图 3:神经网络训练实时报告相似性指数和验证平均值。
我们利用 TruAI 技术创建了一个具有多类配置的标准神经网络,其中包含来自每个 AR 表型的 10 个训练图像。实时报告了神经网络的质量和准确性。如图 3 所示,在训练过程中,相似性指数随着每次连续迭代而增加,直到达到一个平稳期,这表明训练饱和(向下箭头)。每 5,000 次迭代(20% 进度)创建一次检查点,并将最佳检查点连同其相似性指数值一并保存为受过专门训练的 NN。
结果
基于盲数据的多类神经网络的验证
我们使用未用于实际或神经网络训练的图像中的 50% 验证了我们的多类 NN 模型。然后,我们使用这些验证结果,通过应用面积筛选器(最小值 49.83 μm – 最大值 555.44 μm)来消除细胞碎片和错误分割的细胞。受过专门训练的 NN 模型在没有任何核染色的情况下独立预测多类 AR 表型。图 4 显示了使用 10 幅图像的数据的表型类分布。
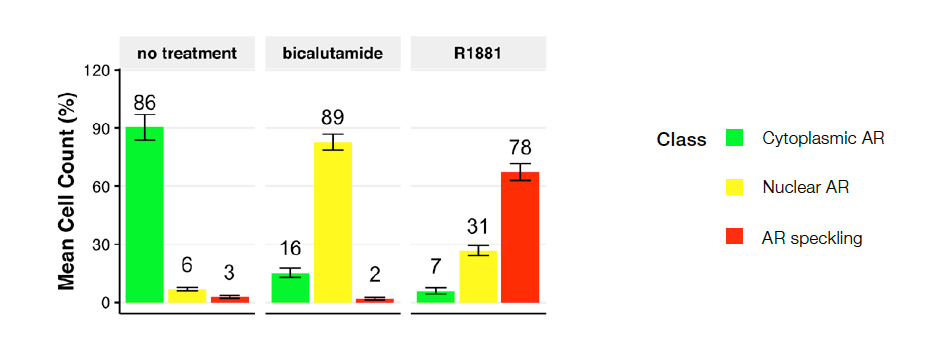
图 4:以下条件下的细胞类分布:空白对照(左)、比卡鲁胺(中)或 R1881(右)。该图反映了分类为 1 类(细胞质 AR)、2 类(核 AR)和 3 类(散斑 AR)的细胞所占百分比。空白对照产生的基本上是 1 类细胞。比卡鲁胺(AR 拮抗剂)诱导向 2 类的转变,R1881(AR 激动剂)诱导向 3 类的转变,活性 AR 局限在转录活性位点。
基于独立数据集的多类 AR 表型预测
接下来,为了评估独立图像上的多类 NN 模型,我们使用两种临床抗雄激素:比卡鲁胺和恩卡鲁胺准备了一个新的数据集(图 5)。
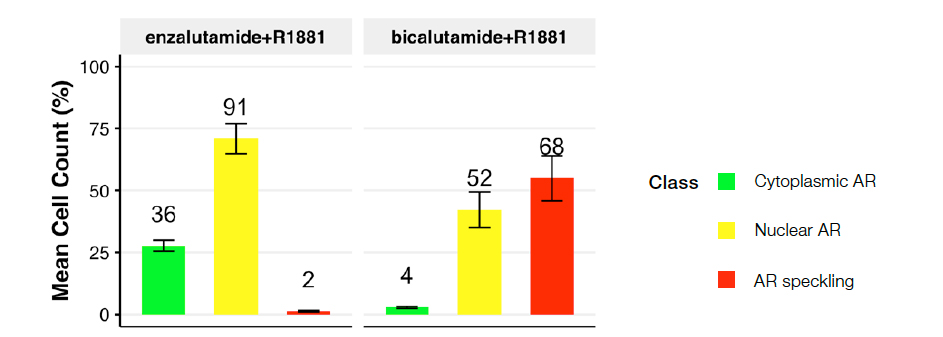
图 5:以下条件下的细胞类分布:R1881 加恩扎鲁胺(左)或比卡鲁胺(右)。该图反映了各种处理条件下预测的细胞核类分布。
用第二代抗雄激素恩扎鲁胺处理导致突出的细胞质 AR(2 类)细胞群,只有很少或没有任何散斑 AR(3 类),表明有效阻断了配体诱导的 AR 激活。相比之下,旧式、疗效较差的抗雄激素比卡鲁胺导致明显朝向活性 AR(3 类)的转变,表明其无法有效阻断 1nM R1881 诱导的 AR 激活。这些数据证实了先前的研究2。
结语
我们使用奥林巴斯 FV3000 共焦显微镜和具有 TruAI™ 深度学习模块的 cellSens™ 软件生成了一个 NN,用于探测和分割未染色的细胞核,以及根据 AR 对药物反应的表型变化对细胞进行详细分类。这种方法有可能通过减少测试时间(实验装置)和成本来提高药物测试效率。
此外,NN 可以通过减少诸如光敏化合物的光毒性、光漂白和失活等假象来提高数据质量,以及探测研究人员和/或传统图像分析途径可能忽略的细微变化。这种深度学习方法可以更广泛地应用于基于细胞核或基于细胞的参数(从蛋白质定位到形态学变化)的细胞类群详细分类。
参考文献
- Risek B, Bilski P, Rice AB, Schrader WT. Androgen receptor-mediated apoptosis is regulated by photoactivatable androgen receptor ligands. Mol Endocrinol. 2008 Sep;22(9):2099-115. doi: 10.1210/me.2007-0426. Epub 2008 Jun 18. PMID: 18562628; PMCID: PMC2631375.
- Sugawara T, Lejeune P, K hr S, Neuhaus R, Faus H, Gelato KA, Busemann M, Cleve A, Lücking U, von Nussbaum F, Brands M, Mumberg D, Jung K, Stephan C, Haendler B. BAY 1024767 blocks androgen receptor mutants found in castration-resistant prostate cancer patients. Oncotarget. 2016 Feb 2;7(5):6015-28. doi: 10.18632/oncotarget.6864. PMID: 26760770;PMCID: PMC4868737.
作者
- 加利福尼亚州洛杉矶南加利福尼亚大学 Lawrence J. Ellison 转化医学研究所的 Harish Sura、Katherin Patsch 和 Seungil Kim
- Olympus Corporation of the Americas 战略项目经理 Shohei Imamura
适于这类应用的产品
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
对不起,此内容在您的国家不适用。