O Hospital Nacional de Paraplégicos (HNP) em Toledo, na Espanha, é um centro de referência nacional espanhol para lesões na medula espinhal. Os objetivos do HNP são fornecer serviços abrangentes de saúde e reabilitação para pessoas com lesão medular, treinar pessoal qualificado e realizar pesquisas científicas no campo da neurociência.
Para apoiar a pesquisa em neurociência, o HNP fornece instalações essenciais para microscopia e análise de imagens, citometria de fluxo, proteômica, ressonância magnética nuclear (NMR) para pequenos animais e alojamento para animais. Sua instalação para microscopia e análise de imagem inclui uma poderosa solução multimodal que oferece suporte ao escaneamento de lâmina inteira e triagem de alto conteúdo (HCS) com um microscópio.
Esta solução combina o sistema de microscópio automatizado IXplore™ Pro com o software de triagem de alto conteúdo scanR* e o software de formação de imagem cellSens™. Ela pode se adaptar a uma ampla variedade de aplicações e técnicas de formação de imagem, incluindo análise de imagem avançada usando a tecnologia de aprendizagem profunda TruAI™.

Figura 1. Vista panorâmica do Hospital Nacional de Paraplégicos em Toledo, na Espanha. Foto cortesia de Juan Carlos Monroy.
A instalação de Microscopia e Análise de Imagem do Hospital Nacional de Paraplégicos é gerido pelo Dr. José Ángel Rodríguez Alfaro e pelo Dr. Javier Mazarío. Recentemente, conversamos com eles para saber mais sobre sua experiência com o sistema IXplore Pro com o software scanR e cellSens nos serviços centrais da instalação.
P: Por que vocês consideraram um sistema de microscópio IXplore com software scanR para sua instalação?
R: Em nossas instalações, temos uma variedade de microscópios ópticos, desde microscópios confocais de escaneamento a laser automatizados até microscópios manuais de rotina. Anteriormente, tínhamos um sistema de análise de alto conteúdo (HCA) de caixa fechada que quebrou após 10 anos de operação e precisávamos de um substituto.
Quando isso aconteceu, estávamos procurando maneiras de exportar os dados obtidos com esse sistema para um software de análise de citometria de fluxo para poder realizar uma análise mais refinada das medições obtidas com nosso sistema HCA.
A primeira coisa sobre o sistema scanR que chamou nossa atenção foi que esse tipo de análise fechada citométrica controlada já estava incorporada ao software. Não encontramos nenhum outro sistema HCA que podia realizar esse tipo de análise. Esse foi um dos principais fatores que nos levaram a escolher um sistema IXplore com o software scanR.
P: Quais outros recursos vocês gostaram no sistema?
R: Adoramos o fato de não ser um sistema de caixa fechada e com o software cellSens teríamos também um microscópio de campo amplo capaz de escanear lâminas. Esta configuração dois em um do sistema foi perfeita para nós, pois nos permitiu aumentar a variedade de serviços que nossas instalações oferecem.
A modularidade do sistema é muito conveniente. Graças a isso, poderemos adicionar mais funcionalidades quando precisarmos, como incorporar uma incubadora para estudos de células vivas.
Outro ponto importante foi a possibilidade de incorporar ao nosso portfólio uma tecnologia de ponta em análise de imagens com inteligência artificial, como o TruAI, que funciona não apenas com imagens cellSens e scanR, mas também com imagens adquiridas com outros sistemas de microscópio.
P: Qual foi o impacto nas suas instalações após a adição de um sistema IXplore? O que mudou?
R: O sistema IXplore combinado com o software scanR rapidamente se tornou o microscópio mais usado nas nossas instalações. O tempo de uso mais que dobra o uso de microscópios confocais, as "estrelas" anteriores da instalação.
Embora este seja um microscópio de epifluorescência, a resolução das imagens é boa o suficiente para que os pesquisadores considerem mudar de formação de imagem confocal para alguns dos seus estudos, principalmente quando a alta velocidade do sistema é levada em consideração e não tanto a resolução Z. Isso permite que eles capturem imagens que eram muito demoradas de adquirir usando nossos microscópios confocais de escaneamento a laser.
E não é apenas uma questão de aumento de velocidade, mas também a possibilidade de usar o suporte de 4 lâminas em combinação com o mapa de foco para configurar experimentos que podem ser adquiridos sem supervisão durante a noite.
A possibilidade de executar análises dinâmicas dos dados adquiridos com o software scanR é outra grande melhoria em comparação com nosso sistema HCA anterior. Assim que a captura da imagem é concluída, obtemos os resultados da análise e podemos passar para a próxima amostra.
A tecnologia de aprendizado profundo TruAI também provou ser uma ferramenta muito útil. A detecção de núcleos em amostras de cultura de células de alta densidade melhorou drasticamente quando comparada às técnicas de segmentação padrão, permitindo que os usuários obtenham dados mais precisos de seus experimentos.
P: Vocês podes fornecer um exemplo de uso do sistema IXplore com o software cellSens na sua instalação? O que os pesquisadores querem ver nas imagens?
R: Muitas vezes, adquirimos imagens de alta resolução de seções transversais ou horizontais da medula espinhal de camundongos, ratos ou até mesmo porcos, onde os pesquisadores procuram alterações nas populações de células gliais, na sobrevivência de neurônios ou na expressão de diferentes marcadores após lesão da medula espinhal (SCI), bem como para axônios que crescem no local da lesão.
Mas não ficamos restritos ao tecido da medula espinhal. Também realizamos escaneamentos de alta resolução de seções cerebrais. Neste exemplo, examinamos uma seção inteira de um cérebro de camundongo usando contraste de interferência diferencial (DIC) para procurar o local da expressão do gene repórter fluorescente mCherry.
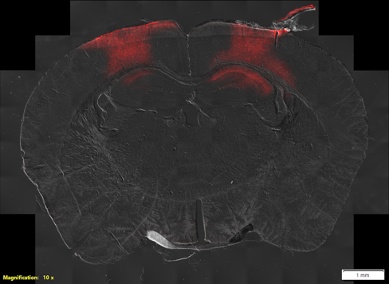
Figura 2. Imagem de lâmina inteira de um cérebro de camundongo usando DIC e fluorescência. Imagem cortesia do Grupo Experimental de Neurofisiologia e Circuitos Neurais do HNP. 14
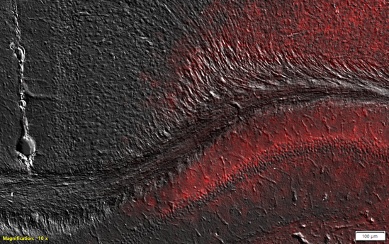
Figura 3. Visão aproximada de uma região da imagem anterior. Imagem cortesia do Grupo Experimental de Neurofisiologia e Circuitos Neurais do HNP. 18
P: Vocês podem fornecer um exemplo de uso do software scanR para HCA na sua instalação? O que os pesquisadores querem ver nos dados?
R: O HCA é muito usado para estudos de proliferação, diferenciação e migração celular em diferentes condições. Em nossas instalações, um dos ensaios mais frequentes que realizamos com o software scanR é a detecção da expressão de genes repórteres (GFP e mCherry) sob diferentes tratamentos. Combinamos o uso de TruAI para a segmentação de núcleos em culturas de células de alta densidade com análise fechada para quantificar o número de células com marcação simples e dupla na população e avaliar como funcionam os diferentes tratamentos.
Também usamos o sistema para a detecção de hibridização in situ fluorescente (FISH) em amostras de sangue e esperma. Neste caso, não usamos apenas o TruAI para a segmentação de núcleos, mas também para a detecção de pontos FISH dentro dos núcleos.
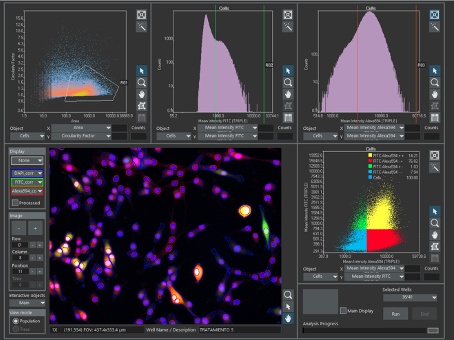
Figura 4. Janela principal do software scanR mostrando a segmentação de células e núcleos, circundada por gráficos de dispersão e histogramas. Nos gráficos de dispersão bidimensionais, cada ponto representa uma célula segmentada. Nos histogramas unidimensionais, cada linha representa um valor de parâmetro, correspondendo a altura da linha ao número de células com esse valor. No primeiro gráfico de dispersão, uma porta (R01) seleciona uma subpopulação de células de acordo com seu tamanho e forma e é transferida para os gráficos de análise seguintes. As células fora de R01 são descartadas. Nos segundo e terceiro gráficos de análise, são criados portas de células verdes positivas (R02) e células vermelhas positivas (R03). No gráfico de análise final, R01, R02 e R03 são combinados para obter a porcentagem de células positivas simples e duplas. Dados cortesia do Grupo de Neurologia Molecular do HNP. 22
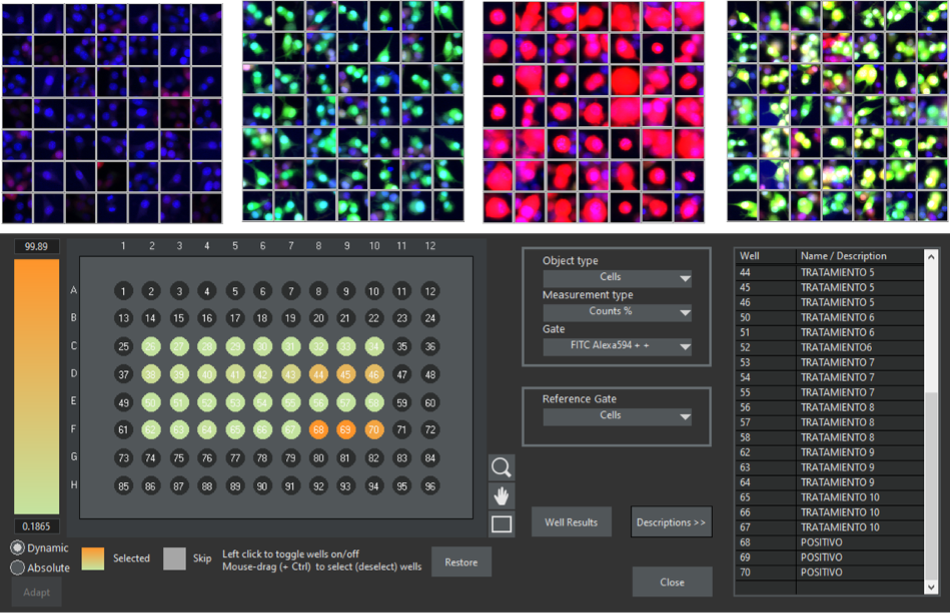
Figura 5. O software scanR permite que você crie galerias das regiões fechadas. Os exemplos mostram galerias para as populações de células: duplo negativo, único positivo verde, único positivo vermelho e duplo positivo verde-vermelho. Os resultados podem ser exportados para tabelas ou visualizados como um mapa de calor no contexto de uma placa de poços. Dados cortesia do Grupo de Neurologia Molecular do HNP.
P: Vocês usam a tecnologia TruAI para desenvolver seus próprios modelos de rede neural de aprendizado profundo?
A: Sim. Temos modelos treinados para segmentação de núcleos de células densamente compactadas, detecção de neurônios em seções da medula espinhal, classificação de diferentes tipos de fibras musculares, reconhecimento de núcleos anormais após determinados tratamentos, etc.
Esta é uma das coisas que mais gostamos no software, a possibilidade de treinar facilmente modelos de redes neurais, não só a partir de imagens adquiridas com este sistema, mas também de imagens obtidas com microscópios que não sejam da Olympus. E o fato de agora podermos usar o OlyVIA, um aplicativo gratuito de visualização, para anotar imagens o torna ainda mais conveniente, já que os usuários podem fazer isso dos seus próprios computadores no escritório ou até mesmo em casa.
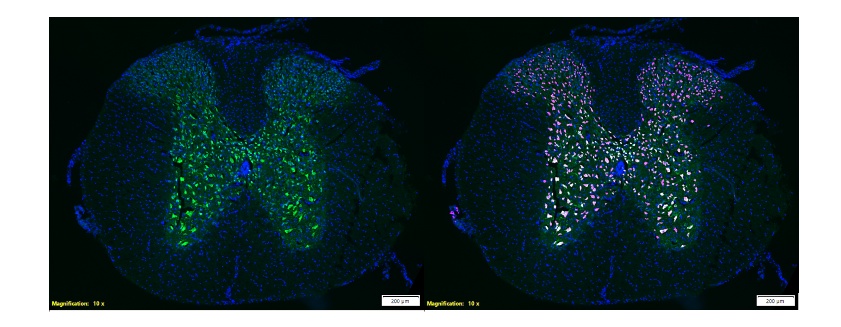
Figura 6. A imagem à esquerda mostra o marcador neuronal NeuN (verde) de uma seção da medula espinhal de um camundongo. A imagem à direita mostra a mesma seção com uma camada mostrando a detecção TruAI de células NeuN positivas (rosa). Imagens cortesia do Grupo de Neuroproteção Molecular do HNP. 28
P: O que vocês mais gostam no sistema?
R: Como mencionamos anteriormente, há muitas coisas de que gostamos neste sistema. Talvez o mais notável de todos seja que, com a combinação do software cellSens e scanR, podemos passar de escaneamento de toda a lâmina para o HCA com apenas um sistema de microscópio. Esta configuração dois-em-um faz todo o sentido para a otimização dos recursos nas nossas instalações. E, claro, o TruAI, que é totalmente compatível com os dois sistemas. Isso nos permite usar modelos de rede neural em ambos os softwares, independentemente de qual foi usado para o treinamento.
Saiba mais sobre os sistemas de microscópio IXplore
A série de microscópios IXplore refere-se a microscópios invertidos adaptados para diferentes aplicações de pesquisa em ciências da vida. Os microscópios invertidos motorizados IXplore Pro incluem recursos de campo claro, fluorescência multicanal, empilhamento em Z e união de imagens.
O sistema IXplore é controlado com o software cellSens, a plataforma para microscópios baseados em câmeras da Olympus que podem se adaptar a uma ampla variedade de aplicações e técnicas de formação de imagem em microscopia de campo amplo. Isso inclui microscopia de rotina, confocal de disco giratório, super resolução, fluorescência de reflexão interna total (TIRF), recuperação de fluorescência após fotobranqueamento (FRAP) e muito mais.
Com o hardware selecionado, o mesmo microscópio IXplore também pode ser controlado com o software scanR, que é dedicado e simplificado para aplicações de triagem de alto conteúdo em placas de poços e lâminas de câmara.
Os entrevistados
Obrigado ao Dr. José Ángel Rodríguez Alfaro e Dr. Javier Mazarío, gerentes da Central de Microscopia e Análise de Imagem do Hospital Nacional de Paraplégicos, na Espanha, por compartilharem suas experiências nesta entrevista. Para esclarecer qualquer dúvida sobre a instalação da Espanha, envie um e-mail para microscopia.hnp@sescam.jccm.es.

Dr. José Ángel Rodríguez Alfaro e Dr. Javier Mazarío, gerentes da instalação principal de Microscopia e Análise de Imagem do Hospital Nacional de Paraplégicos, na Espanha.
*O sistema scanR não é vendido no Japão.
Conteúdo relacionado
Segmentação de instâncias de células e núcleos simplificada usando Aprendizado Profundo
Webinar: Triagem de alto conteúdo: análise personalizada simplificada

