A divisão celular é um processo essencial a todos os seres vivos. Antes de cada divisão, as informações genéticas da célula, que são armazenadas no DNA, precisam ser copiadas com precisão. Esse processo de cópia é conhecido como replicação do DNA e sua regulação correta desempenha um papel vital na saúde e na sobrevivência de um indivíduo. Saber como as células regulam a replicação do DNA para obter maior precisão na duplicação do genoma é uma das questões fundamentais na pesquisa biomédica. Os erros gerados durante a replicação do DNA podem levar à instabilidade do genoma associada a doenças humanas graves, como o câncer. De fato, pesquisas anteriores estimaram que até dois terços de todos os cânceres são causados pelo acúmulo de erros durante a replicação do DNA.
Conheça uma pioneira no campo da replicação do DNA
Como as células replicam seu DNA é também o foco da pesquisa de Hana Polasek-Sedlackova, PhD. Hana começou a trabalhar cedo no laboratório com pesquisa de DNA, em um estágio de pesquisa na Universidade de Masaryk, durante o segundo grau. Desde então, ela se tornou uma grande promessa na área. Hana venceu o renomado prêmio Undergraduate Award in Life Sciences, também chamado de "Prêmio Nobel Júnior" com sua tese de graduação. Depois da graduação, ela recebeu uma bolsa da Novo Nordisk Foundation para participar do programa de doutorado em biociências de Copenhague. Com essa bolsa, Hana trabalhou no laboratório do Professor Jiri Lukas no Novo Nordisk Foundation Center for Protein Research, Universidade de Copenhague, como aluna de doutorado, onde continuou seu treinamento de pós-doutorado. Seu trabalho de pesquisa foi publicado nas revistas científicas mais importantes, como a Science e a Nature. Recentemente, ela formou seu próprio grupo independente de pesquisa no Instituto de Biofísica (IBP) da Academia de Ciências Tcheca em Brno, República Tcheca.
Uma das suas contribuições à ciência foi resolver o paradoxo MCM, que está em debate desde os anos 1990 entre os cientistas da área. Ele descreve os resultados aparentemente contraditórios entre a microscopia e outros métodos científicos usados para elucidar os eventos durante a replicação. Essa contradição foi agora resolvida com a ajuda da nossa estação de escaneamento de alto conteúdo scanR, da qual Hana é uma grande usuária. Encontramos Hana durante uma reunião dos usuários do scanR no Instituto de Genética Molecular em Praga e tivemos a oportunidade de perguntar sobre sua experiência com o sistema e o impacto dele na sua pesquisa.
Nossa entrevista com Hana Polasek-Sedlackova
Evident: ao iniciar seu grupo de pesquisa no IBP, a estação de escaneamento de alto conteúdo scanR foi o primeiro microscópio que você comprou para o grupo. O que levou você a escolher esse sistema?
Hana Polasek-Sedlackova: durante minha experiência profissional, tive a oportunidade de trabalhar com vários microscópios. Com base nas minhas observações, percebi rapidamente que há três parâmetros importantes que deveriam ser considerados ao escolher um microscópio. Esses parâmetros são o hardware, o software e o atendimento ao cliente. Depois de considerar bastante esses critérios, cheguei à conclusão de que o scanR é o melhor sistema de escaneamento de alto conteúdo.
Primeiro, o scanR tem um sistema de hardware simples e adaptável, que pode ser ajustado aos requisitos específicos dos experimentos do usuário. Ele oferece recursos de formação de imagem que abrangem campo vasto, superresolução, técnicas de microscopia de fluorescência e a capacidade de formação de imagem de célula viva, e muito mais.
Segundo, o scanR tem uma plataforma de aquisição e análise fácil de usar e intuitiva, que permite que os usuários obtenham dados de alta qualidade e quantidade em pouco tempo. A análise multiparâmetro em tempo real dos dados de formação de imagem adquiridos possibilita a inspeção de dados imediata e maior otimização dos parâmetros de aquisição, incluindo uma função útil de repetição de escaneamento. (A função de repetição de escaneamento faz um escaneamento rápido da amostra em menor resolução para identificar objetos de interesse, que podem ser escaneados novamente em maior resolução.)
Terceiro, a Evident oferece um atendimento ao cliente excepcional. Os especialistas em aplicação para manutenção de microscópios e suporte à análise são profissionais excepcionais, sempre preparados para ajudar a atender às necessidades, aos desejos e aos problemas dos usuários. E foi graças ao Czech Science Foundation e aos fundos internos do nosso instituto que pudemos comprar e trazer a tecnologia do scanR ao meu novo instituto.
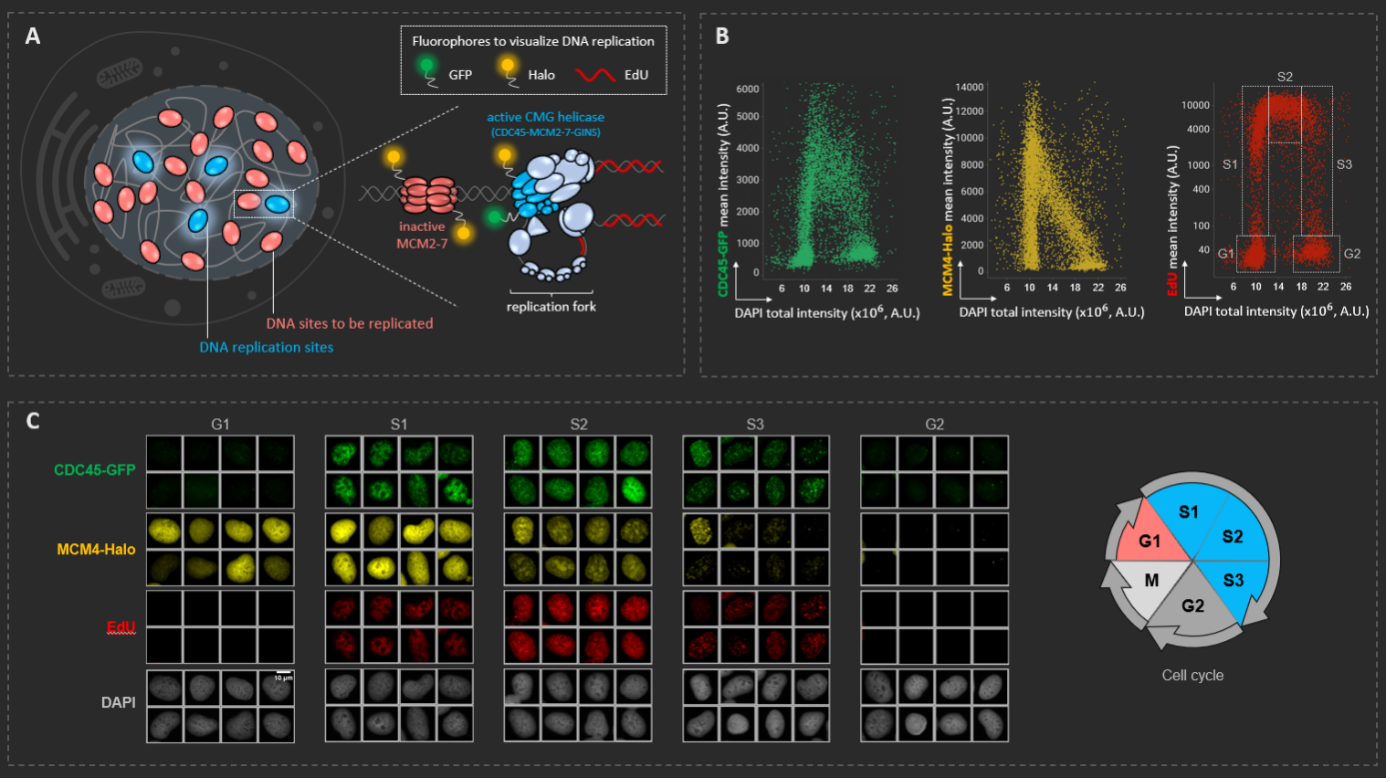
Medição da dinâmica de replicação do DNA pelo sistema scanR. (A) Representação esquemática de uma célula humana com zoom do garfo de replicação único, uma unidade básica da replicação do DNA. Sondas fluorescentes específicas para estudar a ligação do MCM no DNA (halo), a montagem da helicase CMG ativa (GFP) e a síntese de DNA (EdU). (B) A análise multiparâmetro de eventos importantes da replicação de DNA nas células U2OS do osteosarcoma humano feita pelo scanR. (C) Galerias de imagem que ilustram a dinâmica das proteínas de replicação do DNA durante o ciclo celular.
Evident: como o sistema scanR fez sua pesquisa progredir? Você pode dar um exemplo em que o sistema scanR ajudou a revelar novos insights?
Hana: o sistema scanR nos ajudou a fazer descobertas fundamentais no campo de replicação do DNA. Na minha pesquisa, ele teve um papel importante para resolver o paradoxo MCM: um quebra-cabeças duradouro relacionado ao comportamento do complexo de proteína MCM2-7. O complexo MCM forma o núcleo da helicase replicativa, que é responsável por desenrolar dupla hélice de DNA para garantir uma duplicação precisa. Por anos, os pesquisadores ficaram perplexos por dois fatos paradoxais amplamente relatados: (1) Por que as células contêm um grande excesso de MCMs inativos se apenas 5–10% são usados como helicases replicativas ativas? (2) Por que MCMs de eucariotas nunca foram vistos nos locais de replicação dentro de uma célula usando abordagens de microscopia?
Recentemente, revisitamos esse impasse conceitual persistente e esclarecemos o paradoxo MCM. Usamos a edição de genoma CRISPR-Cas9 e a formação de imagem de alto conteúdo do scanR para elucidar dificuldades antigas na formação de imagem de garfos de replicação de DNA em células vidas e revelar o novo caminho de homeostase das origens de replicação do DNA necessário para proteger a integridade do genoma.1-2
Descobrimos que o excesso de MCMs inativos age como locais de pausa naturais da replicação (NRPSs), ajustando a velocidade do garfo de replicação para minimizar os erros durante a replicação do DNA. Com esse recurso, o NRPSs representa outra camada de controle de velocidade do garfo de replicação explorada por células cancerígenas, o que os torna um novo alvo atraente para terapia anticâncer. No momento, minha equipe de pesquisa está explorando os caminhos regulatórios mais apicais das origens de replicação do DNA e sua contribuição aos estados patofisiológicos do desenvolvimento do tecido humano.

A descoberta de variantes de proteínas dos complexos do MCM usando o scanR permite compreender o paradoxo MCM. (A) Um protocolo chamado HaloTag para distinguir pools de proteínas de MCM. A análise de alto conteúdo baseada no scanR revelou dinâmicas diferentes dos complexos de proteína MCM parentais e nascentes durante o programa de replicação do DNA. (B) Um modelo que descreve a função distinta de MCMs parentais e nascentes durante a replicação do DNA. Enquanto os MCMs parentais são convertidos principalmente em helicases CMG ativas, os MCMs nascentes permanecem em grande medida inativos, mas funcionam como replissomas naturais, que pausam locais importantes para definição do movimento correto do garfo de replicação.
Evident: você usa o sistema scanR para quais aplicações e que tipo de amostras são usadas para formar imagens?
Hana: em nossa pesquisa, usamos vários modelos de cultura de tecidos, e a maioria deles é derivada de amostras humanas. Essas amostrar incluem linhas de células cancerígenas de vários tecidos, como ossos, peito, ovário, cérvix ou rim, assim como suas contrapartes não cancerígenas, células-tronco e fibroblastos. Ocasionalmente, também empregamos amostras de animais nos nossos estudos. Usamos essas amostras combinadas com várias abordagens de biologia celular, bioquímica e genética para identificar novas proteínas ou caminhos regulatórios inteiros, que desempenham um papel importante na duplicação correta do genoma. Além disso, investigamos como essas novas proteínas ou caminhos são regulados em células cancerígenas e se isso oferece uma vantagem a elas no desenvolvimento do tumor. Se descobrirmos que sim, poderemos usar nosso conhecimento para desenvolver novas abordagens na terapia anticâncer.
Nosso pipeline experimental comum para identificar novos fatores de proteína na replicação do DNA envolve o perfil de uma grande população de células únicas, e o sistema scanR tem sido de imensa ajuda nesse sentido. Por exemplo, se identificarmos uma nova proteína candidata, podemos primeiro fazer sua downregulation em células, usando vários métodos. Em seguida, usando formação de imagem de alto conteúdo, monitoraremos a precisão da replicação do DNA, etapa a etapa. A vantagem de usar células em nossos experimentos é que podemos observar o processo de duplicação do genoma em seu ambiente natural, mas, sua complexidade também pode levar a desafios na interpretação de dados.
Por esse motivo, ao estudar a replicação do DNA, precisamos examinar com cuidado como o esgotamento do nosso gene de interesse afeta outros processos celulares, como o ciclo celular, a resposta a danos no DNA, a transcrição, a manutenção da cromatina, a manutenção de regiões específicas do cromossomo, estabilidade geral do genoma, etc. Tais perfis neutros e quantitativos de grandes população de células únicas obtidos em um curto período nos ajudam muito a interpretar nossos dados com rapidez e assim fazer nossa pesquisa avançar com mais eficiência.
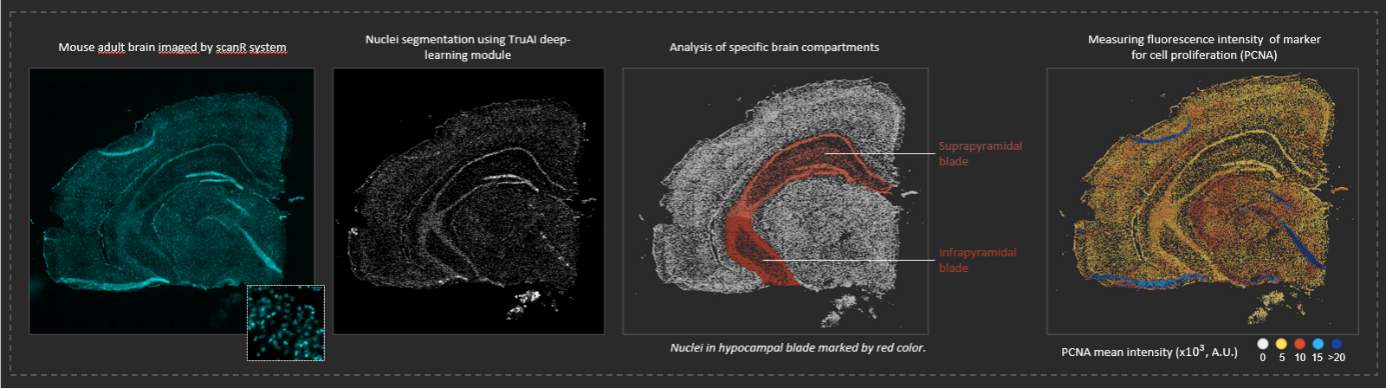
Análise de imagem de células individuais no cérebro de camundongo adulto usando o módulo de aprendizado profundo TruAI. A amostra de cérebro foi preparada pelo Dr. Jana Krejci (Departamento de Epigenética de Biologia Celular do Instituto de Biofísica).
Costumava passar horas ao microscópio capturando manualmente imagens e depois ainda mais tempo no computador para fazer as análises. Eu levava meses para concluir esses experimentos. Agora, com o scanR, tudo pode ser feito em apenas algumas horas.
Evident: como essas aplicações aproveitam os recursos de análise de alto conteúdo e a abordagem de citometria baseada em imagem do sistema?
Hana: no campo da biologia celular, acredito que a combinação de informações quantitativas imparciais com imagens é uma forma confiável e precisa de representar processos celulares vitais. O sistema scanR oferece uma solução elegante e inteligente para esse fim. Como já mencionei, o sistema scanR oferece aquisição e análise de amostras totalmente automatizadas, que nos permite obter informações imparciais e quantitativas sobre os processos celulares. O mais importante é que, com apenas um clique, é possível inspecionar as imagens em que se baseiam os dados.
Além disso, é possível criar galerias de imagem imparciais que ilustram a dinâmica das proteínas de interesse ao longo da vida da célula. Graças a essa união exclusiva entre imagem e dados, é possível ajustar ainda mais a análise dos processos celulares a compartimentos específicos, como o núcleo ou citoplasma, além de compartimentos subnucleares, como foco de reparos de DNA, etc. Essa complementação visual dos dados quantitativos ajuda a resolver problemas biológicos com mais eficácia.
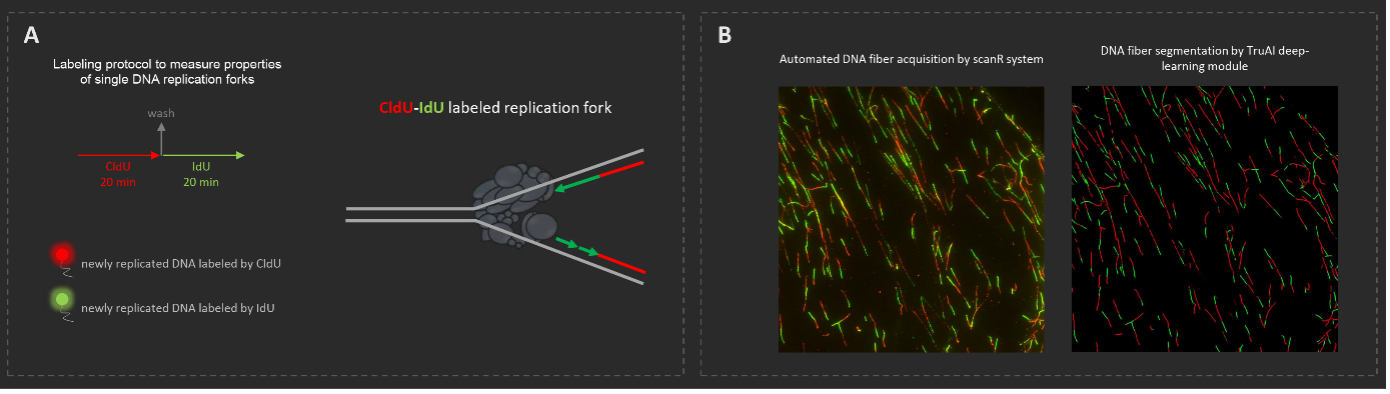
Exemplo de trabalho em andamento na aquisição e análise automatizadas de fibras de DNA com a combinação dos sistemas scanR e cellSens™. (A) Um protocolo de rotulação de CldU-IdU para visualizar garfos de replicação únicos. (B) Exemplos de aquisição s segmentação automatizadas de fibras de DNA pelo sistema scanR e software cellSens™.
Evident: o sistema mudou sua abordagem à microscopia como ferramenta na biologia celular e biologia molecular? Você usa os recursos de aprendizado profundo TruAI™ do sistema?
Hana: durante meu doutorado no laboratório do professor Jiri Lukas, conheci a formação de imagem de alto conteúdo e o sistema scanR. Essa tecnologia revolucionária mudou completamente meu ponto de vista sobre a biologia celular e a pesquisa em geral. Fiquei especialmente impressionada pela capacidade de oferecer uma análise imparcial e totalmente automatizada dos processos celulares. Dada a crise atual de reprodutibilidade na pesquisa científica, acredito que os pesquisadores devam buscar ferramentas que permitam obter e analisar dados de maneira totalmente automatizada, que oferece uma representação imparcial do processo celular.
Na era atual da inteligência artificial (IA), a análise de imagem avançou além dos limites anteriores. O sistema scanR oferece agora o módulo de aprendizado profundo TruAI, que expandiu o escopo da minha pesquisa. Antes, meu foco principal era analisar células únicas em grandes populações de linhagens de células humanas de cultura. No entanto, com o módulo de IA, podemos, por exemplo, conduzir análises de imagem extremamente precisas de células individuais em seções de órgão.
Outro exemplo de como podemos desafiar os limites dos nossos recursos de análise de imagem é com a combinação dos sistemas scanR e cellSens™. Isso nos permite realizar aquisição e análise totalmente automatizadas de moléculas únicas, como garfos de replicação de DNA preparados pela técnica de fibra de DNA. Esses são alguns exemplos de novas aplicações em que estamos trabalhando no meu laboratório, e esperamos que outros pesquisadores aproveitem essas ferramentas em breve para fazer grandes descobertas no campo da biologia celular.
Evident: do que vocês mais gostam no sistema scanR? Como ele é recebido pelos pesquisadores associados?
Hana: gosto de muitos recursos do sistema scanR, mas se precisasse destacar apenas um, seria a conexão única entre aquisição e análise automatizadas, que nos permite realizar análises multiparâmetros de imagens adquiridas em tempo real. Fiquei impressionada com esse recurso quando era aluna de doutorado, e todo novo membro da nossa pequena comunidade de usuários do scanR no nosso instituto também se impressiona.
Um dos meus colegas resumiu bem o assunto: “costumava gastar horas ao microscópio capturando manualmente imagens, e ainda mais tempo no computador fazendo as análises. Eu levava meses para concluir esses experimentos. Agora, com o scanR, tudo pode ser feito em apenas algumas horas.”
Para mais informações sobre o sistema scanR
Queremos estender nossos sinceros agradecimentos à Dra. Hana Polasek-Sedlackova por contar sua experiência com a estação de escaneamento de alto conteúdo scanR. Para mais informações sobre o sistema scanR e nossas outras soluções de pesquisa de ciências da vida, entre em contato diretamente com a Evident ou nosso representante local de vendas.
Foto da Dra. Hana Polasek-Sedlackova por Jana Mensatorova.
Referências
- Polasek-Sedlackova H, Miller TCR, Krejci J, Rask MB, Lukas J. Solving the MCM paradox by visualizing the scaffold of CMG helicase at active replisomes. Nat Commun. 14 de outubro de 2022;13(1):6090. doi: 10.1038/s41467-022-33887-5. PMID: 36241664; PMCID: PMC9568601. https://www.nature.com/articles/s41467-022-33887-5
- Sedlackova, H., Rask, M.-B., Gupta, R., Choudhary, C., Somyajit, K., Lukas, J. (2020, October 21). Equilibrium between nascent and parental MCM proteins protects replicating genomes. Nature News. https://www.nature.com/articles/s41586-020-2842-3
Conteúdo relacionado
Segmentação de instâncias de células e núcleos simplificada usando Aprendizado Profundo
Localização de proteína da levedura classificada usando a tecnologia de aprendizado profundo TruAI™

