Previsões de fenótipos de núcleos multiclasse para testes de medicamentos usando deep learning
Introdução
A quantificação do movimento dinâmico das proteínas, tais como os receptores nucleares em resposta aos medicamentos, requer métodos de segmentação nuclear robustos usando corantes e colorações. No entanto, esse processo possui limitações significativas: os procedimentos de coloração de células vivas são demorados, caros e podem levar à fototoxicidade, ao fotobranqueamento e à desativação de compostos fotossensíveis (por ex., R18811).
Para superar esses desafios, desenvolvemos uma rede neural (neural network, NN) usando o software de formação de imagem cellSens™ com a tecnologia de deep learning TruAI™ para identificar núcleos de células sem usar marcações fluorescentes. Depois, treinamos a NN para distinguir entre os diferentes fenótipos celulares em resposta aos medicamentos. Aqui, aplicamos o processo de trabalho com NN para prever a dinâmica intracelular do receptor de andrógenos (androgen receptor, AR) em células vivas de câncer de próstata tratadas com medicamentos que têm o AR como alvo.
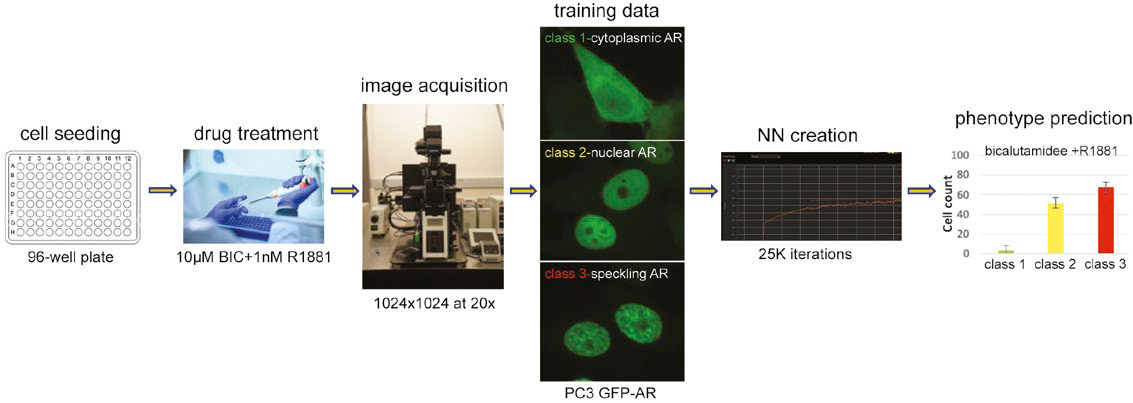
Figura 1: Fluxo de trabalho para prever os fenótipos de núcleos multiclasse usando uma NN treinada.
Após a ativação, o AR citoplasmático se transloca para o núcleo, onde se localiza em locais transcricionalmente ativos, que podem ser visualizados como um padrão de speckle distinto usando a microscopia confocal. Para prever a dinâmica do AR, primeiramente geramos uma NN capaz de detectar núcleos de células sem usar colorações nucleares. Em seguida, classificamos as células com base na translocação nuclear de GFP-AR e no estado de transativação (speckle), ambas leituras de ensaio essenciais para testar compostos quanto à sua capacidade de ter o AR como alvo. Por fim, demonstramos que nosso fluxo de trabalho com NN prevê esses fenótipos de AR críticos com sucesso sem o uso de coloração nuclear.
Vantagens
- Identificação de fenótipos de núcleos sem o uso de marcações fluorescentes
- Economia de tempo e dinheiro gastos com colorações nucleares (coloração e formação de imagem)
- Redução da fototoxicidade e do fotobranqueamento
- Preserva a atividade dos compostos fotossensíveis
Métodos
Geramos um conjunto de dados de treinamento composto dos três principais fenótipos de AR comumente observados em resposta aos medicamentos. Usamos metade dessas imagens para gerar a NN e a outra metade para avaliar seu desempenho. Por fim, aplicamos nossa NN multiclasse a um conjunto de dados independente sem uma coloração nuclear.
Preparação dos dados de treinamento a partir de três fenótipos de AR
Como primeira etapa da construção de uma NN, geramos um conjunto de dados de treinamento composto dos três principais fenótipos de AR:
- AR citoplasmático inativo (controle sem tratamento [no treatment control, NTC])
- AR nuclear ligado, mas inativo (10 μM de bicalutamida)
- Speckle de AR nuclear ativo (1 nM de R1881)
Para gerar os dados ground truth, tingimos os núcleos usando SiR-DNA. Adquirimos imagens com uma taxa de pixels de 1024 x 1024 com uma objetiva de 20X UPLSAPO usando o escâner galvanômetro do microscópio confocal a laser Olympus FV3000. Aqui, dividimos o conjunto de dados completo (60 imagens) em duas partes iguais para o conjunto de treinamento e para o conjunto de validação (30 imagens cada). Para treinar a NN, usamos 10 imagens de GFP-AR projetadas com a intensidade máxima de cada um dos três fenótipos de AR (50% dos dados, 30 imagens no total) com uma coloração nuclear.
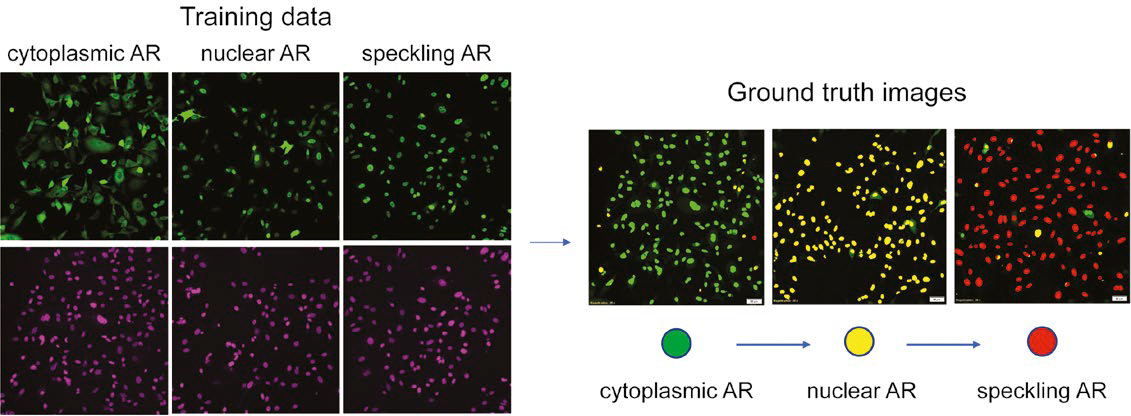
Figura 2: Preparação dos dados de treinamento de fenótipos de AR multiclasse com base na coloração de núcleos. (À esquerda) verde: AR-GFP, violeta: SiR-DNA. (À direita) avaliação dos conjuntos de dados ground truth.
Otimização do ground truth
A Figura 2 demonstra os dados ground truth avaliados pela NN, com o AR citoplasmático em verde, o AR nuclear em amarelo e o AR com speckle em vermelho. Usando esse mapa de segmentação codificado por cores, ajustamos o ground truth corrigindo manualmente os erros na detecção e segmentação nuclear e reclassificando os núcleos que foram categorizados incorretamente.
Criação da rede neural usando a tecnologia de deep learning TruAI™
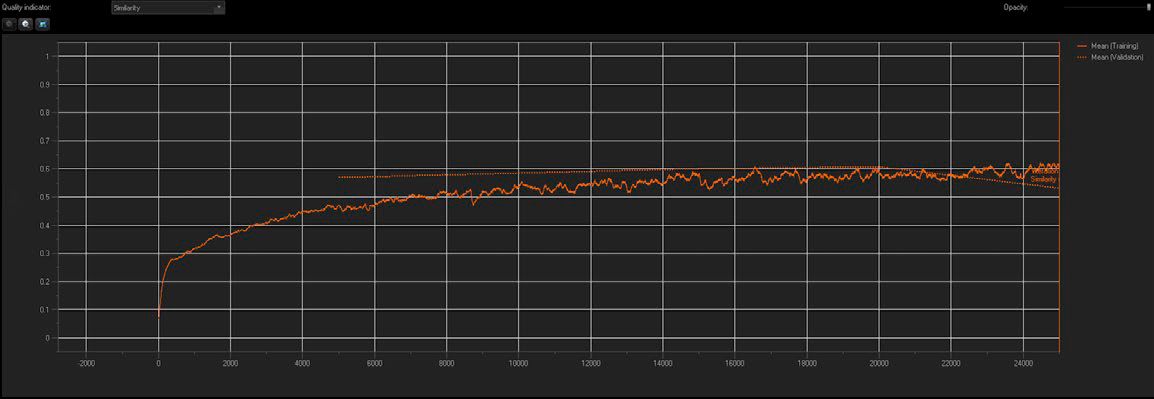
Figura 3: O treinamento da NN relata o índice de similaridade e os valores médios de validação em tempo real.
Usamos a tecnologia TruAI para criar uma NN padrão com configurações multiclasse, incorporando 10 imagens de treinamento de cada fenótipo de AR. A qualidade e a precisão da NN foram relatadas em tempo real. Conforme mostrado na Figura 3, o índice de similaridade aumentou a cada iteração sucessiva durante o processo de treinamento até atingir um platô, indicando uma saturação do treinamento (seta para baixo). Foram criados pontos de verificação a cada 5.000 iterações (20% de progresso), e o ponto de verificação ideal foi salvo como uma NN treinada junto com seu valor de índice de similaridade.
Resultados
Validação de uma rede neural multiclasse em dados cegos
Validamos nosso modelo de NN multiclasse usando 50% das imagens que não foram usadas para o treinamento do ground truth ou da NN (dados cegos). Em seguida, usamos esses resultados de validação para ajustar os parâmetros do modelo aplicando um filtro de área (mín. 49,83 μm – máx. 555,44 μm) para eliminar os restos celulares e as células segmentadas incorretamente. O modelo de NN treinada previu os fenótipos de AR multiclasse independentemente, sem nenhuma coloração nuclear. A Figura 4 mostra a distribuição de classes de fenótipos usando os dados de 10 imagens.
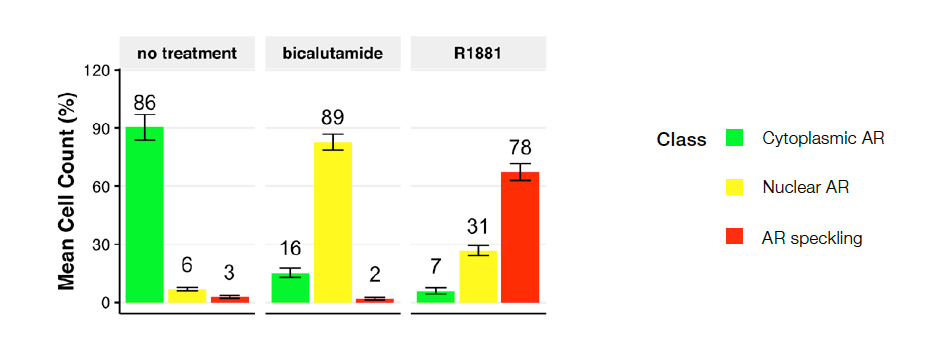
Figura 4: Distribuição da classe celular para as seguintes condições: controle sem tratamento (à esquerda), bicalutamida (centro) ou R1881 (à direita). O gráfico apresenta a porcentagem de células categorizadas como classe 1 (AR citoplasmático), classe 2 (AR nuclear) e classe 3 (AR com speckle). O controle sem tratamento resultou principalmente em células de classe 1. A bicalutamida (antagonista do AR) induziu uma mudança em direção à classe 2, e o R1881 (agonista do AR) levou à classe 3, o AR ativo localizado em locais transcricionalmente ativos.
Previsão dos fenótipos de AR multiclasse em conjuntos de dados independentes
Depois, para avaliar o modelo de NN multiclasse em imagens independentes, preparamos um novo conjunto de dados usando dois antiandrogênicos clínicos: bicalutamida e enzalutamida (Figura 5).
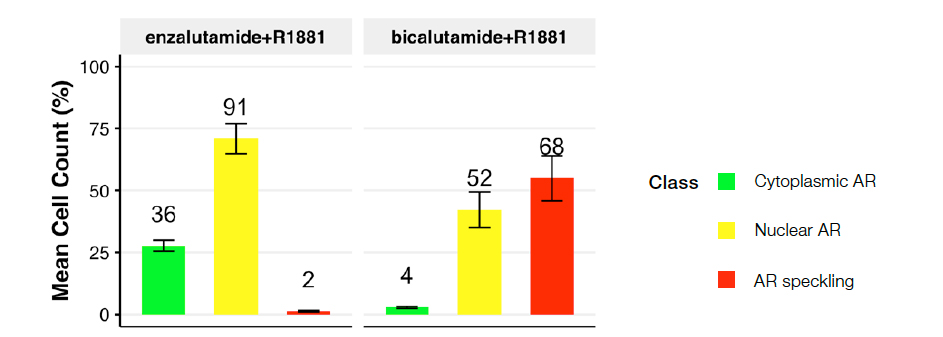
Figura 5: Distribuição da classe celular para as seguintes condições: R1881 mais enzalutamida (à esquerda) ou bicalutamida (à direita). O gráfico retrata a distribuição prevista da classe de núcleos em todas as condições de tratamento.
O tratamento com o antiandrógeno de segunda geração, a enzalutamida, levou a uma população de AR citoplasmático proeminente (classe 2) com pouco ou nenhum AR com speckle (classe 3), sugerindo um bloqueio eficaz da ativação do AR induzido por ligante. Em contraste, o antiandrógeno mais antigo e menos eficaz, a bicalutamida, levou a uma mudança considerável em direção ao AR ativo (classe 3), sugerindo sua incapacidade de bloquear de maneira eficaz a ativação do AR induzida por 1 nM de R1881. Esses dados confirmam os estudos anteriores2.
Conclusão
Usando o microscópio confocal Olympus FV3000 e o software cellSens™ com o módulo de deep learning TruAI™, geramos uma NN para detectar e segmentar núcleos não tingidos e subclassificar as células com base nas alterações fenotípicas do AR em resposta aos medicamentos. Essa abordagem tem o potencial de aumentar a eficácia dos testes de medicamentos ao diminuir o tempo (configuração experimental) e os custos dos testes.
Além disso, as NNs podem melhorar a qualidade dos dados ao reduzir os artefatos, tais como a fototoxicidade, o fotobranqueamento e a inativação de compostos fotossensíveis, e detectar alterações sutis que podem passar despercebidas pelo pesquisador e/ou pipelines de análise de imagem tradicionais. Essa abordagem com deep learning pode ser aplicada de forma mais geral para subclassificar populações de células com base em parâmetros nucleares ou baseados em células, que vão desde a localização de proteínas às mudanças morfométricas.
Referências
- Risek B, Bilski P, Rice AB, Schrader WT. Androgen receptor-mediated apoptosis is regulated by photoactivatable androgen receptor ligands. Mol Endocrinol. 2008 Sep; 22(9):2099-115. doi: 10.1210/me.2007-0426. Epub: 18 de junho de 2008. PMID: 18562628; PMCID: PMC2631375.
- Sugawara T, Lejeune P, K hr S, Neuhaus R, Faus H, Gelato KA, Busemann M, Cleve A, Lücking U, von Nussbaum F, Brands M, Mumberg D, Jung K, Stephan C, Haendler B. BAY 1024767 blocks androgen receptor mutants found in castration-resistant prostate cancer patients. Oncotarget. 2 de fevereiro de 2016; 7(5):6015-28. doi: 10.18632/oncotarget.6864. PMID: 26760770; PMCID: PMC4868737.
Autores
- Harish Sura, Katherin Patsch, e Seungil Kim do Lawrence J. Ellison Institute for Transformative Medicine, University of Southern California, Los Angeles, CA
- Shohei Imamura, Gerente estratégico de projetos, Olympus Corporation of the Americas
Produtos usados nesta aplicação
foi adicionado com sucesso aos seus favoritos
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
Not Available in Your Country
Sorry, this page is not
available in your country.