라이브 셀 이미징을 위한 초고해상도
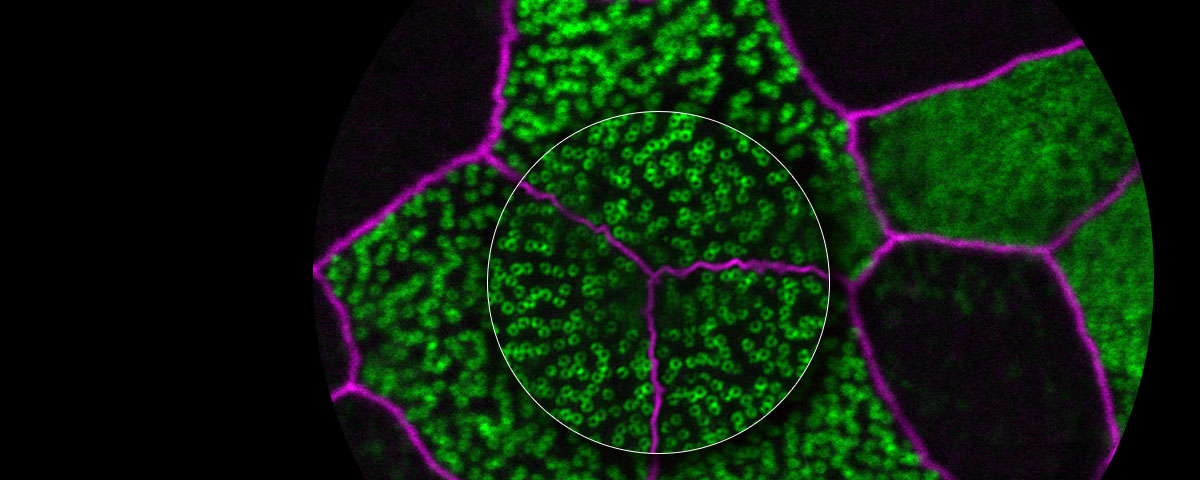
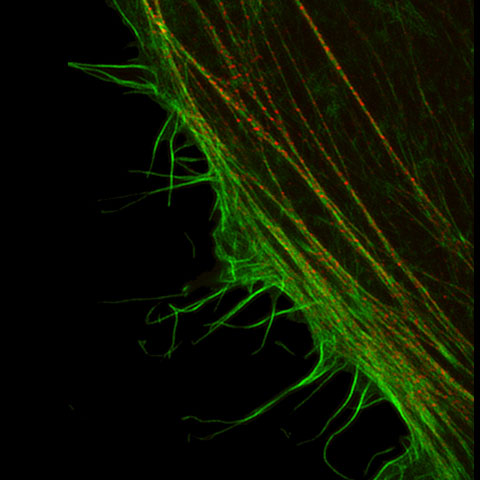
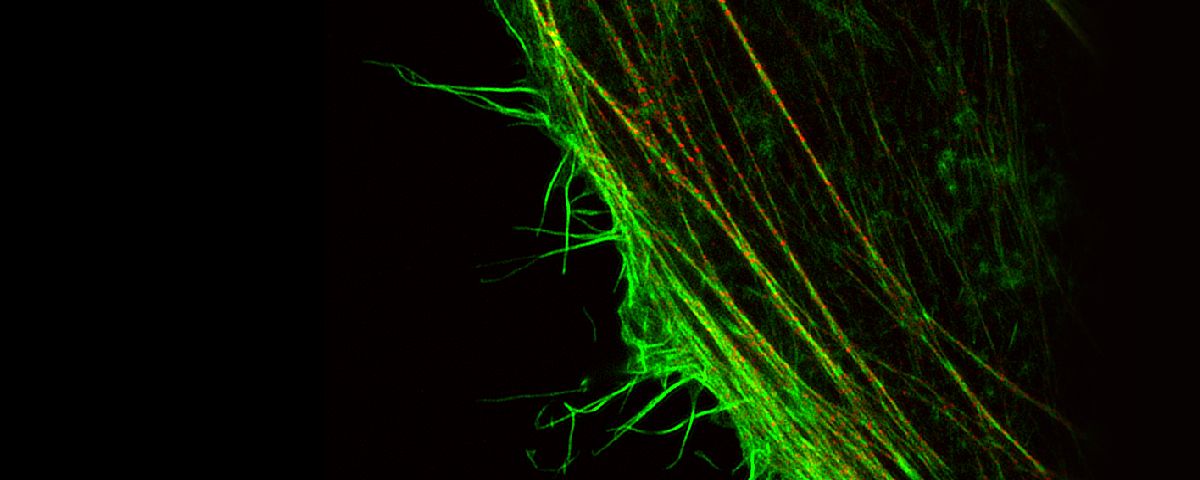
형광 현미경은 형광 프로브를 사용하여 체내 특정 단백질을 식별할 수 있습니다. 형광 현미경들의 대부분의 해상도는 회절 한계에 의해 약 200nm으로 제한되어 미세 구조를 관찰하는 것이 불가능합니다. 하지만 Olympus 초고해상도 기술을 이용하면 수평 방향으로 120nm에 이르는 해상도로 선명한 이미지를 획득할 수 있습니다.
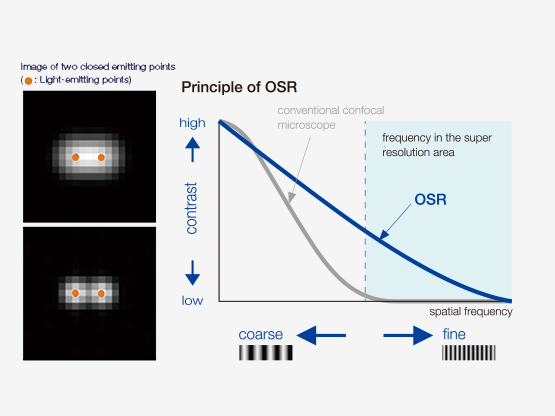
OSR은 어떻게 작동하나요?
| 향상된 신호 검출 능력, 특별한 하드웨어 구성, 최적화된 컨포칼 개구 직경 및 첨단 신호 처리 기술이 결합되어 초고해상도 이미지를 생성합니다. OSR(Olympus Super Resolution) 기술은 120nm에 이르는 공간(XY) 해상도를 구현합니다. 참고문헌: |
OSR(Olympus Super Resolution)
IXplore SpinSR10
FLUOVIEW FV3000에 FV-OSR 사용
|
훨씬 더 많이 보기
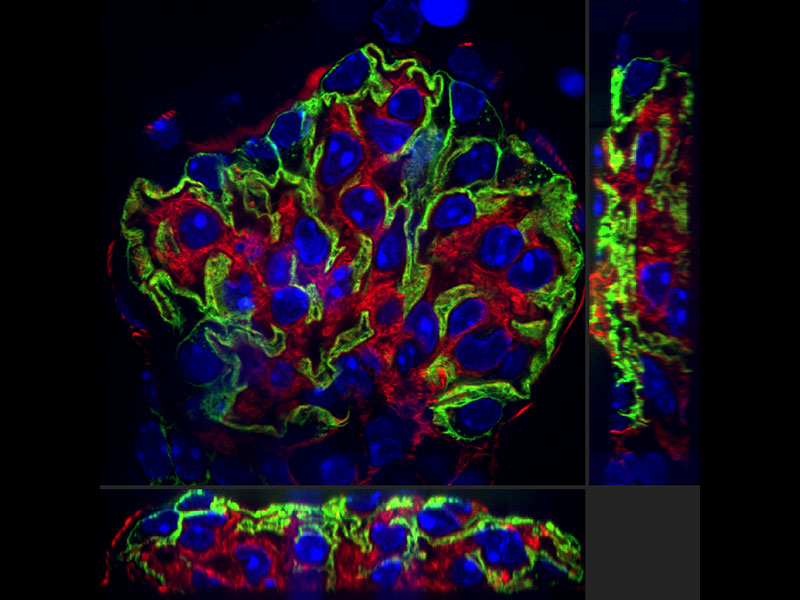
당사의 디콘볼루션 알고리즘을 통해 초고해상도 이미지가 더 맑고 더 선명하게 됩니다. 3D 제약 반복 디콘볼루션은 더 선명한 3D 이미지를 위해 Z 축의 블러링을 제거합니다.
|
추천의 글
Yasushi Okada, M.D. 겸 Ph.D., Riken 정량생물학센터 | 대부분의 세포내 세포소기관 및 초거대분자복합체는 약 100nm 크기 범위이며 일반 광학 현미경으로는 이러한 복합체의 구조를 관찰할 수 없습니다. 오늘날 여러 초고해상도 현미경 사용 방법이 개발되어 있지만, 이러한 방법에 전용 염료, 전용 관찰 조건 또는 특수 광학 시스템이 요구되기 때문에 생물학 분야에서는 이러한 방법을 쉽게 시도할 수 없습니다. 이 Olympus 초고해상도 현미경은 회전 컨포칼 현미경을 쉽게 업그레이드할 수 있습니다. 또한, 실리콘 이멀젼 대물 렌즈와 결합하여 구면 수차를 줄이고 세포의 심층면에서 초고해상도 라이브 이미징을 획득할 수 있으므로 이 기술이 생물학 분야의 대부분의 응용에 사용될 수 있는 도구가 될 것으로 예상됩니다. |
Sachiko Tsukita, 오사카 대학 프런티어 생명 과학 대학원 및 의과 대학원 | 우리는 다중 솜털이 있는 세포의 섬모를 생성하는 기저 소체의 조정된 정렬 및 방향의 배후에 있는 메커니즘을 연구하고 있습니다. 규칙적인 기저 소체 패턴은 섬모파에 대단히 중요합니다. 기관의 1차 배양 시스템의 다중 솜털이 있는 세포의 분화 중에 GFP 센트린 표지 기저 소체를 명확히 관찰하려면 X, Y 및 Z 축에 각기 200nm 이하의 해상도가 필요합니다. SpinSR10 현미경을 포함한 라이브 셀 이미징 시스템을 사용하여 기저 소체의 움직임의 분석이 가능하게 되었습니다. 이러한 방법으로 우리는 기저 소체가 기관의 다중 솜털이 있는 세포에 정단 세포 골격의 자기 조직화에 따라 정렬되는 새로운 메커니즘을 발견하였습니다. |
Yuji Ikegaya, Ph.D., 도쿄 대학교 약학 대학원 화학 약리학 연구소 | SpinSR10 현미경에서 초고해상도 형광을 처음 보았을 때 그 아름다움에 놀랐습니다. 나는 뇌의 미세구조의 가소성, 특히 뒤쪽 신경 접합부(척추), 신경 섬유 및 신경교 세포 과정을 연구하고 있습니다. SpinSR10 현미경을 사용하여 획득된 이미지를 통해 마침내 “볼” 수 있다는 것을 깨달았습니다. 전자 현미경 외에 미세구조를 상세히 획득하는 방법을 이용하여 획득된 데이터의 신뢰도가 향상되었으며 간소화된 데이터 획득 절차로 인해 실험의 효율이 크게 증가되었습니다. 또한, 같은 조직에서 더 긴 타임랩스 이미징 실현을 통해 새 연구 방법이 수립되었습니다. |
참고문헌
S. Hayashi and Y. Okada, “Ultrafast superresolution fluorescence imaging with spinning disk confocal microscope optics,” Mol. Biol. Cell 26(9), 1743–1751 (2015).
S. Hayashi, “Resolution doubling using confocal microscopy via analogy with structured illumination microscopy,” Jpn. J. Appl. Phys. 55(8), 082501 (2016).
A. Nagasawa-Masuda and K. Terai, “Yap/Taz transcriptional activity is essential for vascular regression via Ctgf expression and actin polymerization,” PLoS ONE 12(4), e0174633 (2017).
H. Nakajima, et al., “Flow-Dependent Endothelial YAP Regulation Contributes to Vessel Maintenance,” Dev. Cell 40(6), 523-536.e6 (2017).
K. Tateishi, et al., “Three-dimensional Organization of Layered Apical Cytoskeletal Networks Associated with Mouse Airway Tissue Development,” Sci. Rep. 7, 43783 (2017).
E. Herawati, et al., “Multiciliated cell basal bodies align in stereotypical patterns coordinated by the apical cytoskeleton,” J. Cell Biol. 214(5) 571-586 (2016).
M.-T. Ke, et al., “Super-Resolution Mapping of Neuronal Circuitry With an Index-Optimized Clearing Agent,” Cell Rep. 14(11) 2718–2732 (2016).
Sorry, this page is not
available in your country.
Sorry, this page is not
available in your country.
.jpg?rev=2087)