3D 살아있는/죽은 세포 분석에 대한 분석 워크플로
요약
NoviSight 3D 세포 분석 소프트웨어는 부피, 구형도 및 세포 번호 같은 매개변수를 측정하여 스페로이드 및 기타 3D 객체에 대한 통계 데이터를 제공합니다. 이 애플리케이션 노트에서는 살아있는/죽은 스페로이드 분석에 대한 NoviSight 3D 세포 분석 워크플로를 소개합니다.
서론
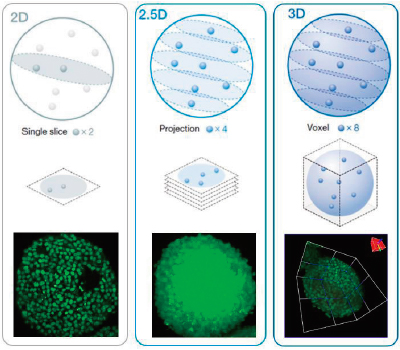
스페로이드 또는 오르가노이드를 사용하는 3D 대용량 분석(HCA)이 어려울 수 있습니다. 이상적으로 말하면, 3D 샘플을 입체적으로 분석하여 형태 및 내부 상태에 대한 정보를 얻을 수 있습니다. NoviSight 소프트웨어의 정확한 3D 분석은 순차 Z 스택 형광 이미지를 기반으로 부피, 세포 수 및 3D 샘플의 세포 상태를 측정하여 이 과정을 단순화할 수 있습니다. 반면에 기존 분석은 단일 이미지 슬라이스(그림 1, 좌측, 2D 분석)를 사용하거나 투사 이미지(그림 1, 중앙, 2.5D 분석)를 사용합니다. 이 두 기법 모두 공간, 형태 및 부피 정보 같은 3D 샘플의 많은 데이터를 상실합니다(그림 1). 이 애플리케이션 노트에서는 살아있는/죽은 스페로이드 분석을 사용하여 NoviSight 소프트웨어로 3D 샘플을 정량적으로 분석하는 방법을 보여줍니다. |
그림 1(좌측에서 우측으로): 2D, 2.5D 및 3D 분석도. |
방법
샘플 제공
스페로이드를 형성하기 위해 8일 동안 PrimeSurface® 96U 플레이트(Sumitomo Bakelite)에 500 HT-29 셀/웰을 배양한 후 다음 날 NucView® 550(Biotium)을 사용하여 다양한 농도의 스타우로스포린(STS)을 적용하였습니다. NucView 550은 카스파제-3 형광 프로브이며, 죽은 세포핵들은 빨간색 형광으로 착색되었습니다. STS와 NucView 550을 사용하여 처리 후 HT-29 스페로이드를 4% 파라포름알데히드로 고정시키고 0.5% Triton X-100/PBS(-)로 투과시켰습니다. 스페로이드의 핵들은 4°C(39.2°F)로 하룻밤 동안 Hoechst 33342로 착색되었습니다. 착색 후 스페로이드를 SCALEVIEW-S4로 세정하였습니다.
이미징 및 분석
FV3000 컨포칼 현미경과 세미 아포크로맷 대물렌즈(LUCPLFLN20X)를 사용하여 샘플의 3D 이미지를 획득하였습니다. 아래에서 획득 조건을 확인할 수 있습니다. Z 스텝
크기는 2μm였습니다. 405nm 레이저를 Hoechst 33342(파란색 형광)에 사용하였고, 561nm 레이저를 NucView 550(마젠타색 형광)에 사용하였습니다. 그림 2는 현미경을 사용하여 획득된
비처리 및 STS 처리 스페로이드의 용적 이미지를 보여줍니다. 8일간 배양된 비처리 스페로이드의 코어는 저영양이나 저산소증으로 죽었을 것으로
예상됩니다. 전반적으로 STS가 HT-29 스페로이드의 세포 죽음을 유도하였습니다.
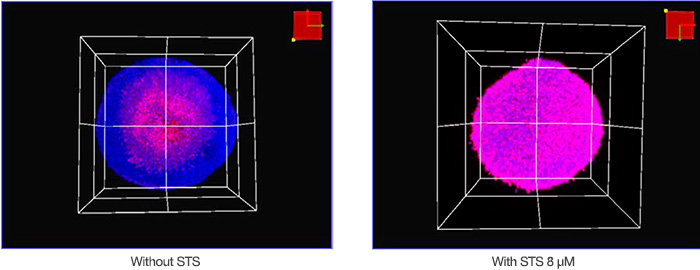
그림 2: STS 적용(우측) 및 미적용(좌측) HT-29 스페로이드의 용적 그림.
이점
- 3D 살아있는/죽은 생존율 테스트 수행이 용이함
- 직관적 사용자 인터페이스를 통해 Z 스택 이미지를 재현가능한 정량적 데이터로 변환 가능
3D 살아있는/죽은 분석 전략
NoviSight™ 소프트웨어를 사용하여 살아있는/죽은 스페로이드를 정량적으로 분석하는 데 표 1에 기술된 단계가 필요합니다. NoviSight 소프트웨어가 Z 평면
이미지의 핵들을 입체적으로 인식하도록 Z 섹션의 스텝 크기를 단일 핵을 검출하기 위한 최대 치수인 3μm으로 설정합니다.
| 단계 | 세부사항 |
| 샘플 제공 |
세포 배양 및 형광 염색
옵션: 고정 및 세정 세포 수를 분석하려면 핵을 착색해야 합니다. ↓ |
| 이미징 |
여러 Z 스택 이미지 획득
예: 컨포칼 현미경 검사, 다광자 현미경 검사 ↓ |
| 분석 |
1. 관심 있는 대상 인식
예: 핵, 부피, 세포기관 2. 분석 매개변수 설정 3. 게이팅별 그래프 분석 4. 통계 분석 |
표 1: 샘플 제공에서 분석에 이르는 3D 워크플로.
NoviSight 소프트웨어 분석 단계
주 객체 인식
살아있는/죽은 세포의 수로 살아있는/죽은 세포 분석을 평가하였습니다. NoviSight 소프트웨어는 핵 염색에서 세포 수를 검출할 수 있습니다. 세포 핵을 인식하기 위하여 다음과 같은 핵 검출을 위해 전문화된 NoviSight 모듈을 사용하였습니다. NuclearL. 해당 세포 크기, 배경 강도 및 Z/XY 비율(핵의 Z 축 신장)을 입력하면 NoviSight 소프트웨어가 3D 공간에서 스페로이드의 핵들을 자동으로 검출할 수 있습니다(그림 3A).
분석 매개변수 설정
Hoechst 33342로 착색된 모든 핵들의 경우 높은 NucView 550 신호 강도의 핵들은 죽은 세포였습니다. 결과적으로 NoviSight 소프트웨어는 인식된 핵 객체에서 CH2(NucView 550) 강도 레벨로 살아있는/죽은 세포를 분류할 수 있습니다(그림 3B). 즉 NoviSight 소프트웨어는 핵 인식 설정만으로 스페로이드의 살아있는/죽은 세포를 분류할 수 있습니다.
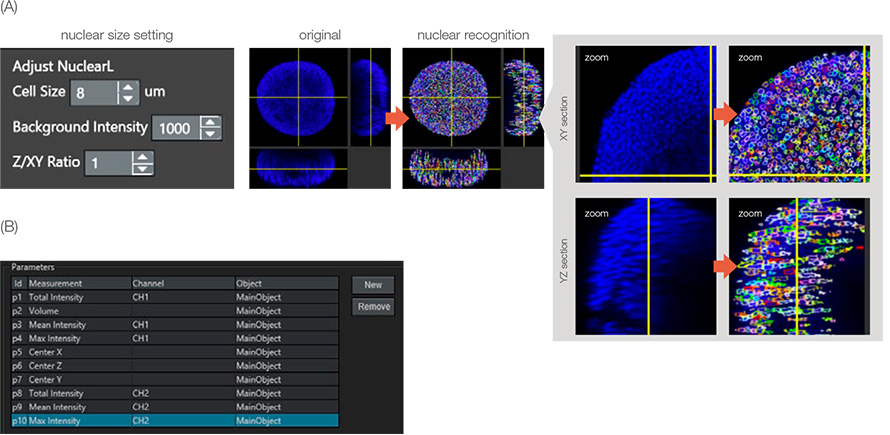
그림 3: 부모 객체의 인식. (A) NuclearL 모듈을 사용하여 핵들을 인식하였습니다. 인식된 각 핵은 다채로운 원으로 둘러싸여 있었습니다. (B) 분석 매개변수를
설정하였습니다. 예를 들어 매개변수 10은 주 객체(핵)에서 CH2(죽은 신호) 최대 강도입니다.
핵 인식을 완료하면 인식된 핵들을 산포도 또는 히스토그램의 객체 데이터로 표시할 수 있습니다. 분석 매개변수에서 산포도의 X 또는 Y 축을 조정할 수
있습니다. 매개변수를 중앙 X/Y에 각기 설정하면 주객체의 중앙 X/Y가 그래프에 표시됩니다(그림 4A). 그래프의 점을 클릭하면 NoviSight 소프트웨어가 원래
이미지의 동일 위치를 가리킬 수 있습니다(그림 4B).
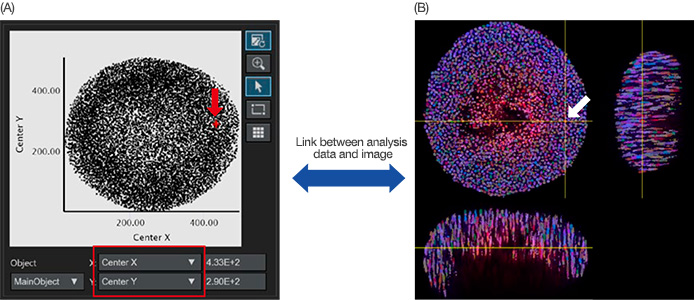
그림 4: 스페로이드의 정량 분석 이미지. (A) 중앙 X 및 Y 분석 매개변수(그림 3B에 설정됨)가 X 및 Y 축에 각기 설정되었을 때 인식된 각 핵이 그래프에
표시되었습니다. 필요에 따라 X/Y 축의 분석 매개변수를 조정할 수 있습니다(빨간색 정사각형). (A/B) NoviSight 소프트웨어는 분석 데이터를 원래 이미지와
시각적으로 연결할 수 있습니다. 그래프에서 인식된 핵을 클릭하면(A, 빨간색 화살표), 동일 위치가 원래 이미지에 자동으로 표시됩니다(흰색 화살표).
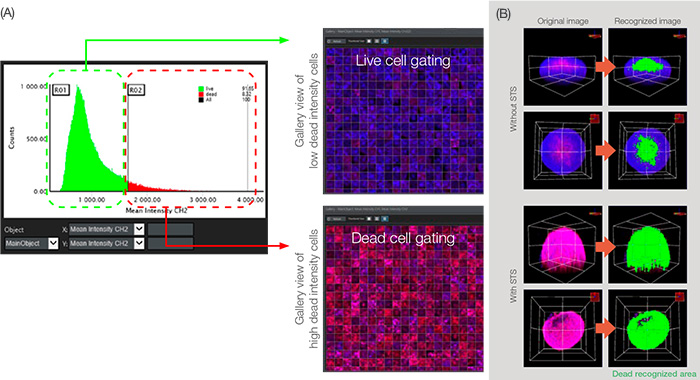
그림 5: 세포 분류를 위한 그래프 분석.
(A) 분석 매개변수 평균 강도 CH2(그림 3B에 설정됨)를 X 및 Y 축에 각기 설정하였을 때 CH2(죽은 신호) 평균 강도 순으로 주 객체 계수(핵)가 그래프에
표시되었습니다. CH2 강도 게이팅을 통해 살아있는 세포와 죽은 세포를 나눌 수 있었습니다. (B) 인식된 죽은 영역의 용적 그림은 녹색으로
표시되었습니다.
통계 분석
NoviSight 소프트웨어는 여러 샘플을 동시에 분석할 수 있습니다. 분석 기반의 정량적 데이터는 열지도(그림 6A)로 표시하거나 추가 분석을 위해 CSV 파일로 내보낼 수 있습니다. 데이터를 각 게이팅에서 출력할 수도 있습니다. 여기서 STS의 다양한 농도가 HT-29 스페로이드 내에서 처리되었을 때 STS는 투여 의존적 방식으로 HT-29 세포 자멸을 유도하였습니다(그림 6B).
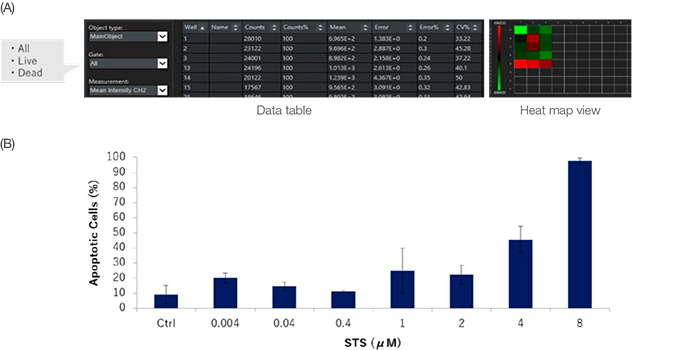
그림 6: STS 적용 또는 미적용 HT-29 스페로이드에 대한 살아있는/죽은 세포 분석의 정량화 결과. (A) NoviSight 소프트웨어가 각 게이트에 대해 정량화 데이터를
출력합니다.
데이터를 열지도로 표시할 수도 있습니다. (B) STS는 투여 의존적 방식으로 HT-29 세포 죽음을 증가시켰습니다.
연결 데이터 및 이미지
데이터와 이미지 간 링크는 대용량 분석의 주요 이점에 해당합니다. 플레이트 리더와 달리 HCA는 세포의 형태, 위치 및 강도를 육안으로 확인하기 위해 원래 이미지를 참조할 수 있습니다. NoviSight™ 소프트웨어는 이러한 기능을 최대한 이용합니다. 예를 들어 다음과 같은 데이터 점이나 객체(그림 7)를 클릭하면 소프트웨어가 해당 이미지를 보여줄 수 있습니다.
- 산포도에서의 점
- 갤러리 뷰에서의 객체 이미지
- 열지도에서의 웰(well)
- 표에서의 수
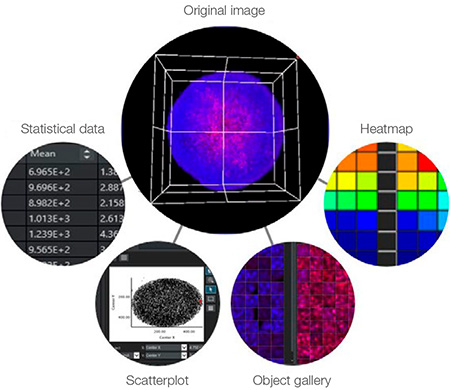
그림 7: 데이터 점이나 객체를 클릭하면 NoviSight 소프트웨어가 해당 이미지의 동일 위치를 보여줄 수 있습니다.
결론
3D 샘플의 3D 정량 분석은 복잡해 보일 수 있지만 NoviSight 소프트웨어는 처음부터 끝까지 워크플로를 간소화할 수 있습니다. 예를 들어 핵 인식은 스페로이드의 살아있는/죽은 분석에 필요한 유일한 매개변수입니다. 이 간소화된 워크플로는 관련된 3D 모델에도 적용될 수 있습니다.
저자
Mayu Ogawa(Olympus)
이 애플리케이션에 사용되는 제품
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
Not Available in Your Country
Sorry, this page is not
available in your country.