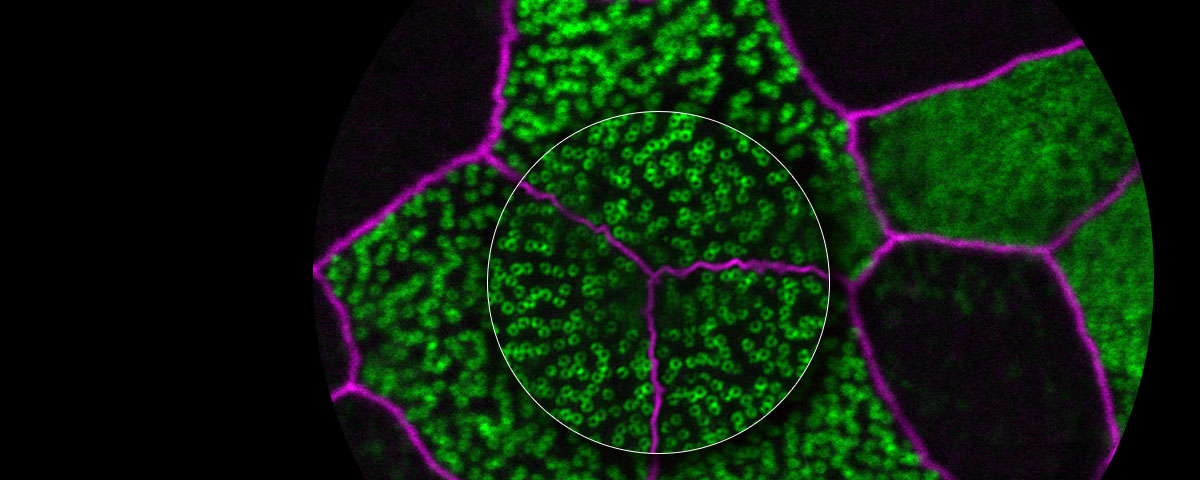
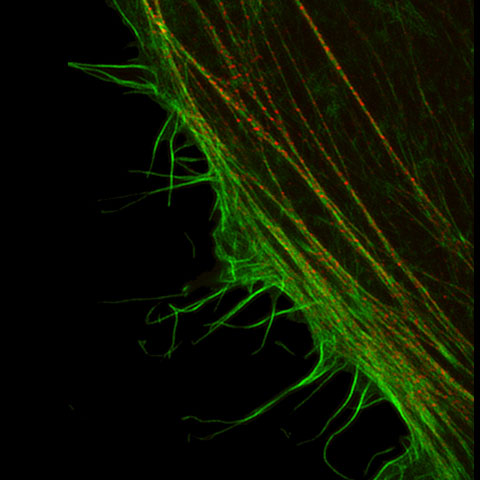
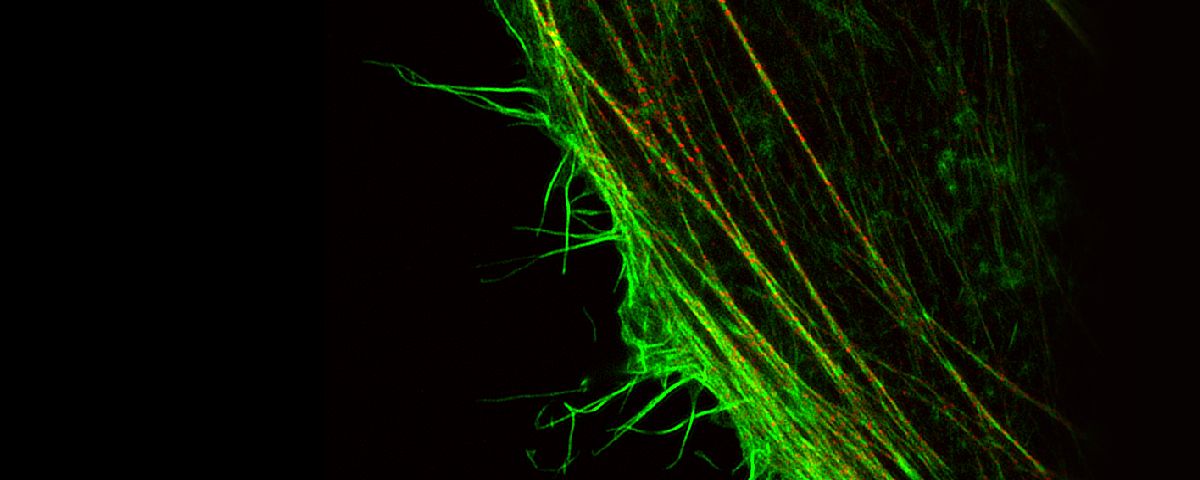
共焦点観察で約120nmの解像を実現
蛍光顕微鏡は蛍光プローブを用いることで生体内の特定のタンパク質などを識別できます。蛍光顕微鏡の分解能は回折限界で決まるおおよそ200nmが限界とされ、それ以上細かい構造を観察することは不可能でした。オリンパスは独自の超解像技術(OSR: Olympus Super Resolution)による水平方向約120nmの分解能と共焦点効果で、クリアな超解像画像を取得できるソリューションを提供します。
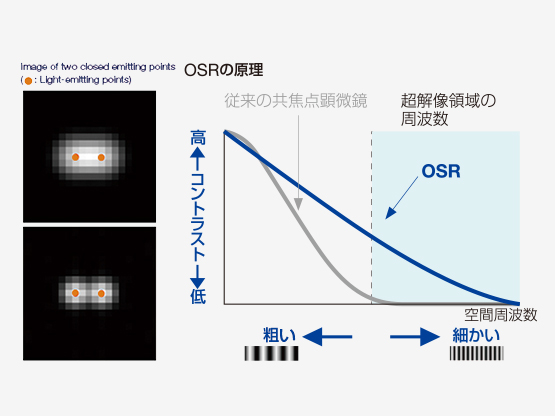
独自技術Olympus Super Resolution (OSR) の原理
| 画像とは様々な空間周波数の集まりにより構成され、分解能が高い画像とは高い周波数成分を有していることを意味しています。共焦点光学系はボケ像を取り除き焦点面のみの情報を取り出すだけでなく、従来の分解能を超える超解像領域の周波数を検出できますが、この超解像領域のコントラストは低く、可視化することが困難でした。Olympus Super Resolution(OSR)技術は、超解像領域をできるだけ画像の中に取り込むために光学系を最適化すると同時に、コントラストの低い超解像領域を本来のコントラストに高速に回復させるオリンパス独自のオプトエレクトロニクス技術により、水平分解能120nmを実現することができます。そのため、特別な蛍光色素を必要とせず、従来の蛍光色素が使用できるため、マルチカラーの超解像イメージングが手軽に実現できます。 |
Olympus Super Resolution (OSR)
SpinSR10
FV3000(FV-OSR)
|
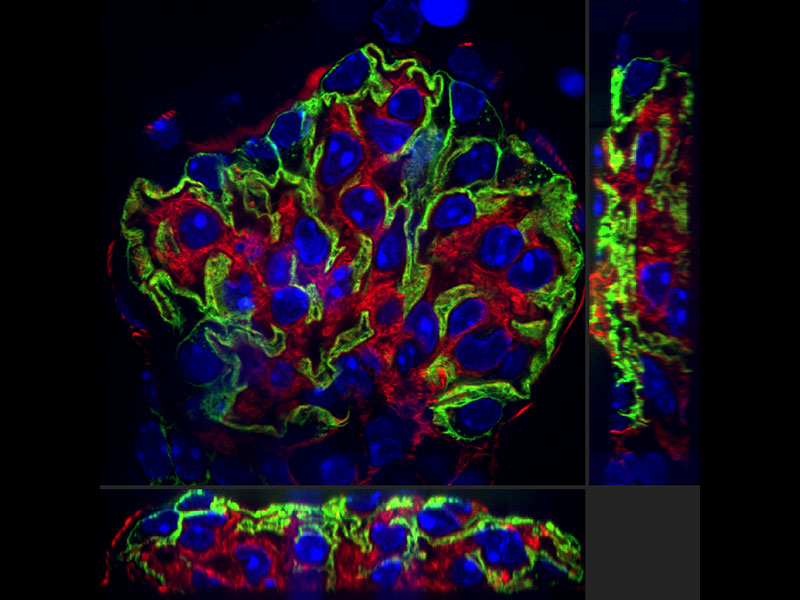
デコンボリューションでより鮮明な画像を
散光によるボケ部分に邪魔されて不鮮明だった画像をボケのない画像にするデコンボリューション技術。特にZ軸方向に発生するボケを取り除き鮮明な画像を構築する3D強制反復デコンボリューションはよりきれいな画像を提供します。
|
|---|
先生からのコメント
国立研究開発法人理化学研究所 生命システム研究センター 細胞極性統御研究チーム チームリーダー 岡田 康志 先生 | 細胞内のオルガネラや超分子複合体の多くは100nm程度の大きさであり、従来の光学顕微鏡では、その構造を観察することは出来ませんでした。近年、様々な超解像顕微鏡法が開発されましたが、特殊な蛍光色素や観察条件あるいは特殊な光学系が必要で、細胞生物学の現場で手軽に利用することは困難でした。 今回のオリンパスの超解像顕微鏡*は、スピニングディスク共焦点顕微鏡から簡単にアップグレードできる方法です。さらにシリコーン浸対物レンズとの組み合わせで球面収差による劣化を抑えて細胞深部まで超解像ライブイメージングが実現可能であり、細胞生物学分野の様々なアプリケーションに対応できるツールとなると期待しています。 *本技術は理化学研究所との共同研究による成果に基いています。
|
大阪大学大学院 生命機能研究科・医学系研究科 月田 早智子 先生 | 気管多繊毛上皮細胞アピカル面での繊毛配列秩序形成過程について、繊毛根元の基底小体の配列と方向性の発生・分化に伴う変化を追うことで、検討を進めています。気管初代培養系において、アピカル表面に多くの動く繊毛を有する多繊毛上皮細胞の発生・分化過程は一週間に及び、その間、基底小体の配列と方向性を明確に解像するためには、XYZ軸各々で200nm以下の解像度が必要とされます。 SpinSR顕微鏡を用いたライブセルイメージングシステムで、基底小体の動きについての解析が可能になり、繊毛配列秩序形成メカニズムについて、有用な新規所見を得ることができるようになりました。 |
東京大学 大学院薬学系研究科 薬品作用学教室 池谷 裕二 先生 | SpinSR10を使用する前から何となく想像はしていましたが、それでも初めて超解像の蛍光画像を見たときには映像美に驚きました。私は、脳のなかでもとくにシナプス後部(スパイン)や神経線維、グリア細胞突起などの微細構造の可塑性を研究しています。このSpinSR10を通して得られた映像で、ようやく「見えた」と実感するに至りました。電子顕微鏡以外で微細構造をつぶさに観察できる方法を手にしたことで、得られるデータの信頼性だけでなく、データ取得の手順も格段に減り、実験のスループットが大幅に高まりました。さらに、同一組織からの長時間にわたる経時観察も実現され、新たな研究の道筋も生まれています。 |
参考文献
S. Hayashi and Y. Okada, “Ultrafast superresolution fluorescence imaging with spinning disk confocal microscope optics,” Mol. Biol. Cell 26(9), 1743–1751 (2015).
S. Hayashi, “Resolution doubling using confocal microscopy via analogy with structured illumination microscopy,” Jpn. J. Appl. Phys. 55(8), 082501 (2016).
A. Nagasawa-Masuda and K. Terai, “Yap/Taz transcriptional activity is essential for vascular regression via Ctgf expression and actin polymerization,” PLoS ONE 12(4), e0174633 (2017).
H. Nakajima, et al., “Flow-Dependent Endothelial YAP Regulation Contributes to Vessel Maintenance,” Dev. Cell 40(6), 523-536.e6 (2017).
K. Tateishi, et al., “Three-dimensional Organization of Layered Apical Cytoskeletal Networks Associated with Mouse Airway Tissue Development,” Sci. Rep. 7, 43783 (2017).
E. Herawati, et al., “Multiciliated cell basal bodies align in stereotypical patterns coordinated by the apical cytoskeleton,” J. Cell Biol. 214(5) 571-586 (2016).
M.-T. Ke, et al., “Super-Resolution Mapping of Neuronal Circuitry With an Index-Optimized Clearing Agent,” Cell Rep. 14(11) 2718–2732 (2016).
このページはお住まいの地域ではご覧いただくことはできません。
このページはお住まいの地域ではご覧いただくことはできません。
.jpg?rev=2087)