蛍光観察は、細胞の複雑な構造を見事な色使いで表すことがでいます。しかし場合によっては、蛍光染色することなく、必要な情報をより速く、より少ないダメージで、あるいはより簡単に取得することができます。
ディープラーニングを活用したソフトウェアがこれを可能にします。このソフトウェアのニューラルネットワークは、明視野画像から細胞と核を自動的に認識することができます。また、自己学習によって、時間とともに性能を向上させることも可能です。
これをセルフラーニングマイクロスコピーと名付け、この概念を当社のライフサイエンス用のイメージングソフトウェアcellSens の最新バージョンに実装しました。以下に、セルフラーニングマイクロスコピーの活用法について具体的に示します。
DAPIからディープラーニングへ:実験
細胞核のセグメンテーションとカウントを行うための確立された技術では、DAPIで核を標識し、蛍光画像を解析します(図1、右)。ここでは、cellSensにディープラーニングモジュールを用いて、非染色の明視野画像(図1、左)を解析した場合に同じ結果が得られるかどうかを検証する実験を行いました。
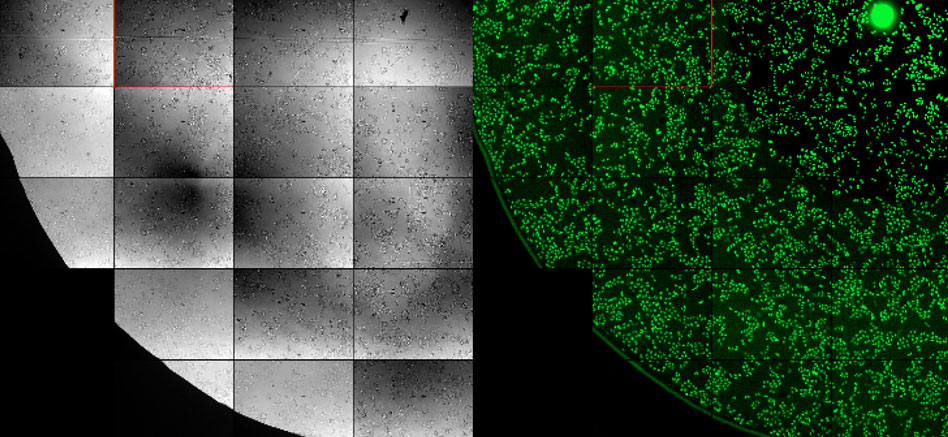
図1:細胞核の明視野画像(左)と蛍光画像(右)
ソフトウェアの学習のため、96ウェルプレートの40箇所の蛍光画像と明視野画像を用意しました。セルフラーニングマイクロスコピーにより、学習は完全自動で、所要時間は約90分で済みました。
結果
図2は、蛍光を用いて検出し、ランダムに選んだ100個の核(左)および対応する明視野画像(中央)、ディープラーニングで予測したオブジェクトの形状(右)を示しています。この図では、2つの方法にかなりの一致が認められますが、ディープラーニングで検出されたオブジェクトの総数の方が3%程度多くなっていました。

図2:蛍光を用いて検出した核(左)および対応する明視野画像(中央)、ディープラーニングで予測したオブジェクトの形状(右)
この差を調べてみると、蛍光の検出結果は不自然に大きなオブジェクトを多く含んでいることがわかりました。該当する画像を比較したところ、大きなオブジェクトの多くにおいて、実際は2つの核が1つのオブジェクトとしてカウントされており、ディープラーニングソフトウェアではこれらのオブジェクトはほとんどが2つの異なるオブジェクトとして正しく検出されていたことがわかりました(図3)。
図3:蛍光画像(左)と比べると、ディープラーニングソフトウェアでは近接した核を互いに明確に区別している(右)
セルフラーニングマイクロスコピーの実験から得られた重要な教訓
この実験では、ディープラーニング技術に基づいた画像解析方法により、蛍光を用いた方法と同等の性能を得やすいだけでなく、それ以上の性能が発揮されることも示しています。さらに、高精度なオブジェクト検出のためのニューラルネットワークの学習は、素早く自動的に行われます。
ライブセルイメージングにおける利点は明らかです。精度の向上に加え、ディープラーニングによる画像解析は、遺伝子組換え(GFP)や核マーカーが不用であることも意味しています。結果として、標本作製にかかる時間を短縮し、蛍光チャンネルを他のマーカーのために利用でき、短い露出時間で光毒性を低減することができます。



