インスタンスセグメンテーションとは、画像をオブジェクトの個々のインスタンス(個々の細胞や核)に分割するタスクです。 タスクには後処理作業が必要でしたが、新しくなった当社のTruAI™ディープラーニングテクノロジーでは、インスタンスセグメンテーションが大幅に簡単になりました。
このブログでは、さまざまな画像例を通して、この簡単になったインスタンスセグメンテーション法について説明します。 顕微鏡画像解析における利点と、可能になる新たな機能について見ていきましょう。
顕微鏡画像でのインスタンスセグメンテーションの仕組み
顕微鏡画像の自動解析では、通常、セグメンテーションが必要です。セグメンテーションとは、画像をオブジェクト、パーツ、背景に分割することです。 比較的簡単なセグメンテーションアプリケーションで、はっきり分かれた核の画像があります(図1を参照)。
図1. DAPI染色によって可視化された核を持つBPAE細胞サンプル。 サンプルの画像取得には、FLUOVIEW™ FV3000共焦点顕微鏡(倍率20x)が使用されました。 サンプルが部分的に退色した結果、蛍光信号がさまざまなレベルになっています。 TruAIテクノロジーの機能を示すため、積分時間は低く設定されました。
この場合でも、ノイズのある核や幅広い信号レベルにわたる核のセグメンテーションに、TruAIテクノロジーを使用することは理にかなっています。 TruAIテクノロジーによって、この標準的なアプリケーションを楽に処理できます(図2を参照)。 画像は、前景と背景という2つのクラスにセグメント化されます。 TruAIテクノロジーでは、必要な場合に最大16の前景クラスを検出可能です。
図2. 図1と同じ画像に、TruAI確率マップを重ね合わせたもの(核のピクセルが赤色で示されている)。
重なったりくっつき合ったりする核は、個々の核に分割するために簡単な後処理ステップが必要になる場合があります。 ニューラルネットワークはすべてのピクセルを核または背景のいずれかとして検出しますが、くっつき合った核の間には、別々のオブジェクトとして区別するための背景ピクセルがないからです。 個々の核のセグメンテーションをインスタンスセグメンテーションといいます。
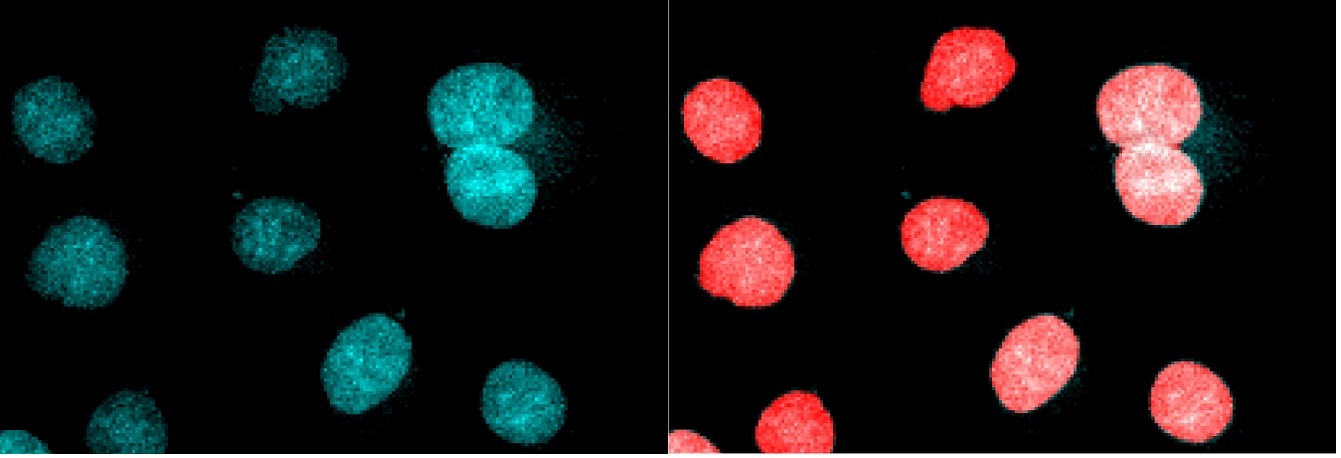
図3. 図1(左)と図2(右)の詳細図。 核ピクセルのセグメンテーションは大変良好ですが、くっつき合った核は後処理ステップで分ける必要があります。
セグメンテーションは、コンフルエント状態の細胞など、もっと複雑な解析タスクにも有用です(図4を参照)。 例えば、細胞で覆われた領域の計測にセグメンテーションを使用できます。
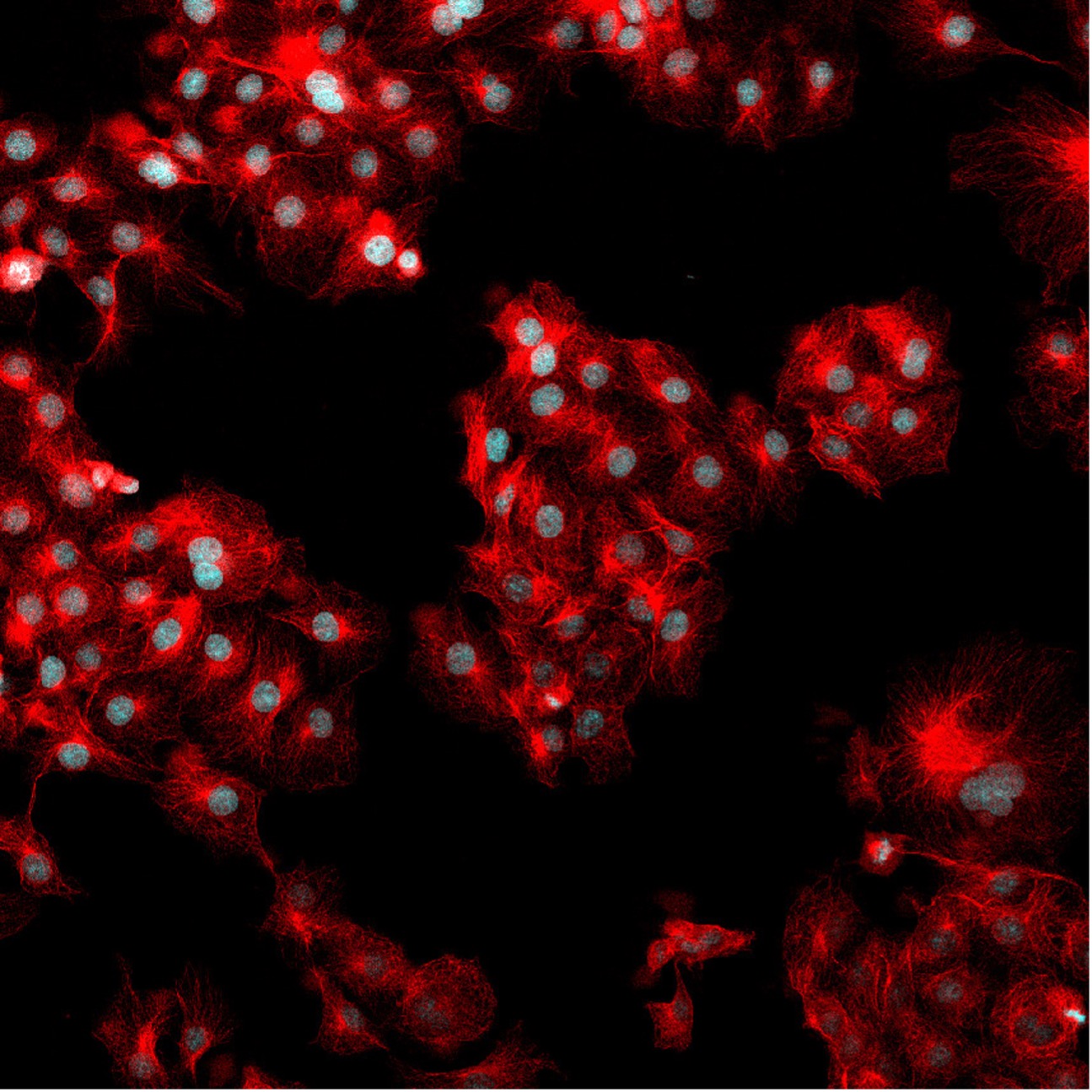
図4. 図1と同じ視野ながら、F-アクチンをマーキングする488ファロイジンの蛍光信号も示されています。
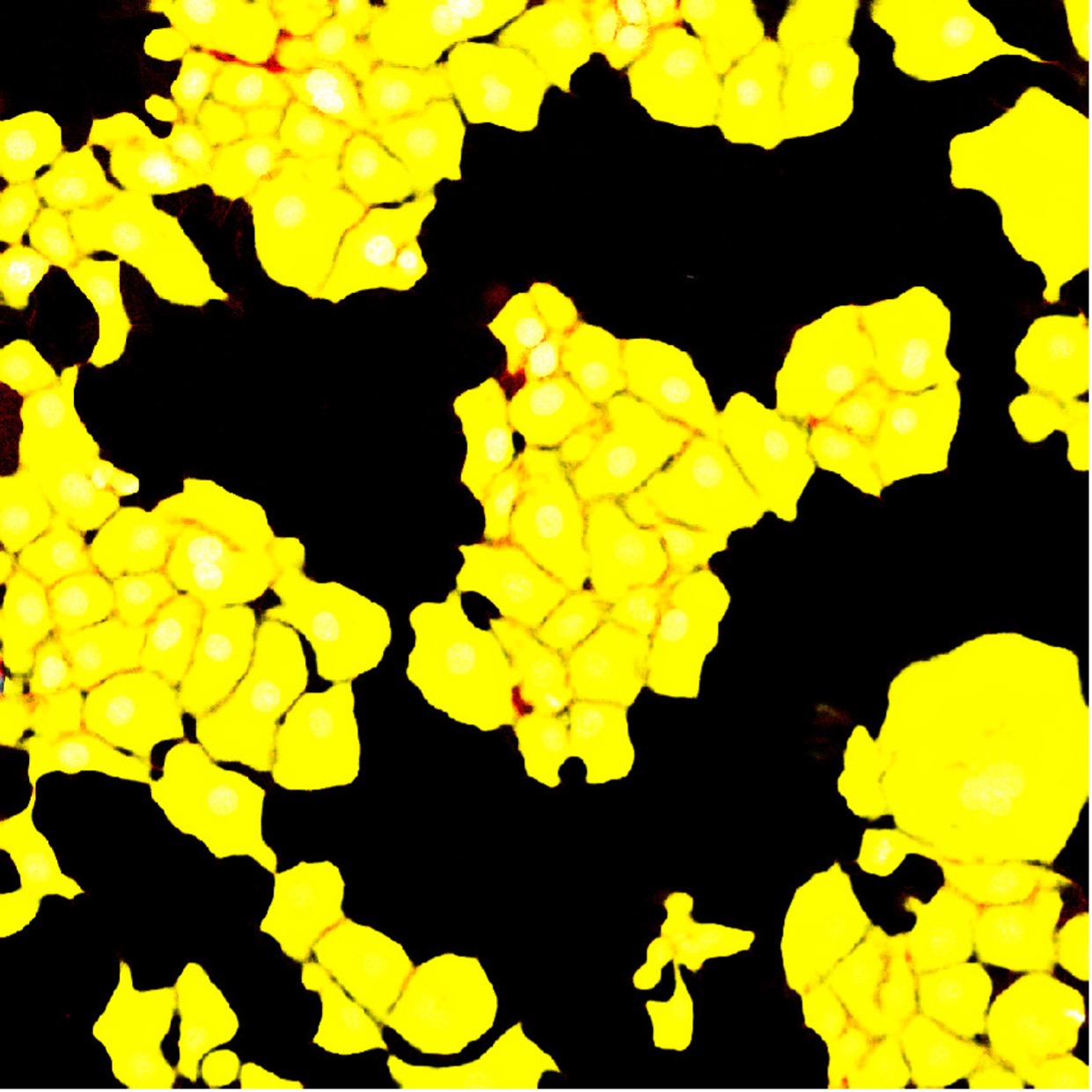
図5. 標準的なセグメンテーションのニューラルネットワークによるF-アクチンチャンネルのセグメンテーション結果。 ニューラルネットワークはくっつき合った細胞間に1 pxの境界を設けるようトレーニングされていますが、セグメンテーション結果では個々の細胞が明確に分かれていません。
また一方で、セグメント化された細胞領域を個々の細胞に分ける(インスタンスセグメンテーション)のは、難しい後処理作業になる可能性があります(図6を参照)。
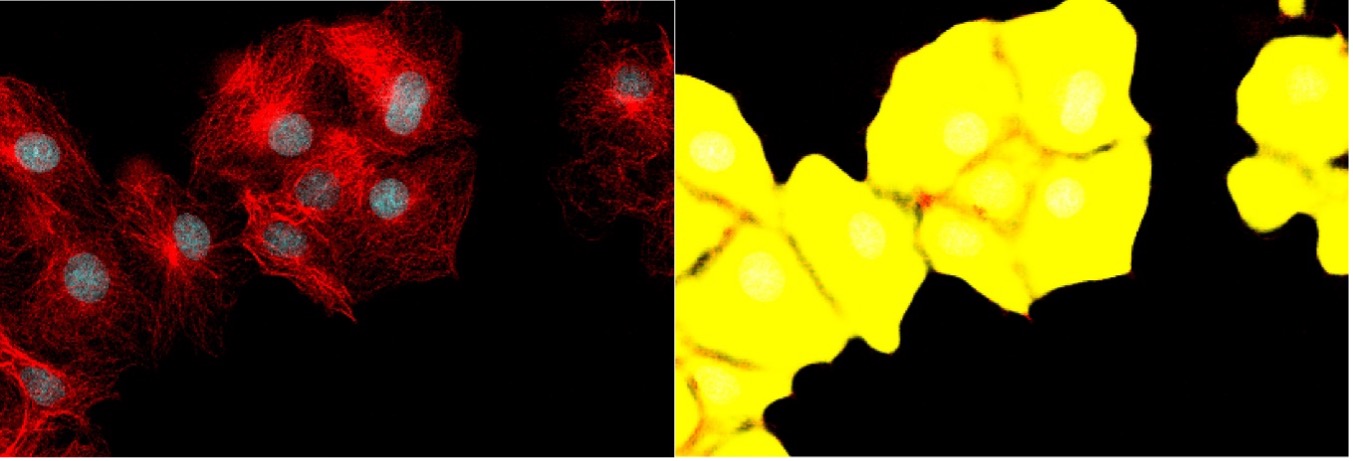
図6. 図4(左)と図5(右)の詳細図。 隣接した細胞間の境界は、前景クラスに対する確率の低さを示しますが、個々の細胞の区分には経験豊富なユーザーによる複雑な後処理が必要になります。
多様な複雑さのある画像に対してこのワークフローを簡単にするため、scanRスクリーニングステーションのTruAIテクノロジーに、新たなインスタンスセグメンテーション機能が追加されました。*
このインスタンスセグメンテーション機能はAIをベースにしており、手動の後処理ステップやパラメーターの調整は必要ありません。 ニューラルネットワークモデルのトレーニングが終われば、クリック1つで新しい画像に適用して、すぐに解析結果が得られます(図7を参照)。
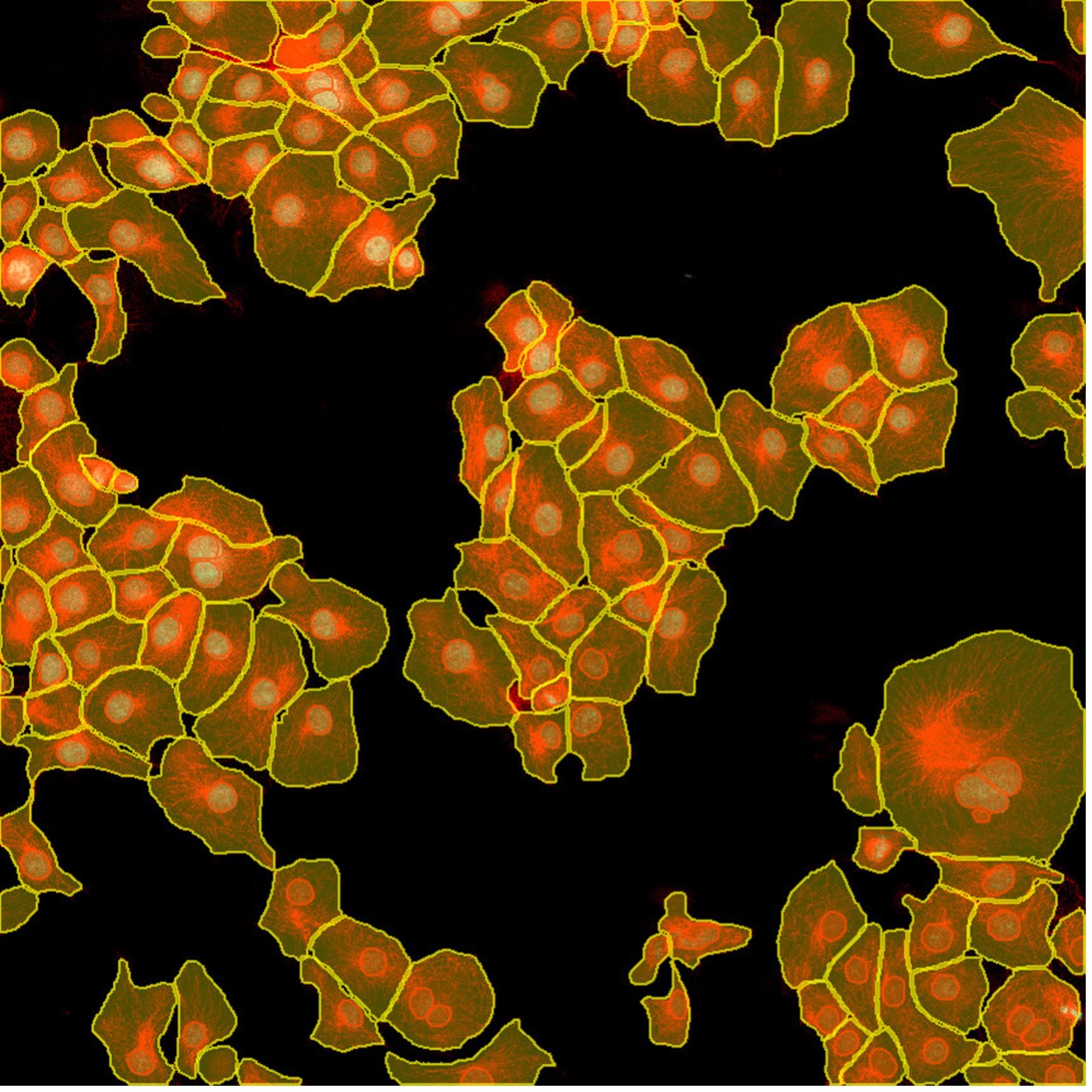
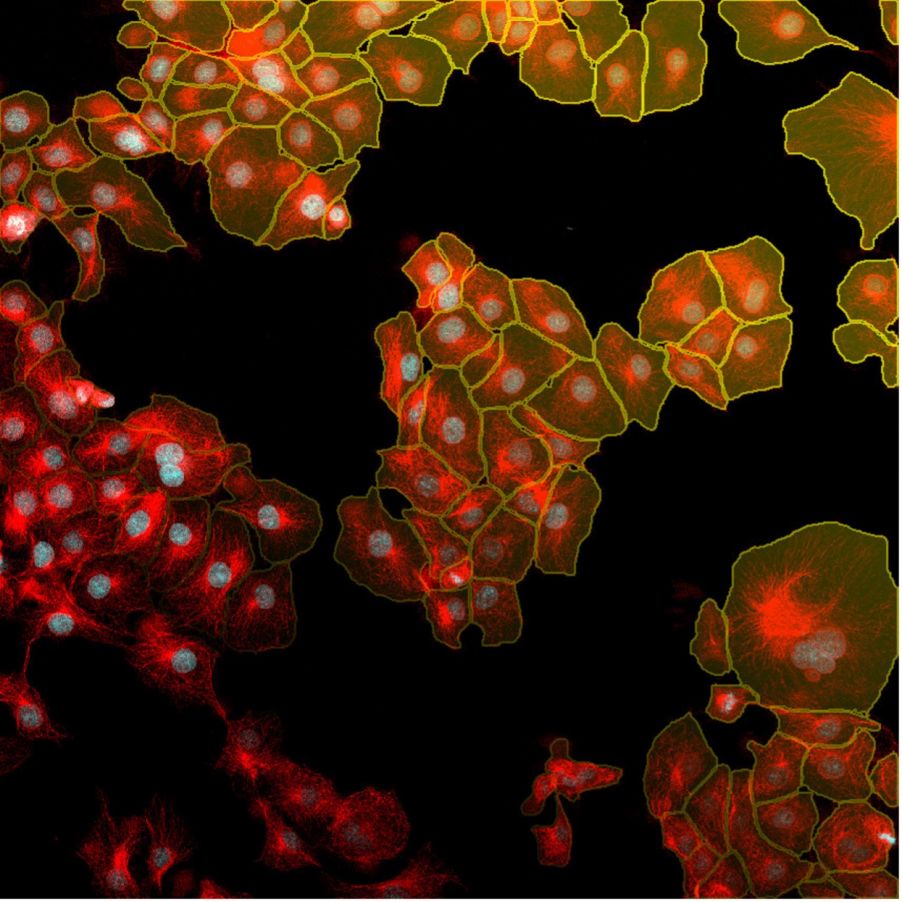
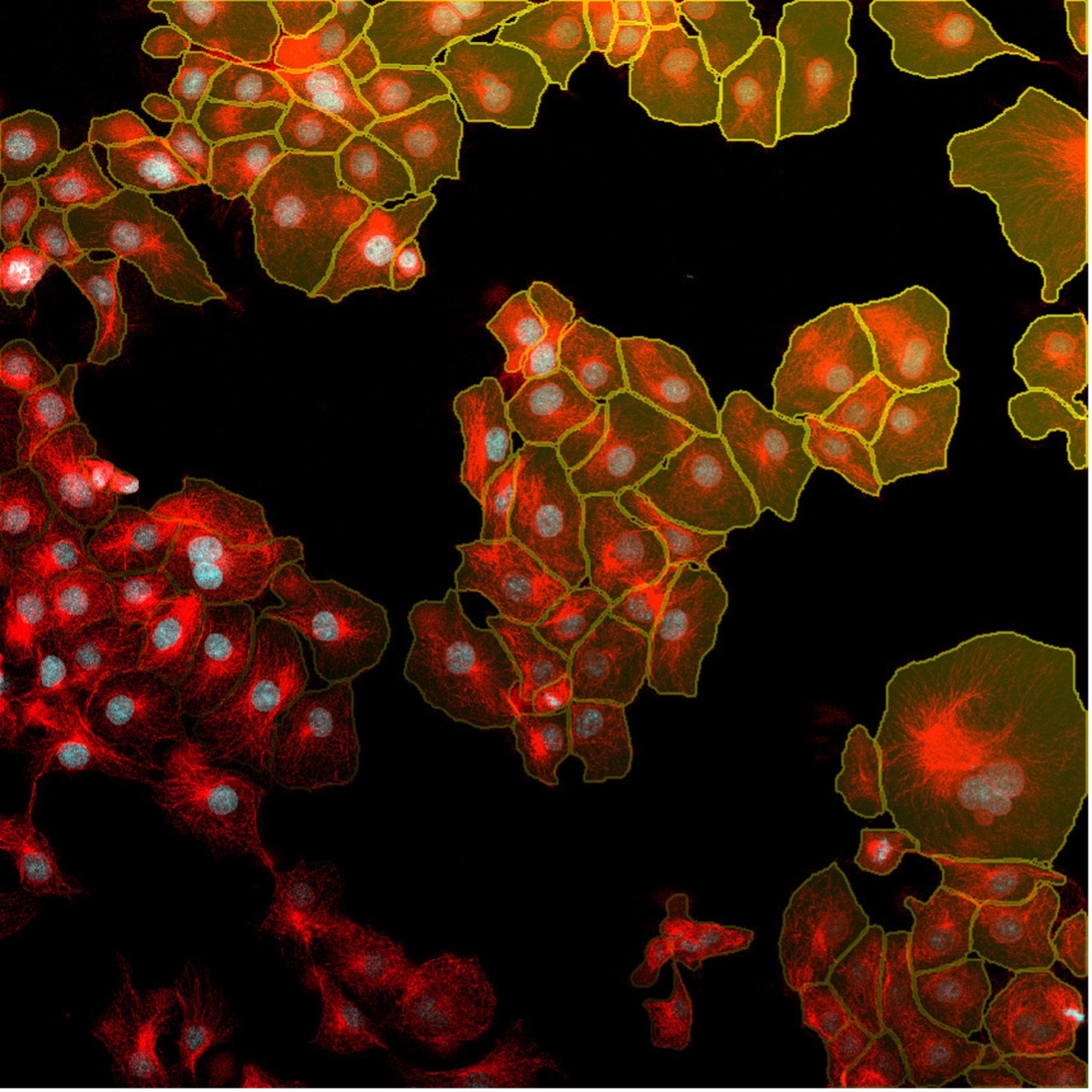
図7. TruAIインスタンスセグメンテーション機能を使用すれば、クリック1つで核(赤色)と細胞(黄色)の検出結果が得られます。 この簡単なインスタンスセグメンテーション法に後処理ステップは必要ありません。
下の図8は、TruAIインスタンスセグメンテーションによって、後処理ステップの必要なく細胞と核の両インスタンスが直接表示される様子を示しています。
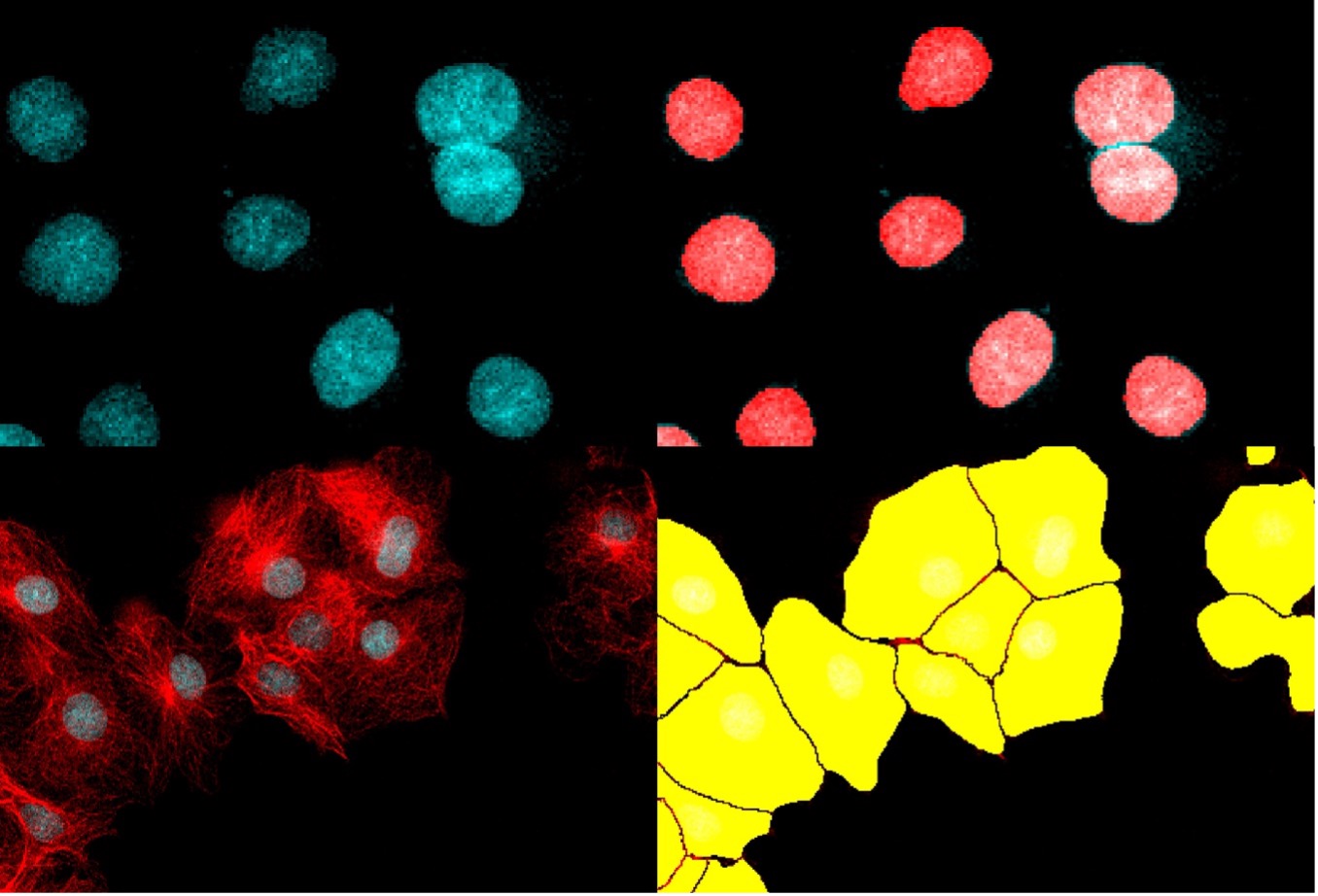
図8. TruAIインスタンスセグメンテーションを使用すると、ニューラルネットワークによって核(上)と細胞(下)の正確な区分が直接出力されます。
下の図9は、標準的なセグメンテーションを使用した場合とTruAIインスタンスセグメンテーションを使用した場合の、細胞区分結果を 比較したものです。
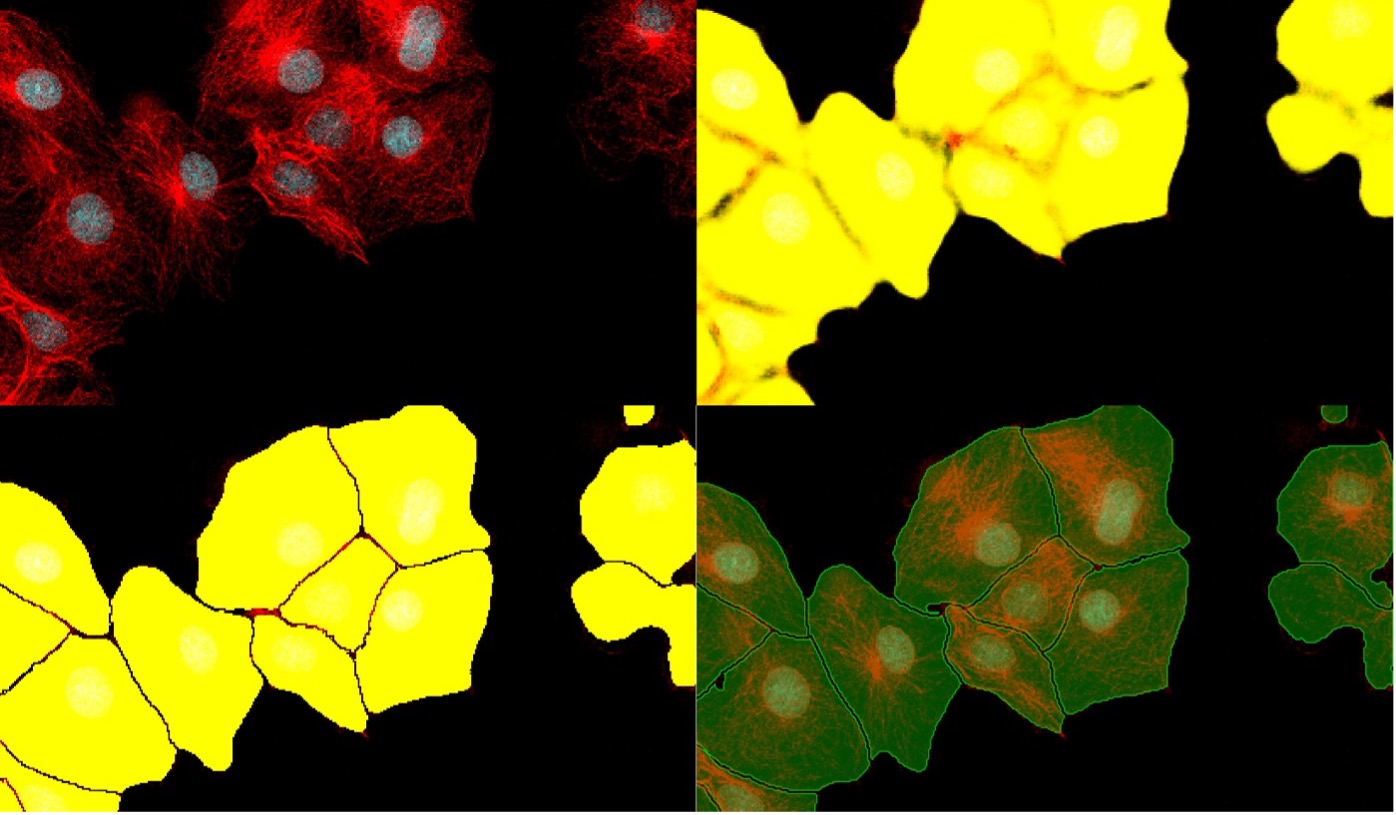
図9. 複雑な画像(左上)を解析した場合の、標準的なセグメンテーション(右上)からTruAIインスタンスセグメンテーション(左下)への進化。 結果(右下)ではほぼ完璧に細胞が区分されています。
AIに基づくセグメンテーション:顕微鏡画像解析の強力なツールセット
新しくなったTruAIテクノロジーは、幅広い画像解析アプリケーションにおいて、個々のニューラルネットワークモデルをトレーニングするための強力なツールセットを提供します。 最適なニューラルネットワークのトレーニングには、注意深くラベリングされた例(当社の使いやすいソフトウェアで対応可能)が必要な一方で、トレーニングされたニューラルネットワークを新たな画像に適用するのは簡単で安定したタスクです。
TruAIインスタンスセグメンテーションでは、標準的なアプリケーション(核や細胞の検出など)に対して信頼性の高い事前トレーニングされたディープラーニングモデルを提供することもできます。 事前トレーニングされたモデルの詳細と例については、今後のブログをチェックしてください。
それまでの間は、以下に挙げるインスタンスセグメンテーションのアプリケーション例をご覧ください。
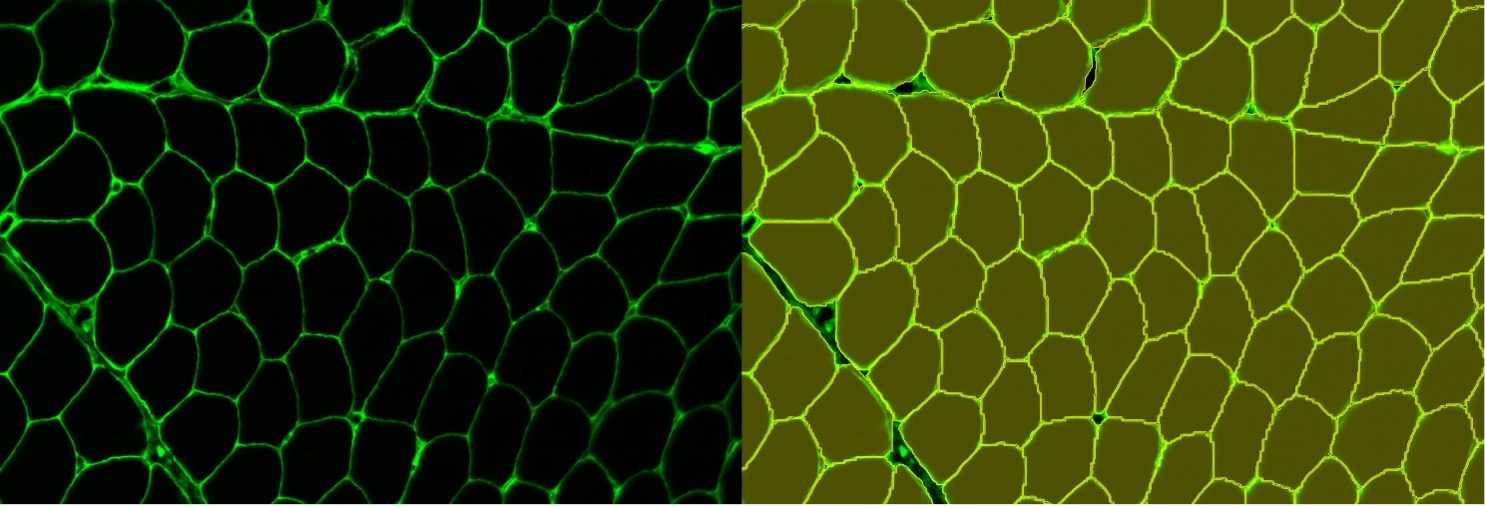
図10. 筋線維染色のインスタンスセグメンテーション
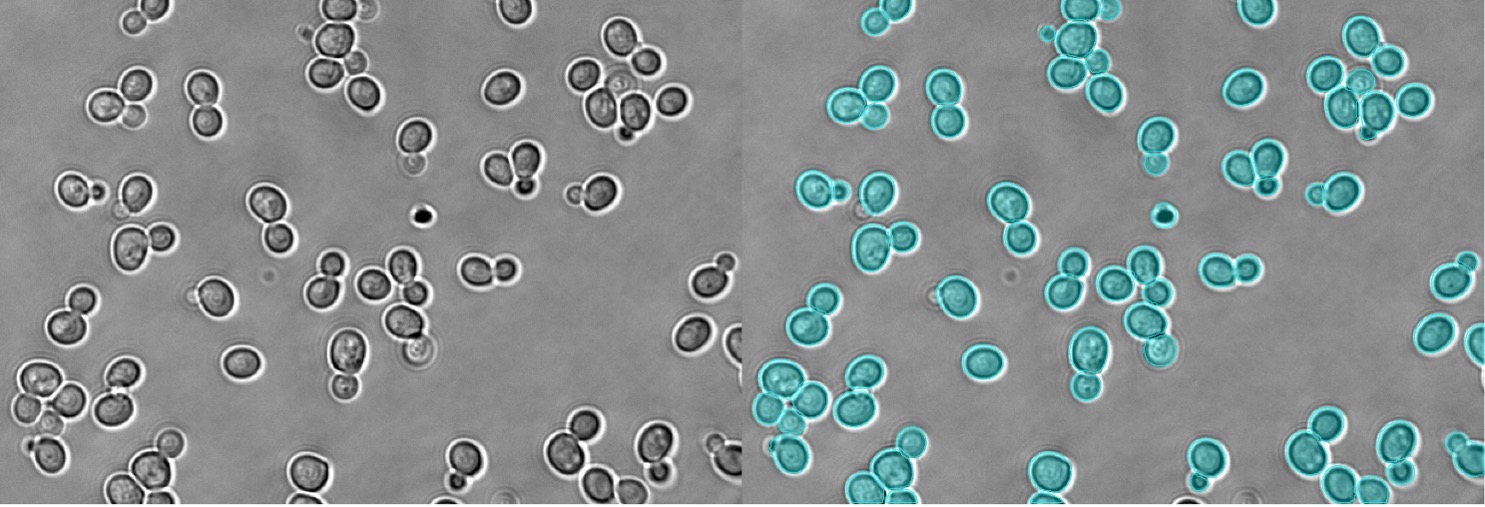
図11. 明視野で観察した酵母のインスタンスセグメンテーション
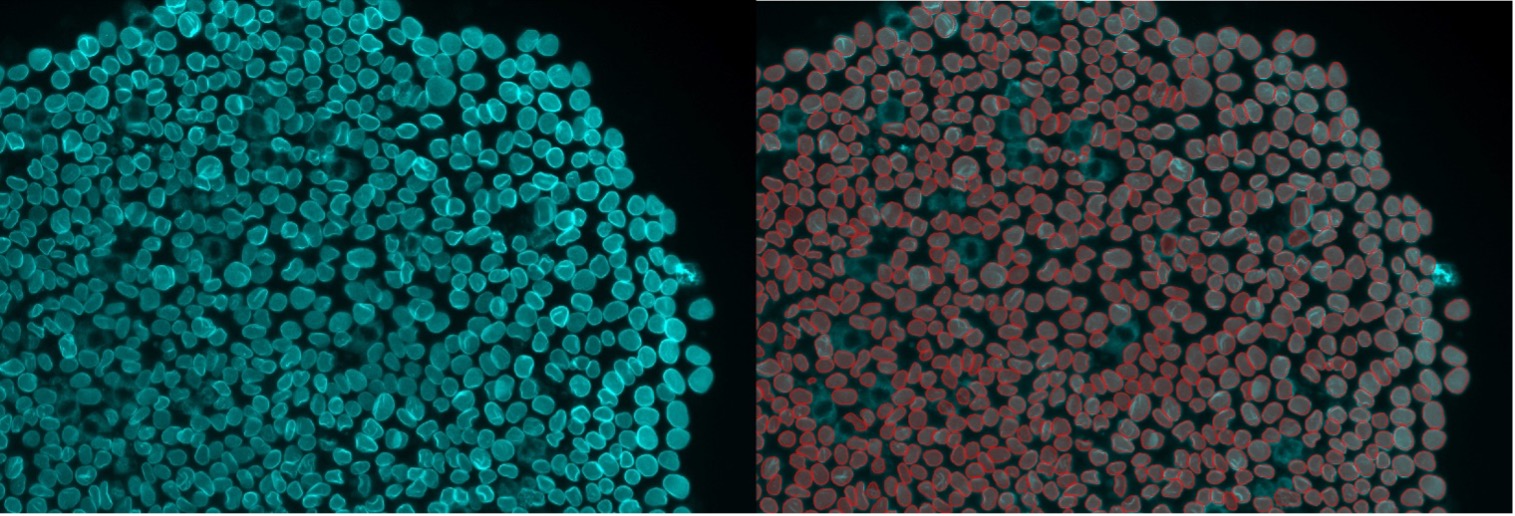
図12. 核膜染色のインスタンスセグメンテーション
*scanRシステムは日本では販売されていません。
関連コンテンツ
ビデオ:セルフラーニング観察法に生かされるディープラーニングの力