Dans ce webinaire, Manoel et Shohei, nos experts en apprentissage profond et en criblage à haut contenu, présenteront le concepteur d’essai du système scanR. Le système scanR, plateforme dédiée au criblage à haut contenu d’Olympus, adopte une approche unique de navigation dans les données optiques de l’échantillon et d’analyse inspirée de la cytométrie de flux.
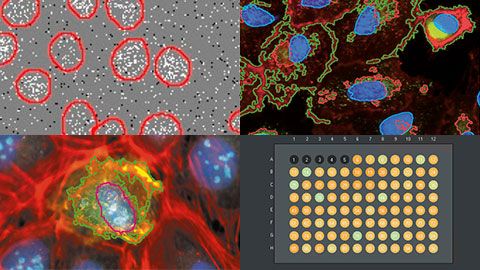
Foire aux questionsFoire aux questions du webinaire | Criblage à haut contenu : l’analyse personnalisée simplifiéeCertaines cellules présentent deux couleurs différentes (par exemple, vert et bleu), ce qui suggère qu’elles se trouvent dans deux stades différents du cycle cellulaire. Est-ce une indication que le modèle ne fonctionne pas correctement ?Le système peut distinguer plusieurs couleurs/canaux. Ainsi, lorsque deux couleurs différentes sont présentes, le logiciel distingue l’arrière-plan du premier plan (le premier plan étant la couche cellulaire). Cependant, comme certains stades du cycle cellulaire peuvent être identifiés par certaines caractéristiques morphologiques, nous suggérons fortement d’utiliser une étiquette de stade cellulaire pour marquer les différents stades cellulaires. Il s’agit d’une étape à part entière pour s’assurer que l’entraînement (l’apprentissage profond) est effectué avec chacun des stades du cycle cellulaire que vous souhaitez différencier. Néanmoins, comme les cellules sont évaluées par pixel et non « par cellule », le système sera dans certains cas capable de discerner automatiquement un stade du cycle cellulaire, même sans entraînement avec ce stade. Comment le système se comporte-t-il lors de l’analyse de cultures de sphéroïdes ou d’autres cultures ?Le système scanR a été conçu pour fonctionner efficacement avec des cultures de sphéroïdes et d’autres types de culture. Nous avons observé que certains détails de la procédure fonctionnent correctement avec cette application. Tout d’abord, nous vous recommandons d’appliquer la déconvolution 3D aux images recueillies, puis d’effectuer une annotation manuelle. Un exemple de procédure pourrait consister à utiliser de 10 à 20 images et à segmenter manuellement 10 cellules. Est-il possible de produire une image des bordures des puits dans des plaques à 384 puits ?Oui, mais avec quelques contraintes. La capacité à produire des images des bordures des puits dépend de la combinaison du type de plaque à puits et de l’objectif utilisé. Les plaques à puits se présentent sous différentes formes : les plaques à jupe, les plaques à demi-jupe et les plaques sans jupe. Par conséquent, un facteur important pour l’imagerie des bords de puits est la différence de hauteur entre le fond de la plaque et le bas de la jupe. Il y a donc des plaques dans lesquelles la distance verticale entre le fond de la plaque et le bas de la jupe empêche l’imagerie avec des objectifs à fort grossissement, car l’objectif risque d’entrer en collision avec la plaque. Dans un tel cas, nous suggérons fortement d’utiliser un objectif à longue distance de travail. Olympus offre une large gamme d’objectifs à longue distance de travail. Comment la mise au point s’effectue-t-elle dans le système scanR ?Le système scanR utilise une combinaison de mise au point automatique matérielle et logicielle. La mise au point matérielle détecte la surface inférieure du fond du puits de la plaque, et la mise au point logicielle localise l’échantillon. Ce processus est particulièrement fiable ; vous n’avez pas besoin de rester à côté du système une fois que vous avez lancé l’expérience. Peut-on utiliser le système à disque rotatif à super-résolution pour faire du criblage à haut contenu ?Oui, vous pouvez utiliser le logiciel scanR pour contrôler le système de microscope IXplore SpinSR d’Olympus. Avec ce système, il est possible d’atteindre une résolution de 120 nm, puis d’importer les images acquises dans le logiciel cellSens en vue d’un traitement plus poussé pour obtenir des résultats encore meilleurs. Quelle est la performance de la segmentation et de l’apprentissage profond sur les tranches de tissu ?D’après notre expérience, la segmentation fonctionne convenablement sur les tranches de tissu, en particulier lorsqu’on utilise le système à disque rotatif. Cependant, il est possible d’acquérir de bonnes images sans le système à disque rotatif en appliquant simplement la déconvolution en 3D. Si les tissus sont particulièrement difficiles à analyser, l’apprentissage profond avec annotation manuelle s’avère efficace. L’annotation manuelle fournit à l’algorithme d’apprentissage profond un ensemble d’entraînement robuste à partir duquel il peut apprendre et mieux prédire les segments sur la base de l’identification de l’utilisateur. |