Prédiction des classes de phénotypes nucléaires à l’aide de la technologie d’apprentissage profond dans le cadre d’essais de médicaments
Introduction
La quantification des mouvements dynamiques des protéines, comme ceux des récepteurs en réponse à des médicaments, nécessite de solides méthodes de segmentation nucléaire utilisant des fluorophores et des colorants. Ce processus a toutefois ses limites. En effet, les procédures de marquage des cellules vivantes sont chronophages et onéreuses et peuvent entraîner une phototoxicité, un photoblanchiment et une désactivation des composés photosensibles (par ex., R18811).
Pour contourner ces obstacles, nous avons mis au point un réseau neuronal (NN) à l’aide du logiciel d’imagerie cellSens™ et de la technologie d’apprentissage profond TruAI™ en vue d’identifier les noyaux cellulaires sans utiliser de marqueurs fluorescents. Ensuite, nous avons entraîné le réseau neuronal afin de lui apprendre à faire la distinction entre les différents phénotypes cellulaires de réponse à des médicaments. Dans le cas présent, nous utilisons la procédure de réseau neuronal pour prédire les mouvements intracellulaires du récepteur des androgènes (AR) dans des cellules cancéreuses prostatiques vivantes traitées avec des médicaments ciblant le récepteur des androgènes.
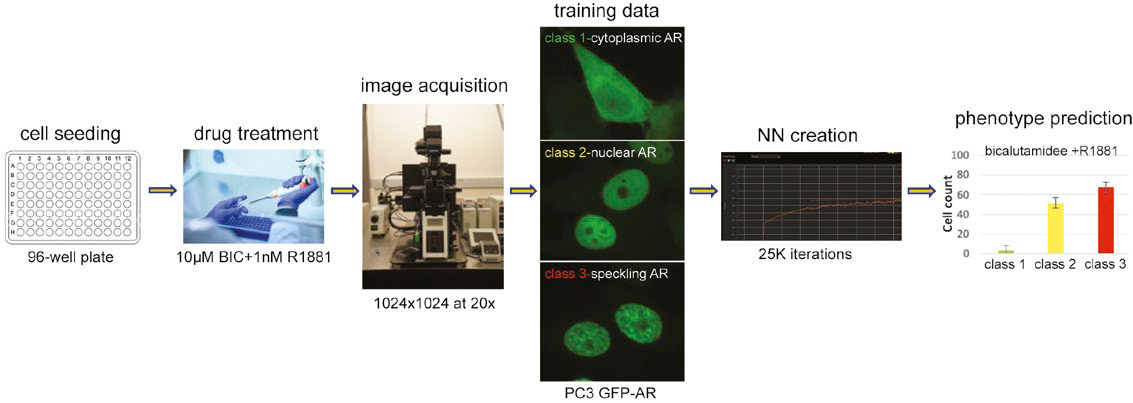
Figure 1 : Procédure permettant de prédire les classes de phénotypes nucléaires à l’aide d’un réseau neuronal entraîné.
Une fois activé, le récepteur des androgènes cytoplasmique migre vers le noyau où il se concentre au niveau de sites de transcription actifs, qui apparaissent sous la forme de points distincts en microscopie confocale (moucheture). Pour prédire les mouvements du récepteur des androgènes (AR), nous avons tout d’abord généré un réseau neuronal capable de détecter les noyaux cellulaires sans recourir à des marqueurs nucléaires. Ensuite, nous avons utilisé une protéine de fusion GFP-AR pour classer les cellules en fonction de l’état de translocation de la protéine GFP-AR dans le noyau et de transactivation par GFP-AR (moucheture), deux essais essentiels pour tester la capacité des composés à cibler le récepteur des androgènes. Enfin, nous avons démontré que notre réseau neuronal était en mesure de prédire ces phénotypes critiques du récepteur des androgènes sans avoir recours au marquage nucléaire.
Avantages
- Identification des phénotypes nucléaires sans utiliser de marqueurs fluorescents
- Gain de temps et réduction des coûts liés à l’utilisation de fluorophores nucléaires (marquage et imagerie)
- Réduction de la phototoxicité et du photoblanchiment
- Préservation de l’activité des composés photosensibles
Méthodes
Nous avons créé un ensemble de données d’entraînement rassemblant les trois principaux phénotypes du récepteur des androgènes généralement observés en réponse à des médicaments. Nous avons utilisé la première moitié des images pour générer le réseau neuronal, et l’autre moitié pour évaluer ses performances. Enfin, nous avons utilisé notre réseau neuronal de classification avec un ensemble de données indépendant sans marquage nucléaire.
Préparation des données d’entraînement correspondant aux trois phénotypes de réponse du récepteur des androgènes
Pour constituer un réseau neuronal, nous avons d’abord généré un ensemble de données d’entraînement correspondant aux trois principaux phénotypes de réponse du récepteur des androgènes (AR) :
- AR inactif localisé dans le cytoplasme (contrôle sans traitement, NTC)
- AR localisé dans le noyau lié au ligand, mais inactif (10 µM de bicalutamide)
- Ar actif localisé dans le noyau, sous forme de moucheture (1 nM de R1881)
Pour générer les données de référence factuelles (ground truth), nous avons marqué les noyaux avec du SiR-DNA. Nous avons acquis des images de 1024 x 1024 pixels avec le scanner galvanométrique du microscope confocal à balayage laser Olympus FV3000 couplé à un objectif UPLSAPO 20X. À ce stade, nous avons divisé l’ensemble de données (60 images) en deux parts égales ; la première constituant l’ensemble d’entraînement et la seconde l’ensemble de validation (30 images chacune). Pour entraîner le réseau neuronal, nous avons utilisé 10 images GFP-AR de projection d’intensité maximale pour chacun des trois phénotypes de réponse du récepteur des androgènes (50 % des données, 30 images au total) avec un marqueur nucléaire.
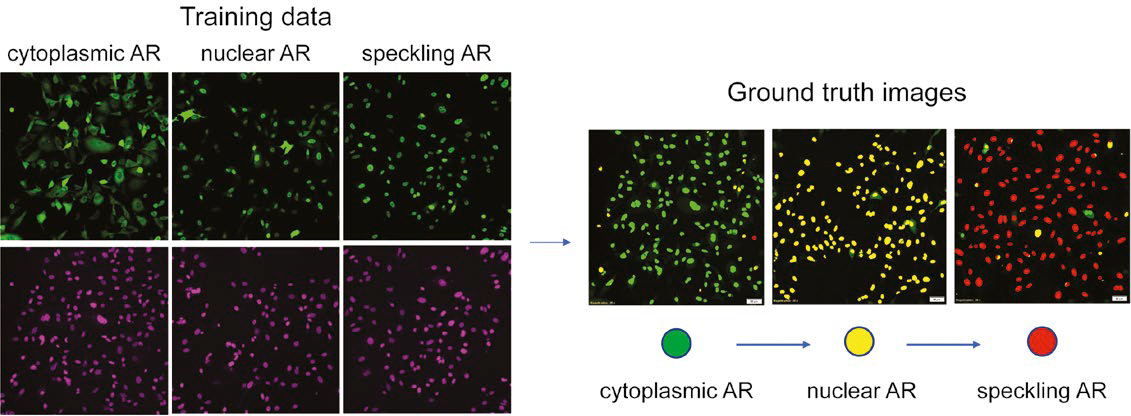
Figure 2 : Préparation des données d’entraînement à la classification des phénotypes de réponse de l’AR en fonction du marquage nucléaire. (Gauche) vert : AR-GFP, violet : SiR-DNA. (Droite) évaluation des ensembles de données de référence factuelles.
Optimisation des données de référence factuelles
La figure 2 représente les données de référence factuelles analysées par le réseau neuronal, avec l’AR localisé dans le cytoplasme en vert, l’AR localisé dans le noyau en jaune et les mouchetures d’AR en rouge. En utilisant cette carte de segmentation avec ce code de couleurs, nous avons affiné les données de référence factuelle en corrigeant manuellement les erreurs de détection et de segmentation des noyaux et en reclassant les noyaux mal classés.
Création du réseau neuronal à l’aide de la technologie d’apprentissage profond TruAI™
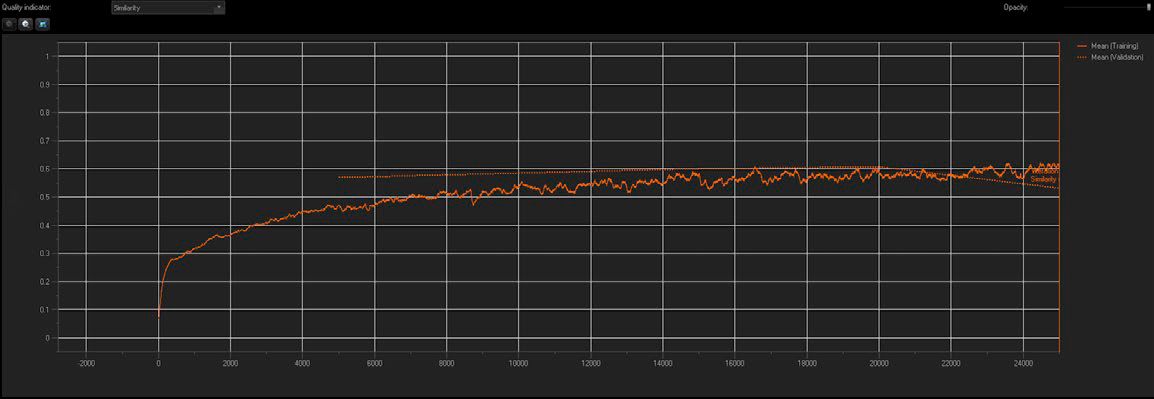
Figure 3 : L’entraînement du réseau neuronal indique l’indice de similitude et les valeurs moyennes de validation en temps réel.
Nous avons utilisé la technologie TruAI pour créer un réseau neuronal standard avec des configurations multiclasses en intégrant 10 images d’entraînement pour chaque phénotype de réponse du récepteur des androgènes. La qualité et la précision du réseau neuronal sont indiquées en temps réel. Comme représenté dans la figure 3, l’indice de similitude a augmenté après chaque itération au cours de la phase d’entraînement jusqu’à atteindre un plateau indiquant une saturation de l’entraînement (flèche vers le bas). Des points de contrôle ont été créés toutes les 5 000 itérations (20 % d’avancement de l’entraînement). Le point de contrôle optimal correspondant à un réseau neuronal entraîné a été enregistré avec son indice de similitude.
Résultats
Validation d’un réseau neuronal de classification à l’aide de données aveugles
Nous avons validé notre réseau neuronal de classification en utilisant l’autre moitié des images non utilisées pour la génération des données de référence factuelle ou l’entraînement du réseau neuronal (données aveugles). Nous avons ensuite utilisé ces résultats de validation pour affiner les paramètres du modèle en appliquant un filtre de surface (49,83 µm min. - 555,44 µm max.) afin d’éliminer les débris cellulaires et les cellules mal segmentées. Le réseau neuronal entraîné a prédit les différentes classes de phénotypes de réponse du récepteur des androgènes sans aucun marquage nucléaire. La figure 4 illustre la répartition des classes de phénotype déterminées à partir des données de 10 images.
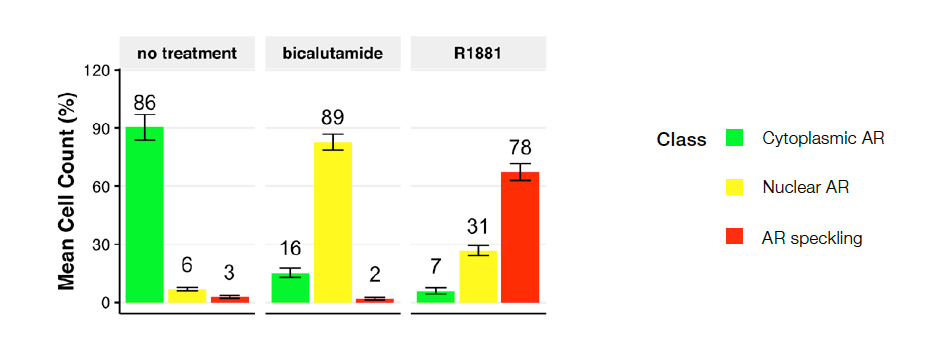
Figure 4 : Répartition des classes de cellules dans les conditions suivantes : aucun traitement (gauche), bicalutamide (milieu) ou R1881 (droite). Le graphique représente le pourcentage de cellules de classe 1 (AR localisé dans le cytoplasme), de classe 2 (AR localisé dans le noyau) et de classe 3 (moucheture d’AR). En l’absence de traitement, la majorité des cellules analysées sont de classe 1. Le traitement par le bicalutamide (antagoniste de l’AR) a induit un déplacement de la répartition des phénotypes vers la classe 2, tandis que le traitement par R1881 (agoniste de l’AR) a induit un déplacement vers la classe 3, à savoir une localisation de l’AR actif au niveau de sites de transcription actifs.
Prédiction des différentes classes de phénotypes de réponse du récepteur des androgènes sur des ensembles de données indépendants
Ensuite, pour évaluer le modèle de réseau neuronal de classification sur des images indépendantes, nous avons préparé un nouvel ensemble de données à l’aide de deux antiandrogènes cliniques : le bicalutamide et l’enzalutamide (figure 5).
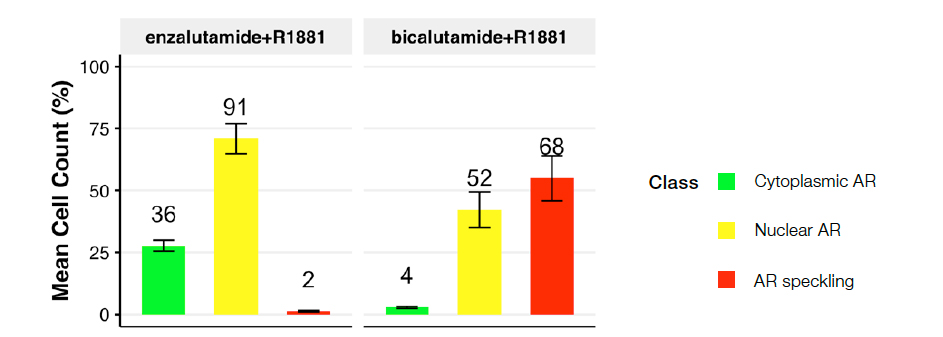
Figure 5 : Répartition des classes de cellules dans les conditions suivantes : R1881 plus enzalutamide (gauche) ou bicalutamide (droite). Le graphique représente la répartition prédite des classes de noyaux en fonction du traitement utilisé.
Le traitement par l’enzalutamide, un antiandrogène de deuxième génération, a conduit à une population de cellules principalement de classe 1 (AR localisé dans le cytoplasme) avec très peu, voire aucune, de cellules de classe 3 (mouchetures d’AR), ce qui suggère que l’activation du récepteur des androgènes induite par le ligand est efficacement bloquée. A contrario, le traitement par le bicalutamide, un antiandrogène d’ancienne génération moins efficace, conduit à un déplacement important de la population vers les cellules de classe 3 (AR actif), ce qui suggère que le bicalutamide est incapable de bloquer efficacement l’activation du récepteur des androgènes induite par 1nM de R1881. Ces données vont dans le sens des précédentes études2.
Conclusion
Grâce au microscope confocal Olympus FV3000 et au logiciel cellSens™ doté du module d’apprentissage profond TruAI™, nous avons généré un réseau neuronal capable de détecter et de segmenter les noyaux non marqués et de classifier les cellules en fonction des phénotypes de réponse du récepteur à des médicaments. Cette approche pourrait permettre d’augmenter l’efficacité des essais de médicaments en réduisant la durée des essais (préparation des expériences) et les coûts.
En outre, les réseaux neuronaux peuvent améliorer la qualité des données en réduisant les artefacts, tels que la phototoxicité, le photoblanchiment et la désactivation des composés photosensibles, et peuvent détecter des variations subtiles que les chercheurs et/ou les méthodes d’analyse des images traditionnelles n’ont pas repérées. Cette approche d’apprentissage profond peut être utilisée plus largement pour subdiviser les populations de cellules en fonction de paramètres définis (au niveau nucléaire ou cellulaire) allant de la localisation de protéines aux changements morphométriques.
Références
- Risek B, Bilski P, Rice AB, Schrader WT. Androgen receptor-mediated apoptosis is regulated by photoactivatable androgen receptor ligands. Mol Endocrinol. 2008 Sep;22(9):2099-115. doi: 10.1210/me.2007-0426. Epub 18 juin 2008. PMID: 18562628; PMCID: PMC2631375.
- Sugawara T, Lejeune P, K hr S, Neuhaus R, Faus H, Gelato KA, Busemann M, Cleve A, Lücking U, von Nussbaum F, Brands M, Mumberg D, Jung K, Stephan C, Haendler B. BAY 1024767 blocks androgen receptor mutants found in castration-resistant prostate cancer patients. Oncotarget. Fév. 2016 2;7(5):6015-28. doi: 10.18632/oncotarget.6864. PMID: 26760770; PMCID: PMC4868737.
Auteurs
- Harish Sura, Katherin Patsch et Seungil Kim du Lawrence J. Ellison Institute for Transformative Medicine, University of Southern California, Los Angeles, CA, États-Unis
- Shohei Imamura, Responsable de projets stratégiques, Olympus Corporation of the Americas
Produits utilisés pour cette application
a bien été ajouté à vos favoris
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
Not Available in Your Country
Sorry, this page is not
available in your country.