Suivi de la dynamique du cycle cellulaire lors de la différenciation de cellules souches à l’aide de la technologie d'apprentissage profond TruAI™ du système scanR
Introduction
Lors des premières phases du développement des mammifères, les cellules souches embryonnaires (CSE) se spécialisent et donnent naissance à toutes les couches de cellules germinales embryonnaires, ce qui génère à terme une multitude de types cellulaires différents dans l'organisme du mammifère adulte. Les cellules souches embryonnaires se caractérisent par leur incroyable capacité à s'autorenouveler et à proliférer et par de courts cycles de division dus à des phases GAP tronquées (G1 et G2). Lors de la différenciation cellulaire, des événements cellulaires spécifiques se produisent : les cellules changent de morphologie, grandissent d'environ dix fois leur taille initiale, modifient leur rapport nucléocytoplasmique et prennent une forme plate et allongée ; les programmes d'expression génique relatifs à la fonction des cellules sont activés et la structure de la chromatine est modifiée. Mais surtout, le cycle cellulaire ralentit. Les cellules somatiques différenciées ont des phases G1 et G2 longues, un contrôle strict des points de contrôle et des cycles de division bien régulés (Padgett and Santos 2020) (figure 1).
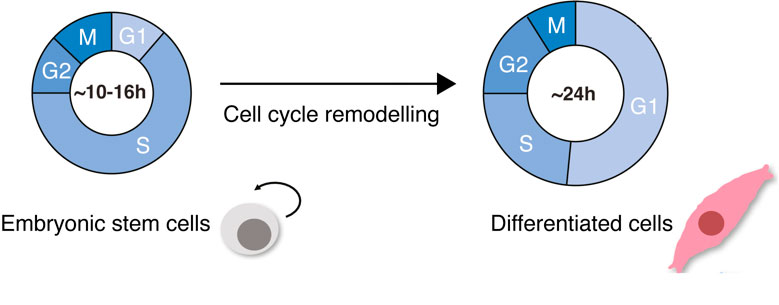
Le remodelage du cycle cellulaire n'est pas un phénomène qui survient uniquement au cours de la différenciation des cellules souches embryonnaires. Des modifications de la dynamique du cycle cellulaire se produisent souvent en biologie ; par exemple, lorsque les cellules se régénèrent après une infection virale ou lors de la transformation en cellules malignes. L'étude de la modification de la dynamique du cycle cellulaire dans le cadre de la différenciation cellulaire est donc importante pour comprendre les mécanismes du contrôle du cycle cellulaire potentiellement conservés et aura un impact sur notre compréhension de l'équilibre entre les états sains et pathologiques.
Les modifications observées lors de la différenciation peuvent être surveillées au niveau de chaque cellule en mesurant l'activité des protéines génétiquement encodées impliquées à différents stades du cycle cellulaire (Araujo et al. 2016 ; Sakaue-Sawano et al. 2008). Miyawaki et ses collègues (Sakaue-Sawano et al. 2017) ont mis au point le système FUCCI(CA) (fluorescent ubiquitination-based cell cycle indicator), un indicateur de cycle cellulaire par fluorescence dépendant de l'ubiquitination, qui permet de distinguer les phases G1, S et G2 grâce aux variations de l’émission de fluorescences de trois couleurs distinctes (rouge, vert et jaune) associées à la dégradation de Cdt1 et de la géminine, deux protéines importantes du cycle cellulaire, observables via les canaux de détection de la YFP et de la mCherry d'un microscope (figure 2).
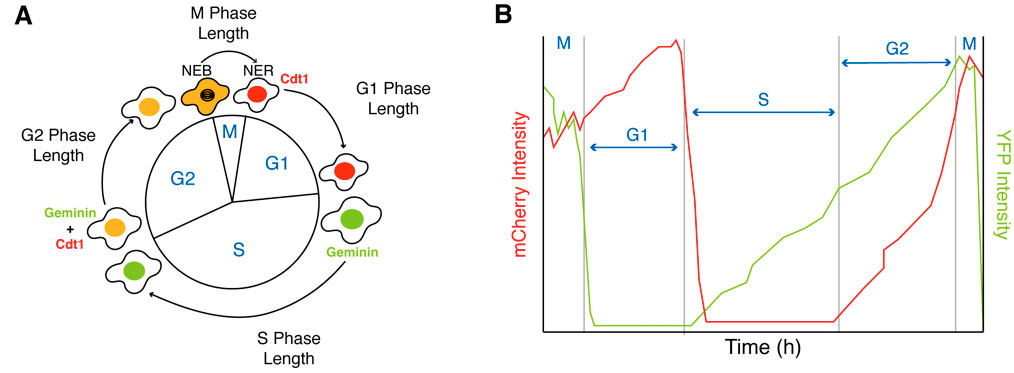
Figure 2. Mesure des phases G1, S, G2 et M du cycle cellulaire à l'aide de système FUCCI(CA). (A) Représentation schématique du système FUCCI(CA). (B) Exemple de tracés de la durée des phases G1, S, G2 et M mesurée en fonction des niveaux d'expression de Cdt1 (canal de la mCherry) et de la géminine (canal de la YFP).
Le suivi de la dynamique du cycle cellulaire pendant la différenciation cellulaire présente à la fois des défis liés à l'imagerie et à l'analyse des données. L'imagerie de cellules vivantes sur le long terme requiert une température stable et le contrôle du taux de CO2, un éclairage de faible intensité pour réduire au maximum les risques de phototoxicité et des routines de mise au point automatique solides afin d'acquérir des images de bonne qualité tout au long du processus de prise de vues à intervalles.
En matière d'analyse, le plus gros défi est la segmentation puis le suivi des cellules individuelles sous de faibles intensités de fluorescence et dans des conditions de forte confluence tandis que les cellules souches embryonnaires se déplacent, se divisent et changent radicalement de forme au cours de leur différenciation.
Tous ces défis peuvent être surmontés à l’aide du microscope scanR à épifluorescence et haut contenu équipé d'un système d'incubation cellVivo, d'un système de compensation de la dérive en Z pour la mise au point automatique, de la technologie d'apprentissage profond TruAI™ pour la segmentation et d'un module cinétique pour le suivi.
Objectifs
Dans cette note d'application, nous montrons que l'utilisation de la technologie TruAI permet d'améliorer de manière significative la détection et le suivi d'une cellule souche embryonnaire par imagerie sur le long terme en association avec le système FUCCI(CA). Cette technologie est particulièrement puissante pour les cellules sans marqueur nucléaire permettant d’assurer leur suivi et permet d’éviter les problèmes de phototoxicité en réduisant l’éclairage.
La technologie TruAI est utilisée pour générer un réseau neuronal profond capable d'identifier de manière fiable la position des cellules souches embryonnaires pluripotentes et des cellules différenciées à tous les temps de l’imagerie, même lorsque l'intensité de la fluorescence est très faible. Cela permet un excellent suivi des phases G1, S et G2 des populations de cellules dans le temps. Ensuite, l’utilisation du module cinétique scanR, nous permet de suivre des milliers de cellules, d’évaluer leurs déplacements sur de longues périodes et de recueillir des informations dynamiques quantitatives sur les du apportées au cycle cellulaire à l'échelle d'une seule cellule.
Illustration de la polyvalence du système scanR en tant qu'outil de recherche, toutes les données présentées dans cette note d’application, y compris les graphiques, les diagrammes de dispersion, les galeries d'objets, les suivis et les tracés cinétiques, sont générées uniquement avec le logiciel scanR.
Dispositif expérimental
Des cellules souches embryonnaires humaines H1 (hESC) exprimant le système FUCCI(CA) sont traitées avec 50 ng/ml de protéine osseuse morphogénique 4 (BPM4) pour induire leur différenciation en cellules de la lignée mésendodermique. Des images de ces cellules H1 sont ensuite prises toutes les 15 minutes pendant 6 jours via les canaux de détection de la mCherry et de la YFP d'un microscope scanR équipé d'un objectif 10X UPLANSAPO.
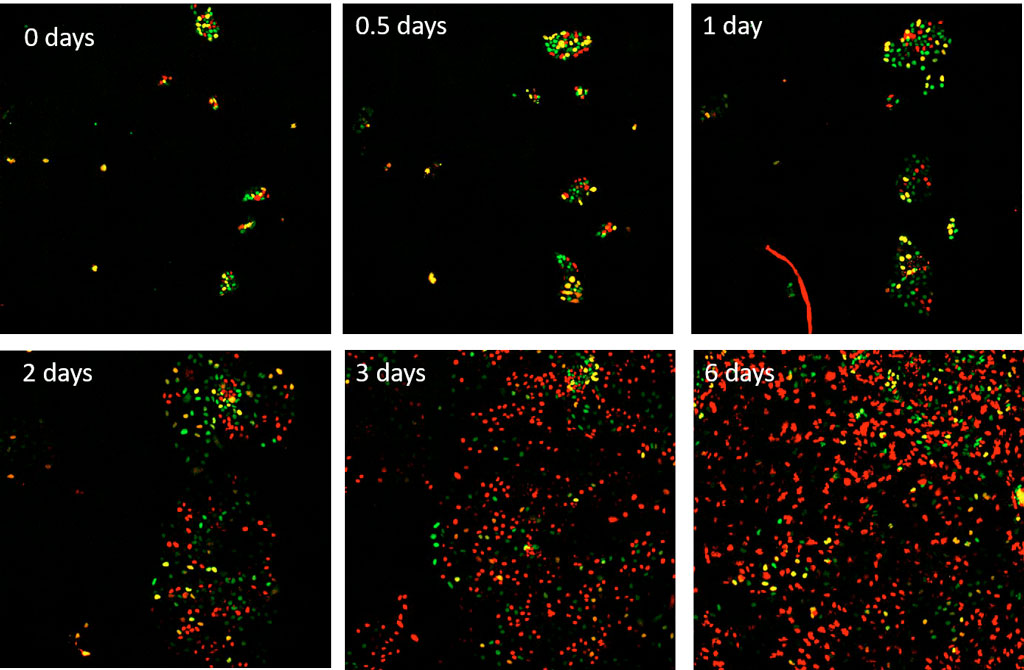
Figure 3. Série d’images chronologique sur 6 jours de cellules hESC H1 exprimant le système FUCCI(CA) stimulées avec de la BMP4 au début de la session d'imagerie. Les points rouges, verts et jaunes correspondent respectivement aux phases G1, S et G2.
Développement d'un réseau neuronal profond pour détecter les cellules sur des images de cellules vivantes
Une cellule exprimant le système FUCCI(CA) peut présenter de fortes variations de l'intensité de la fluorescence lorsqu’elle est observée pendant plusieurs jours. L'intensité totale peut notamment être très faible lors du passage de la phase G1 à la phase S (figure 2b). Cela peut empêcher les algorithmes de suivi de fonctionner correctement à cause de l’impossibilité de collecter des données aux temps où les cellules sont difficiles à détecter.
Pour assurer le suivi sur plusieurs jours du plus grand nombre de cellules possible, il faut développer un réseau neuronal profond capable de détecter des cellules à très faibles intensités. Dans un précédent article technique, nous avons montré que cela est possible en utilisant des paires d'images de cellules fluorescentes, l’une acquise en exposition longue et sous éclairage intense et l’autre en exposition courte et sous faible éclairage (Woerdemann 2020). En résumé, les images acquises en exposition longue sont utilisées pour la segmentation des cellules et un masque est généré. Ce masque est utilisé comme données de terrain pour entraîner le réseau neuronal profond à détecter les cellules sur les images prises avec de faibles niveaux d'exposition.
Il est également possible d’entraîner le réseau neuronal en réalisant des annotations manuelles sur l'ensemble de données à analyser. Pour cette tâche, il est possible d'utiliser le logiciel cellSens™ d'Olympus. Les modèles de réseaux neuronaux profonds développés dans le logiciel cellSens peuvent être importés dans le logiciel scanR. Pour cette note d'application, nous avons utilisé cette dernière méthode manuelle et avons fourni des centaines d'annotations de cellules de différentes intensités à différents temps pour entraîner le modèle de réseau neuronal profond.
Segmentation : comparaison de la technologie d'apprentissage profond TruAI aux méthodes classiques
Une fois entraîné, le réseau neuronal profond est appliqué à chaque canal de détection de la fluorescence mCherry et YFP. Cela crée une carte de probabilité calculée par IA par pixel pour chaque canal. Plus la probabilité IA est élevée dans un pixel, plus la confiance que le pixel est associé à une cellule est grande. Les cartes de probabilité IA des deux canaux sont ensuite additionnées, et la segmentation est réalisée en appliquant un seuil d'intensité à cette somme (figures 4e et 4f).
Pour comparer les résultats avec ceux des méthodes classiques, les canaux de détection de la fluorescence mCherry et YFP sont corrigés pour éliminer le bruit de fond à l'aide d'un algorithme rolling ball, puis additionnés. La somme est alors segmentée à l’aide d’un seuil d'intensité (figure 4c) ou d’une méthode de détection des contours (figure 4d). Toutes les étapes de traitement des images décrites ci-dessus sont réalisées avec le logiciel d'analyse scanR.
Les outils statistiques du système scanR sont utilisés pour créer le tableau 1 dans lequel les résultats correspondent à un seul champ de vision sur une période de six jours.
Tableau 1. Comparaison du nombre de cellules segmentées par les différentes méthodes
| Temps | Méthodes de segmentation | ||
|---|---|---|---|
| Seuil d'intensité | Détecteur des contours | Technologie TruAI | |
| 0 h | 78 | 103 | 93 |
| 24 h | 209 | 188 | 240 |
| 48 h | 421 | 281 | 475 |
| 72 h | 847 | 654 | 986 |
| 96 h | 1480 | 1330 | 1590 |
| 160 h | 1340 | 1460 | 1610 |
|
Somme de toutes les images
0 - 160 h | 504131 | 573179 | 659199 |
Une simple comparaison entre les images de fluorescence et l'image de probabilité IA (figure 4a et 4b) révèle que l'approche TruAI a une sensibilité supérieure et détecte tous les types de cellule. Cela est confirmé par les résultats rassemblés dans le tableau 1 ainsi que par les images sur toute la période. La méthode classique du seuil d'intensité ne permet pas de détecter les cellules faiblement fluorescentes et de déterminer leur forme exacte (figure 4c). La méthode de détection des contours améliore les contours des cellules détectées, mais ne parvient pas à détecter de nombreuses cellules faiblement fluorescentes (figure 4d). La technologie d'apprentissage profond TruAI permet de détecter de manière fiable les cellules faiblement fluorescentes et leurs contours sont bien définis (figures 4e et 4f).
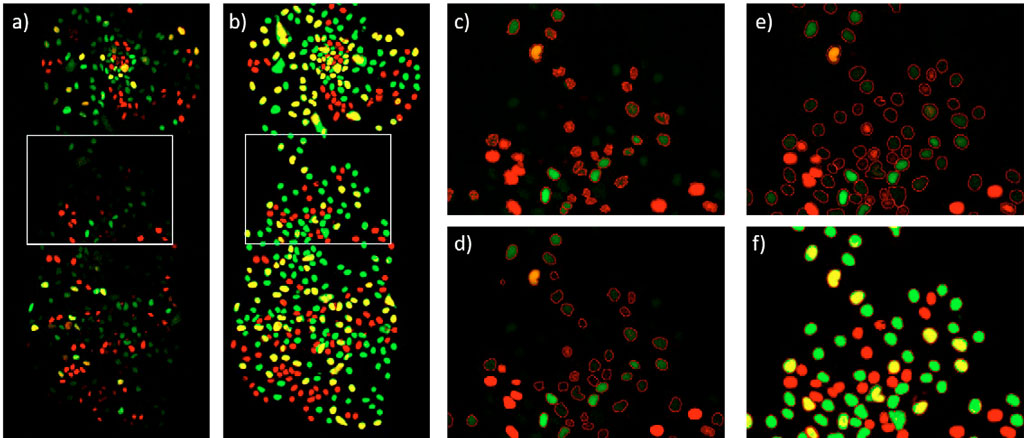
Figure 4 : Colonie de cellules ESC à 48 h. a) Canaux de détection de la fluorescence mCherry (rouge, phase G1) et YFP (vert, phase S). Le jaune, la combinaison de la mCherry et de la YFP, correspond à la phase G2. b) Probabilité TruAI dans le canal mCherry (rouge) et dans le canal YFP (vert). Les cellules présentant une forte probabilité IA dans les deux canaux sont affichées en jaune. c) Segmentation à l’aide d’un seuil d'intensité dans la somme des canaux de détection de la fluorescence mCherry et YFP d) Segmentation à l’aide d’un détecteur de contours dans la somme des canaux de détection de la fluorescence mCherry et YFP e) Segmentation dans la somme des intensités de probabilité TruAI, recouverte par l'image de fluorescence f) Identique à l'image e) mais recouverte par la probabilité TruAI.
Analyse des populations cellulaires dans le temps
Les intensités de la fluorescence de la mCherry et de la YFP des cellules segmentées par la technologie TruAI sont représentées sous forme de diagramme de dispersion à différents intervalles de temps (figure 5). Les cellules dont l'intensité ne reflète que celle de la mCherry sont des cellules en phase G1, les cellules dont l'intensité ne reflète que celle de la YFP sont des cellules en phase S et les cellules dont les intensités reflètent les deux canaux correspondent aux cellules en phase G2. La figure 5 montre que la majorité des cellules sont au début en phase S et que plus le temps avance, plus le nombre de cellules en phase G1 augmente. Ce déplacement de population correspond respectivement aux profils du cycle cellulaire attendus : cellules pluripotentes et différenciées (figure 1).
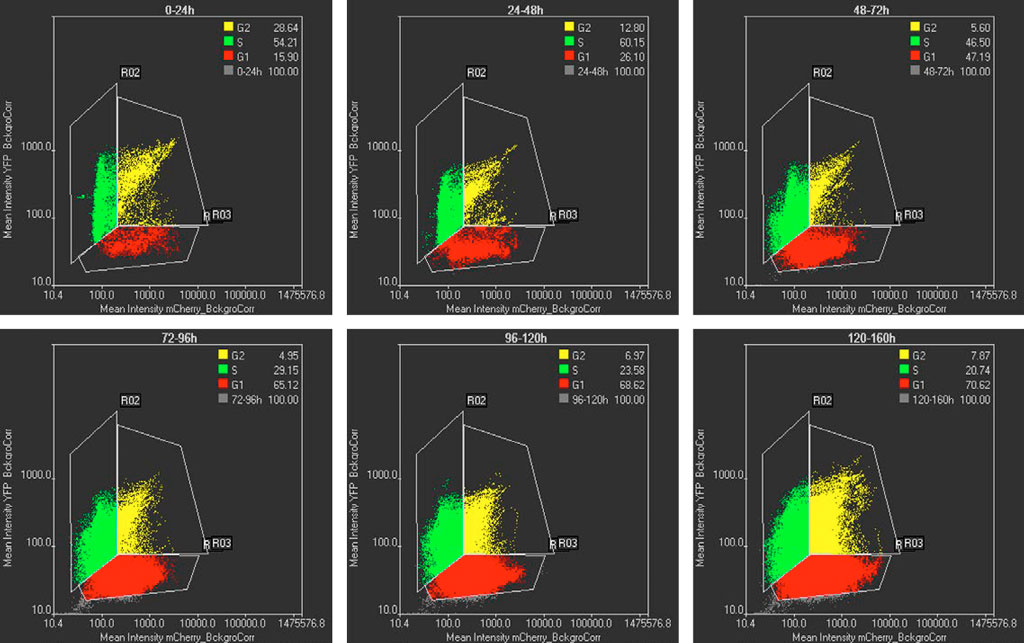
Figure 5 : Diagrammes de dispersion des intensités de fluorescence mCherry et YFP à différents intervalles de temps. Chaque point sur le diagramme de dispersion représente une cellule segmentée par la technologie TruAI. Les cellules en phase G1, S et G2 sont respectivement représentées en rouge, vert et jaune. Les pourcentages des populations G1, S et G2 sont indiqués dans la légende. Les intervalles de temps sont indiqués en haut de chaque graphique.
Suivi des cellules : comparaison de la technologie TruAI aux méthodes classiques
Les cellules segmentées observées avec les trois méthodes (seuil d'intensité, détecteur des contours et technologie TruAI) ont été suivies à l’aide des mêmes paramètres de suivi du module de cinétique de scanR. Les outils statistiques du système scanR sont utilisés pour créer le tableau 2 dans lequel les résultats correspondant à un seul champ de vision sur une période de six jours.
Tableau 2. Comparaison du nombre de cellules suivies par les différentes méthodes
| Temps | Nombre de cellules suivies | ||
|---|---|---|---|
| Seuil d'intensité | Détecteur des contours | Technologie TruAI | |
| 24 h ou plus | 302 | 1030 | 1965 |
| 48 h ou plus | 16 | 126 | 426 |
| 72 h ou plus | 0 | 27 | 72 |
| 96 h ou plus | 0 | 0 | 5 |
Le tableau montre que l'utilisation de la technologie TruAI pour la segmentation améliore clairement les résultats de suivi qui suit. Pour cet ensemble de données, étant donné un nombre moyen de 200 cellules par champ de vision à 24 heures (tableau 1), plus de 70 cellules peuvent être suivies sur 3 jours. Il est même possible de suivre les cellules pendant 4 jours ou plus. La capacité à suivre ce nombre élevé de cellules sur de si longues périodes est importante, car cela permet l'étude de la dynamique du cycle cellulaire sur de nombreuses divisions cellulaires d’une manière statistiquement significative.
Analyse des suivis des cellules
Pour chaque suivi généré par le système scanR, de nombreux paramètres peuvent être extraits.
- Pour identifier les suivis les plus longs, il est possible de représenter le paramètre « lifetime » (durée du suivi) sur un histogramme et de créer une fenêtre pour sélectionner les suivis de plus de 24 heures (figure 6a).
- Pour filtrer un peu plus les suivis, les paramètres « First (time) » (Premier [temps]) et « Last (time) » (Dernier [temps]) peuvent être représentés sur un diagramme de dispersion (figure 6b). Cela peut nous aider à identifier les cellules suivies uniquement pendant les premiers jours (cellules souches embryonnaires avec durées de cycle courtes), les cellules suivies uniquement pendant les derniers jours (cellules différenciées avec durées de cycle longues) et les cellules suivies du début jusqu'à la fin de l’imagerie à intervalle (cellules pour lesquelles le processus de différenciation est suivi). Une fois ces suivis identifiés, les intensités de la mCherry et de la YFP peuvent être représentées en fonction du temps afin d'obtenir des informations quantitatives sur la dynamique du cycle cellulaire (figures 6 c, d, e).
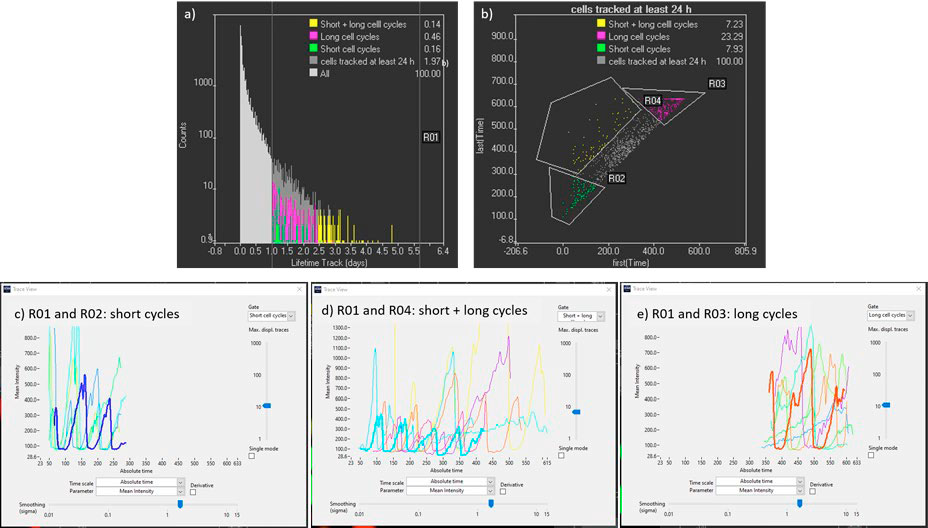
Figure 6. a) Histogramme des durées des suivis utilisé pour filtrer les suivis longs (R01). b) Diagramme de dispersion avec le temps initial et le temps final des suivis pour identifier les cellules suivies du début à la fin de l'imagerie par intervalle (R01 et R04). c), d) et e) Tracés cinétiques des cellules mCherry suivies pendant plus de 24 h avec des temps de début et de fin de cycle précoces (c), un temps de début précoce et un temps de fin tardif (d) et des temps de début et de fin tardifs. Les temps sont espacés de 15 minutes.
Sur les figures 6c à 6e, on observe que la durée du signal de mCherry (phase G1) augmente avec le temps puisqu'elle passe de 2 heures environ pour les cellules souches embryonnaires à 24 h une fois les cellules différenciées. Pour approfondir l'analyse de la dynamique du cycle cellulaire, il est possible de sélectionner les suivis des régions R01 et R04 dans lesquelles la différenciation des cellules souches embryonnaires est suivie sur un minimum de quatre divisions cellulaires (figure 6d).
La figure 7 représente le suivi d'une cellule souche embryonnaire en cours de différenciation au cours de cinq cycles cellulaires. Sur l'illustration 7a, la position de la cellule souche embryonnaire au sein de sa colonie est indiquée du temps 52 (13 h) au temps 422 (105 h). La figure 7b est l’image prise au temps 280 (70 h). Notez que l'intensité de cette cellule est extrêmement faible et que la cellule n'a pas été détectée par les méthodes classiques. La figure 7c est l'image de probabilité TruAI au même temps que l’image 7b. L'intensité de probabilité de la cellule suivie est aussi forte quel celle du reste des cellules. Les figures 7d et 7e représentent une galerie des cellules suivies toutes les 15 minutes pour, respectivement, la fluorescence et la probabilité TruAI. Notez que lorsque la phase G1 se termine (rouge), l'intensité totale est toujours très faible. La figure 7f représente les oscillations de l'intensité de la fluorescence de la mCherry (graphique du dessus) et de la YFP (graphique du dessous) à partir desquelles les phases G1, S, G2 et M du cycle cellulaire peuvent être extraites.
Ces données nous indiquent que cette cellule passe plus de temps en phase G1 et moins de temps en phase S et G2 à mesure que le temps avance. La phase M est toujours très courte ; sa position exacte coïncide avec une baisse du signal de la mCherry et une chute brutale du signal de la YFP.
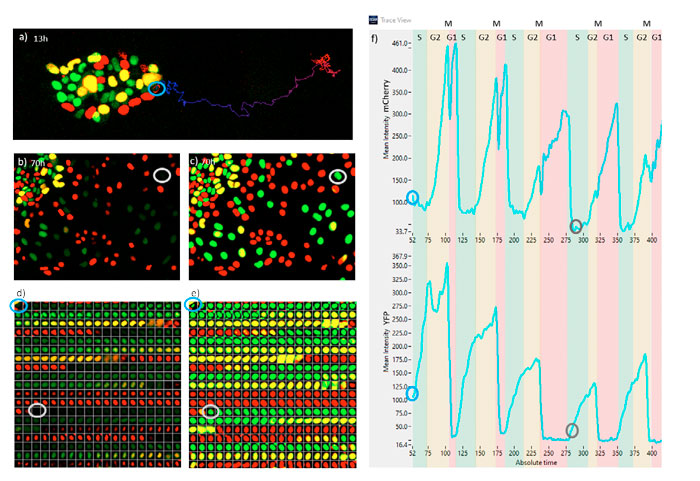
Figure 7. a) Image de la fluorescence à 13 h. b) Image de la fluorescence à 70 h. c) Image TruAI à 70 h. d) Séquence d’images de la fluorescence de la cellule suivie prises à intervalle de 15 minutes. e) Séquence d’images TruAI de la cellule suivie prises à intervalle de 15 minutes. f) Tracés de la fluorescence de la mCherry et de la YFP de la cellule suivie indiquant les différentes phases du cycle cellulaire. Les intervalles de temps sont de 15 minutes. Les cercles bleus et gris sur les figures a à f indiquent la même cellule à, respectivement, 13 ou 70 h.
Conclusion
Cette nouvelle approche d'apprentissage profond pour l'analyse de la dynamique du cycle cellulaire lors de la différenciation des cellules souches embryonnaires associée au module cinétique du système de criblage à haut contenu scanR permet d'acquérir des données quantitatives reproductibles et statistiquement significatives sur la manière dont la dynamique du cycle cellulaire évolue au niveau de cellules individuelles lors de leur différenciation. Des milliers de cellules dont la forme et la morphologie sont en train de changer peuvent être reconnues et suivies pendant plusieurs jours au cours de leur différenciation en mesurant leur fluorescence. Il s'agit d'un outil puissant permettant d’étudier la dynamique du cycle cellulaire au cours du développement précoce et de la reprogrammation ainsi que celle de la transformation maligne des cellules dans laquelle la régulation du cycle cellulaire joue un rôle essentiel.
Références
Padgett, J., et Santos, S.D.M. 2020. From clocks to dominoes: lessons on cell cycle remodeling from embryonic stem cells. FEBS Letters. 10.1002/1873-3468.13862.
Araujo, A.R., Gelens, L., Sheriff, R.S.M., et Santos, S.D.M. 2016. Positive feedback keeps duration of mitosis temporally insulated from upstream cell cycle events. Molecular Cell 64, 362–375.
Sakaue-Sawano et al. 2008. Visualizing Spatiotemporal Dynamics of Multicellular Cell-Cycle Progression. Cell 132, 487–498.
Sakaue-Sawano et al. 2017. Genetically Encoded Tools for Optical Dissection of the Mammalian Cell Cycle. Molecular Cell 68, 626–640.
Woerdemann, M., et Genenger, M. 2020. TruAI™ Technology with Deep Learning for Quantitative Analysis of Fluorescent Cells with Ultra-Low Light Exposure. Note d'application Olympus. www.olympus-lifescience.com/en/resources/white-papers/ultra-low_light_exposure_analysis/
Auteurs
Joe Pagdett et Silvia Santos
Quantitative Stem Cell Biology Lab, The Francis Crick Institute, 1 Midland Road, NW1 1AT, Londres, Royaume-Uni
Manoel Veiga Gutierrez
Olympus Soft Imaging Solutions GmbH, Johann-Krane-Weg 39, 48149 Muenster, Allemagne
Produits utilisés pour cette application
a bien été ajouté à vos favoris
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
Not Available in Your Country
Sorry, this page is not
available in your country.