Numérisation d’échantillons fluorescents très épais à l’aide du scanner de lames pour la recherche SLIDEVIEW VS200

Introduction
Les chercheurs de l’Institut de biologie cellulaire moléculaire et de génétique Max Planck situé à Dresde, en Allemagne, étudient les capacités de régénération de la planaire d’eau douce Schmidtea mediterranea à l’aide du scanner de lames pour la recherche SLIDEVIEW VS200 d’Olympus. La planaire S. mediterranea est un modèle fréquemment utilisé pour l’étude de la biologie de la régénération des systèmes.
Son utilisation présente les avantages suivants :
- Le faible coût de leur culture et de leur maintien en nombre suffisant en laboratoire
- Un génome récemment séquencé
- Les outils et les méthodes établis pour les études de perte de fonction génétique par interférence par ARN
Comprendre les capacités de régénération des planaires Schmidtea mediterranea
Les chercheurs de l’Institut exploitent les qualités exceptionnelles des planaires Schmidtea mediterranea. Même lorsqu’elle est coupée en petits morceaux, chaque section d’une planaire peut se régénérer pour reformer une planaire miniature complète et parfaitement proportionnée. Les cellules souches adultes jouent un rôle important dans ce phénomène, une seule d’entre elles pouvant permettre la régénération d’un ver entier.
De nombreux chercheurs travaillent aujourd’hui à mieux comprendre le processus de régénération de S. mediterranea. Une étape importante vers cet objectif a été la réalisation du premier assemblage génomique hautement contigu de S. mediterranea, lequel a été publié par les chercheurs de l’Institut Max Planck en collaboration avec ceux de l’Institut d’études théoriques de Heidelberg dans un article de la revue scientifique Nature*.
L’assemblage en question révèle un génome contenant de nouveaux éléments répétitifs géants et de nouveaux gènes spécifiques aux vers plats. Cependant, d’autres gènes que l’on pensait absolument essentiels pour le maintien en vie d’un animal sont manquants. Cette découverte a des implications potentielles dans les domaines de la recherche sur la régénération, de la biologie des cellules souches et de la bio-informatique.
Images de fluorescence à haute résolution de planaires Schmidtea mediterranea
Les chercheurs de l’Institut Max Planck exploitent les puissantes capacités des systèmes de numérisation de lames dans le cadre de leurs études. Ils utilisent le scanner de lames VS200 pour acquérir des images de fluorescence à haute résolution.
« En plus de nous permettre d’acquérir facilement des images à haute résolution, le scanner VS200 d’Olympus permet la correction en temps réel des images floues grâce à sa fonction TruSight Live », a expliqué le Dr Tobias Boothe de l’Institut Max Planck.
Les spécimens utilisés ne sont pas coupés avant d’être immobilisés sur une lame et sont donc assez épais (200 à 300 μm). Avec une telle épaisseur, les fonctions TruSight Live ou d’empilement selon l’axe Z du système sont essentielles.
La fonction TruSight Live permet aux chercheurs de réduire la lumière diffusée parasite des zones situées au-dessus et en dessous du plan focal. Les données de l’image sont ensuite recalculées à l’aide d’un algorithme spécial de déconvolution 2D. Il en résulte des images d’une netteté et d’une clarté améliorées.
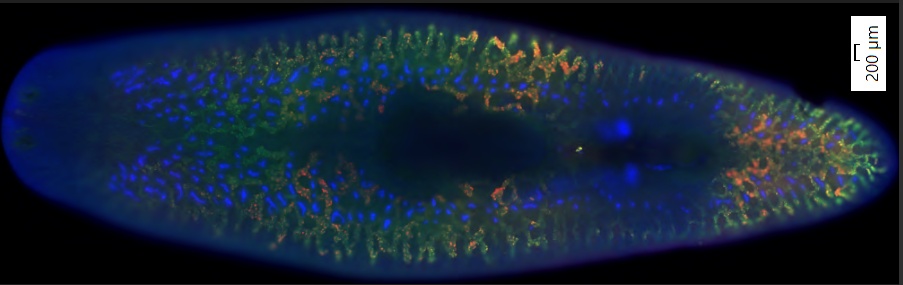
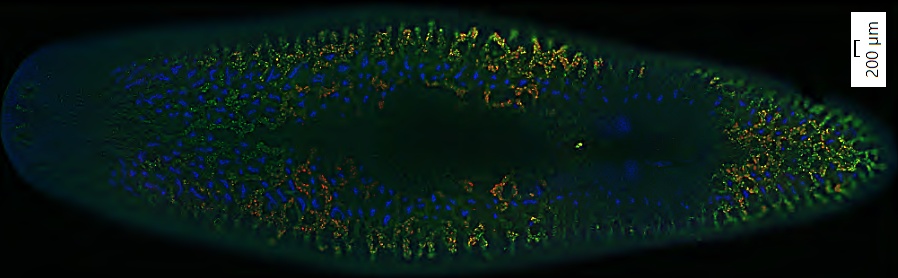
Dans le cadre de cette recherche, les spécimens de S. mediterranea ont d’abord été immobilisés sur une lame, ils ont ensuite été marqués par double hybridation in situ fluorescente (rouge et vert) spécifique de deux marqueurs des glandes vitellines des individus sexuellement matures et, pour finir, ils ont été numérisés à un grossissement de 10× au moyen d’un système VS200. La contre-coloration nucléaire au DAPI a également été acquise. Les deux marqueurs étaient la ferritine et la protéine B du surfactant, qui sont tous spécifiques du vitellus chez les planaires.
L’image de gauche ci-dessous montre une numérisation où aucun défloutage en ligne n’a été appliqué. Dans l’image de droite, la fonction TruSight Live a été appliquée aux trois canaux (DAPI, FITC et CY3) pendant la numérisation.
« Les images et les données que nous acquérons actuellement sont une source d’information essentielle pour notre recherche en cours, laquelle vise à comprendre la relation entre l’abondance du tissu reproducteur chez les planaires adultes et leur capacité de régénération. Pour y arriver, j’ai besoin de plusieurs marqueurs, comme les gènes de la protéine B du surfactant et de la ferritine, tel qu’illustré dans l’image », a déclaré le Dr Miquel Vila-Farré de l’Institut Max Planck.
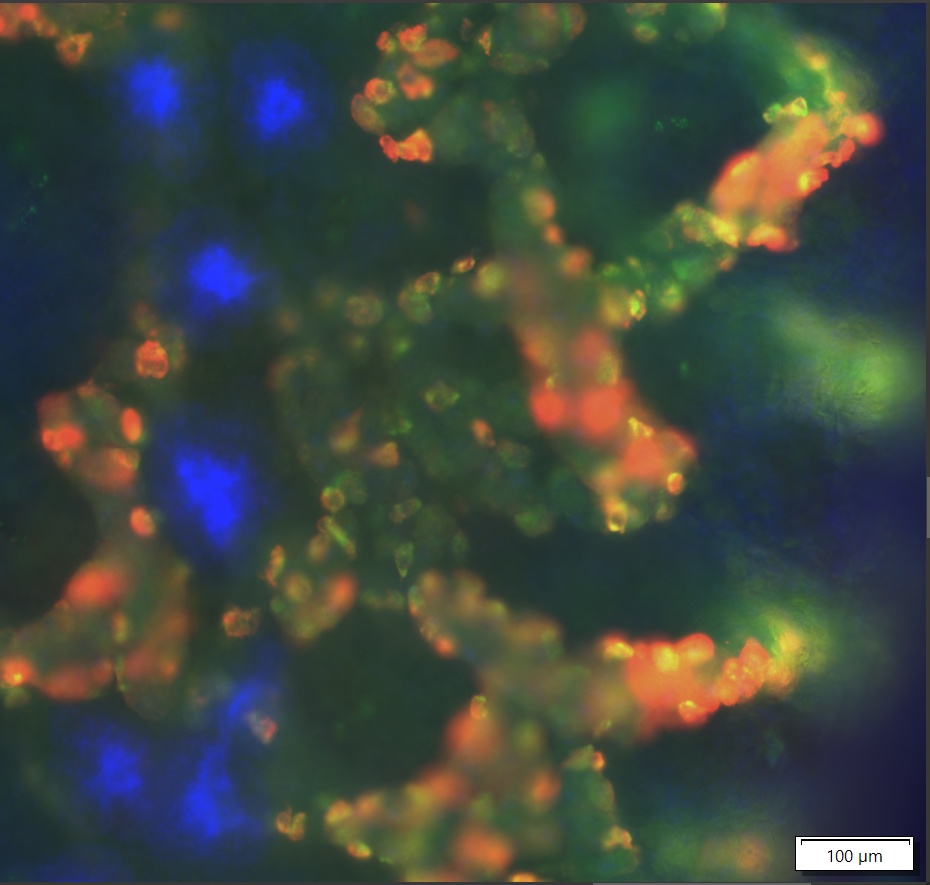
Une autre solution que la fonction TruSight Live est l’acquisition d’un empilement selon l’axe Z virtuel contenant un maximum de 31 plans, comme illustré par les images ci-dessous.
Une fois l’image acquise, il est facile de faire la mise au point au travers de l’empilement selon l’axe Z pour voir la distribution des signaux dans toute l’épaisseur de l’échantillon.
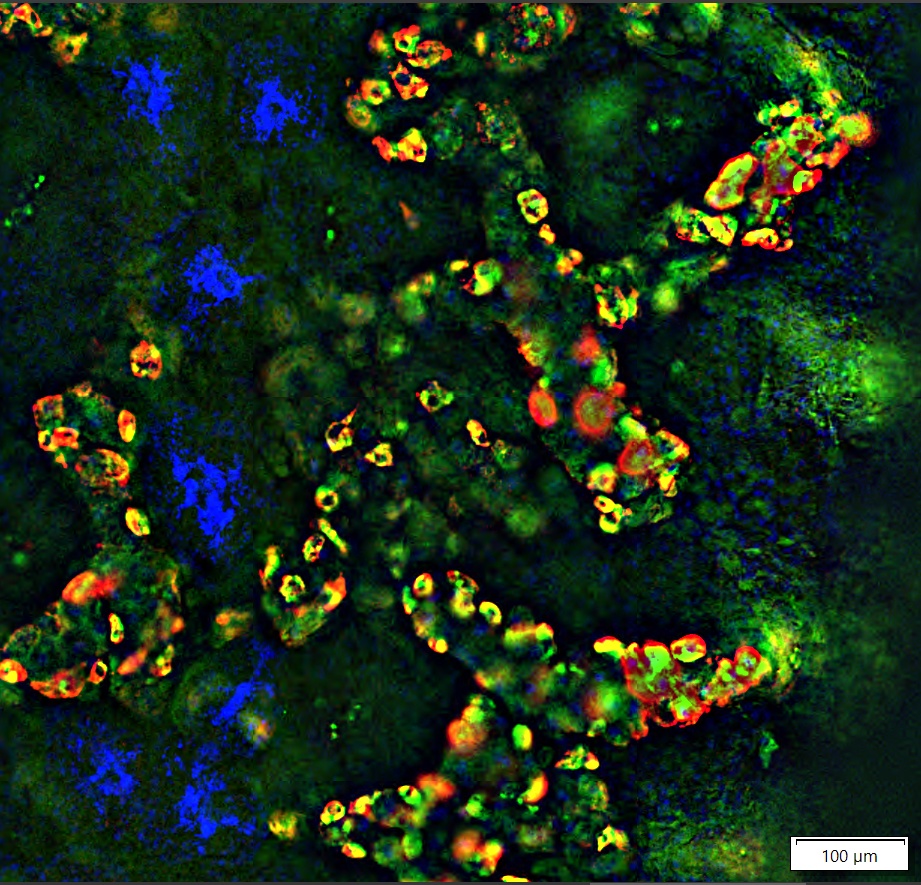
Dans l’image ci-contre, vous pouvez voir une double hybridation in situ fluorescente ciblant deux marqueurs du système excréteur dans la planaire S. mediterranea, Smed-cubilin-1 (FITC, vert) et un nouveau gène (dd_2920, CY3 rouge). Le DAPI (bleu) a été utilisé pour le marquage nucléaire. « Pour comparer l’organisation structurelle du système excréteur de la planaire à celle du néphron des vertébrés, nous avons utilisé Smed-cubilin-1 et dd_2920 comme marqueurs pour visualiser deux types cellulaires différents du système excréteur de la planaire, à savoir, respectivement, les cellules tubulaires proximales et les cellules filtrantes », nous a expliqué le Dr Hanh Vu de l’Institut Max Planck. | 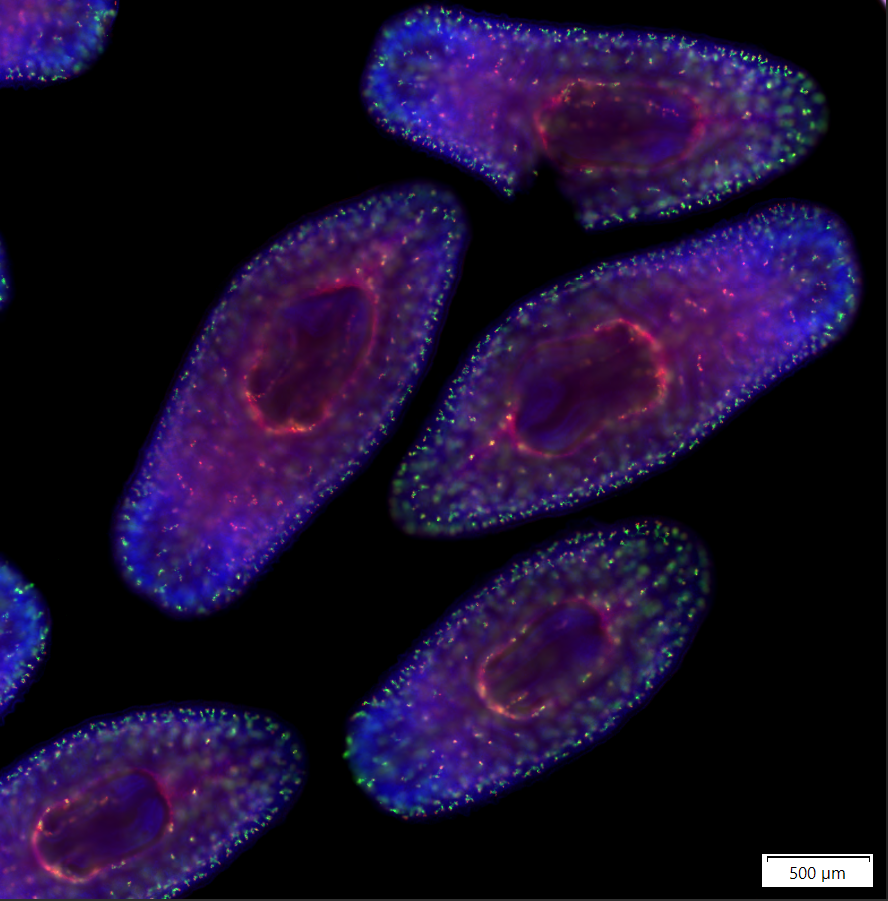 Double hybridation in situ fluorescente (rouge et vert) ciblant deux marqueurs du système excréteur dans la planaire Schmidtea mediterranea. |
Conclusion
Le scanner de lames pour la recherche VS200 d’Olympus capture des images de fluorescence à haute résolution d’échantillons très épais, comme la planaire Schmidtea mediterranea. Les fonctions TruSight Live et d’empilement selon l’axe Z du système sont des capacités essentielles qui aident les chercheurs à observer et à analyser les plus fins détails de ces échantillons épais.
Remerciements
Cette note d’application a été préparée en collaboration avec des chercheurs de l’Institut de biologie cellulaire moléculaire et de génétique Max Planck situé à Dresde, en Allemagne et le Dr Daniel Göttel, spécialiste des applications, Olympus Soft Imaging Solutions, Mü̈nster, Allemagne.
- Dr Tobias Boothe, chercheur postdoctorant à l’Institut de biologie cellulaire moléculaire et de génétique Max Planck, Dresde, Saxe, Allemagne.
- D. Dr Miquel Vila-Farré, chercheur postdoctorant à l’Institut Max Planck de biologie cellulaire moléculaire et de génétique, Dresde, Saxe, Allemagne.
- Dr Hanh Vu, chercheur postdoctorant à l’Institut Max Planck de biologie cellulaire moléculaire et de génétique, Dresde, Saxe, Allemagne.
- Dr Daniel Göttel, spécialiste des applications, Olympus Soft Imaging Solutions GmbH, Mü̈nster, Allemagne
Références
*Nature ; 24 January, 2018 (DOI: 10.1038/nature25473).
Produits utilisés pour cette application
a bien été ajouté à vos favoris
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
Not Available in Your Country
Sorry, this page is not
available in your country.