Observation 3D de foie de souris clarifié à l’aide du microscope FLUOVIEW FV3000
Observation tridimensionnelle du foie en haute résolution
L’observation 3D d’échantillons de tissus épais nécessite généralement l’utilisation d’un microscope à excitation à deux photons en raison de l’augmentation de l’absorption et de la diffusion de la lumière qui se produit lors de l’imagerie en profondeur dans l’échantillon de tissu. Bien que le foie soit généralement un tissu à forte diffusion, l’utilisation de méthodes de clarification associées à des composants optiques appropriés peut permettre l’observation 3D des tissus épais au moyen du microscope confocal FV3000. Dans cette expérience, l’objectif à immersion dans l’huile de silicone x30 d’Olympus avec une ouverture numérique (ON) de 1,05 et une distance de travail (DT) de 0,8 mm a permis une observation 3D en haute résolution de la structure de l’arbre biliaire d’échantillons de foie de souris clarifiés.
Résumé de l’expérience
Clarification optique d’échantillons de tissus de foie de souris
Tissu de foie prélevé sur une souris |
|
Le tissu est coloré pour la fluorescence par immunocoloration |
|
Le tissu hépatique est clarifié à l’aide de SeeDB |
D’après : K. Kamimoto, K. Kaneko, CY. Kok, H. Okada, A. Miyajima et T. Itoh, « Heterogeneity and stochastic growth regulation of biliary epithelial cells dictate dynamic epithelial tissue remodeling », Elife, 19 juillet 2016 ;5, pii : e15034, doi : 10.7554/eLife.15034.
Protocole expérimental de visualisation d’un réseau biliaire complexe en 3D. Après le prélèvement, les tissus biliaires sont soumis à une immunocoloration afin de visualiser un réseau biliaire complexe en trois dimensions. Les échantillons immunocolorés sont ensuite clarifiés à l’aide de SeeDB1).
1) Référence : Ke MT, S. Fujimoto et T. Imai, « SeeDB: a simple and morphology-preserving optical clearing agent for neuronal circuit reconstruction », Nat Neurosci, 16 août 2013 ; (8) : 1154–61, doi : 10.1038/nn.3447, Epub 23 juin 2013, PMID : 23792946
Observation tridimensionnelle des structures de l’arbre biliaire d’un foie de souris présentant des lésions avec un objectif x20 (UPLSAPO20X [ON : 0,75, DT : 0,6 mm])
![Observation tridimensionnelle des structures de l’arbre biliaire d’un foie de souris présentant des lésions avec un objectif x20 (UPLSAPO20X [ON : 0,75, DT : 0,6 mm])](https://static2.olympus-lifescience.com/data/Image/Application/3d-biliary-tree-structure/appnote_3d-biliary-tree-structure_02.jpg?rev=998C) | Le but de l’expérience était de mesurer la longueur et l’épaisseur des branches des structures de l’arbre biliaire de foies de souris présentant des lésions. Pour la sélection initiale des échantillons, nous avons utilisé un objectif à sec x20 sur le microscope FV3000 afin d’obtenir des images tomographiques consécutives, sur un large champ de vision, du tissu biliaire (vert, marqueur de cellule épithéliale biliaire CK19) sur des échantillons de tissu hépatique clarifiés d’une épaisseur de 200 µm. Cette configuration a permis une observation quantitative efficace d’un grand nombre d’échantillons, et nous a permis d’identifier rapidement les échantillons à examiner à une résolution plus élevée. Dans l’image ci-dessus, l’échelle graphique représente 100 µm. |
Observation tridimensionnelle des structures de l’arbre biliaire d’un foie de souris avec un objectif x30 (UPLSAO30XS [ON : 1,05, DT : 0,8 mm])
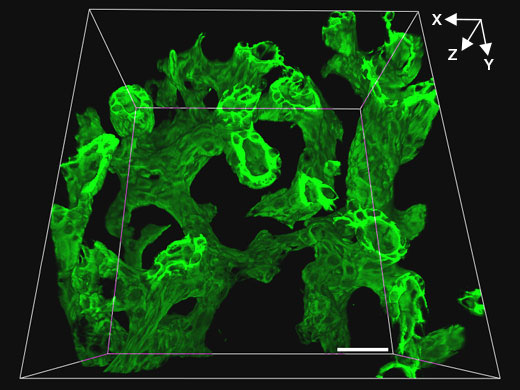 Souris témoin | 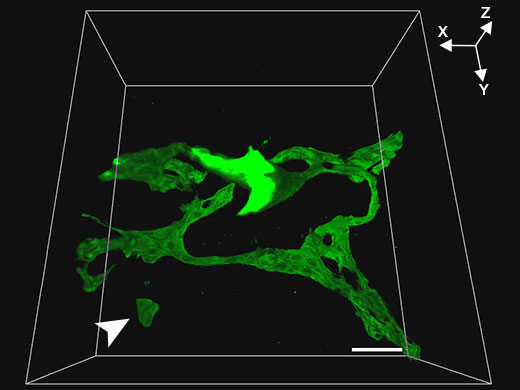 Souris Klf5-LKO |
Related VideosVidéo : Structure de l’arbre biliaire de la souris témoin | Pour obtenir des images tridimensionnelles à une résolution plus élevée, nous avons utilisé le microscope FV3000 et un objectif à immersion dans l’huile de silicone Olympus x30 afin de produire des images tomographiques consécutives (intervalle axial Z de 1 µm) du tissu biliaire (vert, marqueur de cellule épithéliale biliaire CK19) sur des échantillons de tissu hépatique d’une épaisseur de 200 µm clarifiés à l’aide de SeeDB. Cette combinaison a permis une observation en haute résolution des arbres biliaires des souris témoin et Klf5-LKO tout en conservant un large champ de vision. Chez la souris Klf5-LKO, les chercheurs ont observé des agrégats de cellules CK19+ (flèche blanche) séparés spatialement de l’arbre biliaire. |
Commentaire du Dr Okada :
Visualisation tridimensionnelle et analyse structurelle fine de structures biliaires complexes
Dr. Hajime Okada | Dans cette expérience, nous avons analysé les structures 3D d’arbres biliaires liées à un remodelage du foie à la suite d’une lésion hépatique générée par un régime. Les structures biliaires des tissus hépatiques clarifiés des souris Klf5-LKO et témoin ont été comparées au moyen d’un microscope FV3000. Le détecteur très sensible du microscope a permis l’acquisition d’images à large champ de vision avec une résolution et une luminosité élevées, ainsi que des observations plus rapides utilisant moins d’images et de moyennes. Cela a permis d’obtenir de nombreuses images exploitables lors de l’analyse quantitative. De plus, l’objectif à immersion dans l’huile de silicone Olympus a permis une imagerie en profondeur avec une résolution élevée. Nous avons ainsi pu identifier des agrégats de cellules CK19+ séparées spatialement de l’arbre biliaire. Ces résultats suggèrent que la structure biliaire lors du remodelage de tissus dans diverses conditions de lésions hépatiques pourrait être régulée par des mécanismes moléculaires. |
Commentaire du Dr Itoh :
Imagerie tridimensionnelle de foies clarifiés2)
Dr Tohru Itoh | Jusqu’à présent, la recherche médicale et biochimique sur le foie s’appuyait principalement sur des méthodes d’observation 2D conventionnelles utilisant des sections de tissus au lieu d’organes intacts entiers. Toutefois, avec ces méthodes, il est difficile de détecter et de comprendre les « vraies couleurs » du foie dans les conditions physiologiques et diverses pathologies hépatiques. Notre groupe de recherche a mis au point une nouvelle technologie de visualisation pour colorer et clarifier les tissus hépatiques, qui a permis de réaliser avec succès l’observation 3D de la structure biliaire de foies de souris intacts pour la première fois. Cette combinaison de technologies de clarification et de coloration nous a permis de détecter des changements dynamiques de la structure biliaire (remodelage biliaire) et d’effectuer des recherches sur ses mécanismes de régulation et ses fonctions physiologiques. Au cours des expériences présentées ci-dessus, nous avons réussi à établir un système expérimental permettant une visualisation efficace de la structure biliaire en 3D à l’aide du réactif de clarification SeeDB et d’un microscope confocal. Certains des mécanismes moléculaires de remodelage biliaire ont été clarifiés grâce à l’utilisation d’un microscope FV3000. Avec les techniques ici présentées, un microscope confocal est utile pour l’analyse 3D des changements dynamiques des différents tissus et cellules du foie, y compris la structure biliaire. Nous pensons que notre nouvelle technologie de visualisation peut contribuer au perfectionnement de diagnostics et de traitements des pathologies hépatiques ainsi que de la médecine régénérative, grâce à une meilleure compréhension du processus de développement et de régénération du point de vue du remodelage tissulaire et des interactions intercellulaires. 2) Références : K. Kamimoto, K. Kaneko, CY. Kok, H. Okada, A. Miyajima et T. Itoh, « Heterogeneity and stochastic growth regulation of biliary epithelial cells dictate dynamic epithelial tissue remodeling », Elife, 19 juillet 2016 ;5, pii : e15034, doi : 10.7554/eLife.15034. K. Kaneko, K. Kamimoto, A. Miyajima et T. Itoh, « Adaptive remodeling of the biliary architecture underlies liver homeostasis », Hepatology, juin 2015 ;61(6):2056-66, doi : 10.1002/hep.27685, Epub 22 avril 2015. |
L’apport du microscope confocal FV3000 pour notre expérience
Le système intégralement spectral permet une sensibilité élevée
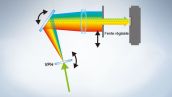
La gamme FV3000 repose sur la technologie de détection TruSpectral d’Olympus, qui diffracte la lumière en la transmettant à travers une unité holographique de phase volumique. Cette technologie permet d’obtenir une luminosité largement supérieure par rapport aux unités de détection spectrale conventionnelles équipées de réseaux de type à réflexion, tout en minimisant la puissance laser requise pour l’observation des tissus profonds.
Les objectifs à immersion dans du silicone pour l’imagerie de cellules vivantes assurent une observation haute résolution en profondeur

L’indice de réfraction de l’huile de silicone (n≈1,40) est proche de celui d’un tissu vivant (n≈1,38), ce qui permet d’effectuer des observations haute résolution en profondeur dans un tissu vivant avec une très faible aberration sphérique causée par la différence des indices de réfraction. De plus, l’huile de silicone ne sèche pas et ne durcit pas ; il n’est donc jamais nécessaire d’en rajouter. Elle est ainsi idéale pour les longues observations à intervalles de temps réguliers.
Remerciements
Cette note d’application a été rédigée avec l’aide des chercheurs suivants :
Dr Hajime Okada, Division of Mammalian Development, Genetic Strains Research Center, National Institute of Genetics
Dr Tohru Itoh, Project Associate Professor, Laboratory of Stem Cell Therapy, Institute for Quantitative Biosciences, University of Tokyo
Produits utilisés pour cette application
a bien été ajouté à vos favoris
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
Not Available in Your Country
Sorry, this page is not
available in your country.





