La división celular es un proceso crucial en todos los organismos vivos. Antes de cada división, la información genética de la célula, que se encuentra almacenada en el ADN, debe ser copiada con exactitud. Este proceso de copia se conoce como replicación del ADN y su regulación adecuada juega un papel vital en la salud y supervivencia de un individuo. Comprender cómo las células regulan la replicación del ADN para lograr exactitud en la duplicación del genoma es una de las preguntas fundamentales en la investigación biomédica. Los errores que se generan durante la replicación del ADN pueden provocar una inestabilidad genómica que conlleva a enfermedades graves en la salud humana, como el cáncer. De hecho, previas investigaciones han estimado que hasta dos tercios de todos los cánceres son causados por la acumulación de errores durante la replicación del ADN.
Conozca a una pionera en el campo de la replicación del ADN
La Dra. Hana Polasek-Sedlackova también enfoca su investigación en cómo las células duplican su ADN. Hana tuvo la oportunidad de acceder al entorno del laboratorio y a la investigación del ADN en una etapa temprana de su vida mientras participaba en una pasantía de investigación científica en la Universidad de Masaryk durante la escuela secundaria. Desde entonces, es una figura en ascenso en este ámbito. La tesis universitaria de Hana le valió el renombrado Premio Universitario de Grado en Ciencias de la Vida a nivel mundial, al que a menudo se hace referencia como el premio «Nobel Junior». Después de graduarse, se le otorgó la beca de la Fundación Novo Nordisk para cursar el programa de doctorado de Biociencias en Copenhague. Por medio de esta beca, Hana se incorporó al laboratorio del profesor Jiri Lukas, en el Centro de Investigación de Proteínas de la Fundación Novo Nordisk en la Universidad de Copenhague, como estudiante de doctorado y continuó allí su formación postdoctoral. Su trabajo de investigación fue publicado en las reconocidas revistas científicas, como Science y Nature. Hace poco, ha iniciado su propio grupo de investigación independiente en el Instituto de Biofísica (IBP) de la Academia de Ciencias de la República Checa en Brno, República Checa.
Una de sus contribuciones a la ciencia fue resolver la paradoja del mantenimiento del minicromosoma (MCM), que ha sido debatida desde los años 90 entre los científicos del ámbito. Se describían resultados aparentemente contradictorios entre la microscopía y otros métodos científicos utilizados para dilucidar los eventos durante la replicación. Tal contradicción fue resuelta con éxito gracias al respaldo de nuestra estación de cribado de alto contenido scanR, del cual Hana es un ávido usuario. Conocimos a Hana durante una reunión de usuarios de scanR, que tuvo lugar en el Instituto de Genética Molecular en Praga, y se aprovechó la oportunidad para preguntarle sobre su experiencia con el sistema y el impacto que éste ha causado en su investigación.
Entrevista con Hana Polasek-Sedlackova
Evident: ¿Cuando inició su grupo de investigación en el IBP, la estación de cribado de alto contenido scanR fue el primer microscopio que adquirió para el grupo? ¿Qué la motivó a elegir este sistema?
Hana Polasek-Sedlackova: Durante mi experiencia profesional, he tenido la oportunidad de trabajar con varios microscopios. Gracias mis observaciones, me di cuenta rápidamente de que hay tres parámetros importantes que deben considerarse al elegir un microscopio. Estos parámetros son el hardware, el software y servicio al cliente. Tras sopesar cuidadosamente estos criterios, descubrí que el scanR es el mejor sistema de cribado de alto contenido.
Primero, el scanR tiene un sistema de hardware sencillo y flexible que puede adaptarse a los requisitos experimentales específicos del usuario. Ofrece funciones de procesamiento de imágenes que van desde el campo amplio hasta la superresolución, técnicas avanzadas de microscopía de fluorescencia y la capacidad de digitalizar células vivas, etc.
Segundo, el scanR presenta una plataforma de análisis y adquisición fácil de usar a carácter intuitivo que permite a los usuarios obtener datos de alta calidad y en cantidad a través de un corto período de tiempo. El análisis multiparamétrico instantáneo de los datos adquiridos a partir de las imágenes permite la inspección inmediata de estos últimos y una mayor optimización de los parámetros de adquisición, como la útil función Rescan. [La función Rescan (Nuevo escaneo) ejecuta un rápido escaneo de la muestra usando una resolución más baja para identificar los objetos de interés, que después pueden ser escaneados nuevamente con una resolución más alta.]
Tercero, Evident ofrece un servicio al cliente excepcional. Sus especialistas de aplicaciones, dedicados al mantenimiento de microscopios y soporte analítico, son profesionales consumados que siempre están preparados para ayudar a los usuarios a través de las necesidades, deseos o problemas que se plantean. Y gracias a la Fundación Científica Checa y a los fondos internos de nuestro instituto pudimos comprar y llevar la tecnología scanR a mi nuevo instituto.
Medición de la dinámica de replicación del ADN mediante el sistema scanR. (A) Representación esquemática de una célula humana con zoom a la horquilla de replicación única: una unidad básica de replicación del ADN. Las sondas fluorescentes específicas permiten estudiar la unión en el complejo MCM del ADN (Halo), el ensamblaje de la helicasa de forma activa a partir de las proteínas CMG (GFP) y la síntesis del ADN (EdU). (B) Análisis multiparamétrico scanR de los eventos clave al momento de la replicación del ADN en células U2OS de un osteosarcoma humano. (C) Galería de imágenes que ilustran las dinámicas de las proteínas en la replicación del ADN a lo largo del ciclo celular.
Evident: ¿En qué forma el sistema scanR ha permitido el avance de su investigación? Puede darnos un ejemplo en donde el sistema scanR haya sido de gran utilidad para obtener nuevos conocimientos.
Hana: El sistema scanR nos ha permitido descubrir dinámicas fundamentales en el campo de la replicación del ADN. A lo largo de mi investigación, este sistema ha formado parte esencial en la resolución de la paradoja sobre el MCM: un enigma persistente sobre el comportamiento del complejo proteico MCM2-7. El complejo MCM forma el núcleo de la helicasa replicativa, que es responsable de desenrollar el ADN de doble hélice para garantizar una duplicación exacta. Durante años, los investigadores quedaban vacilantes con respecto a dos hechos paradójicos ampliamente difundidos: (1) ¿Por qué las células portan un enorme exceso de proteínas MCM inactivas si sólo entre el 5 % y el 10 % son usadas como helicasas replicativas activas? (2) ¿Por qué nunca se han visualizado proteínas MCM eucariotas en los sitios de replicación dentro de una célula mediante métodos de microscopía?
Hace poco, hemos revisado este persistente atascamiento conceptual y brindado aclaraciones a la paradoja del MCM. Con ayuda de la edición del genoma CRISPR-Cas9 y las imágenes de alto contenido scanR, dilucidamos desafíos de larga data al procesar en imágenes las horquillas de replicación del ADN en células vivas y revelamos la nueva vía homeostática de los orígenes de la replicación del ADN necesaria para proteger la integridad del genoma.1-2
Se descubrió que el excedente de proteínas MCM inactivas sirve como sitio de pausa de replicación natural (NRPS), cuyo fin es ajustar la velocidad de la horquilla de replicación para minimizar los errores durante la replicación del ADN. A partir de este concepto, los NRPS representan otra capa de control de velocidad para la horquilla de replicación, cuya explotación es frecuente por las células cancerosas; esto los convierte en un nuevo objetivo atractivo para el tratamiento oncológico. Actualmente, mi equipo de investigación estudia las vías reguladoras más apicales de los orígenes de la replicación del ADN y su contribución a los estados fisiopatológicos del desarrollo del tejido humano.

Descubrimiento de variantes proteicas de los complejos MCM con el scanR que esclarece la paradoja del MCM. (A) Protocolo de marcado HaloTag para distinguir grupos de proteínas MCM. El cribado de alto contenido del scanR reveló diferentes dinámicas de los complejos proteicos MCM parentales y nacientes durante el programa de replicación del ADN. (B) Modelo que describe la función distinta de los MCM parentales y nacientes durante la replicación del ADN. Si bien los MCM parentales se convierten principalmente en helicasas CMG activas, los MCM nacientes permanecen en gran medida inactivos pero funcionan como sitios de pausa de replicación naturales clave para determinar el movimiento adecuado de la horquilla de replicación.
Evident: ¿Para qué aplicaciones usa el sistema scanR y qué tipo de muestras son digitalizadas?
Hana: En nuestra investigación, usamos una amplia variedad de cultivos tisulares, que derivan en su mayoría de muestras humanas. Entre los más frecuentes están las líneas celulares cancerosas provenientes de diversos tejidos (p. ej., hueso, mama, ovario, cuello uterino o riñón), así como sus contrapartes no cancerosas (p. ej., células madre y fibroblastos). Hay casos en los que también usamos muestras de animales en nuestros estudios. Estas muestras son usadas en combinación con diversos enfoques de la biología celular, bioquímica y genética para identificar nuevas proteínas o vías reguladoras completas que desempeñan un papel importante en la duplicación del genoma sin errores. También, investigamos cómo se regulan estas nuevas proteínas o vías en las células cancerosas, y si esto las dota de una ventaja en el desarrollo de tumores. Si descubrimos una posible relación, podremos usar nuestro conocimiento para desarrollar enfoques novedosos para el tratamiento oncológico.
Nuestro proceso experimental típico para identificar nuevos factores proteicos en la replicación del ADN implica perfilar una gran población de células individuales, y el sistema scanR ha sido de gran ayuda en este sentido. Por ejemplo, si identificamos una nueva proteína candidata, primero se la reduce en las células a través de varios métodos. Después, mediante imágenes de alto contenido, se monitoriza la precisión de la replicación del ADN paso a paso. La ventaja de utilizar células en nuestros experimentos es que se puede observar el proceso de duplicación del genoma en su entorno natural, pero su complejidad también puede plantear desafíos a nivel de la interpretación de los datos.
Por tanto, cuando se estudia la replicación del ADN, es necesario examinar cuidadosamente cómo la disminución de nuestro gen de interés afecta otros procesos celulares: el ciclo celular, la respuesta al daño del ADN, la transcripción, el mantenimiento de la cromatina, el mantenimiento de regiones cromosómicas específicas, la estabilidad general del genoma, etc. Estos perfiles ecuánimes y cuantitativos de grandes poblaciones unicelulares, obtenidos en un período de tiempo muy corto, nos ayudan enormemente a interpretar nuestros datos rápidamente y, por ende, favorecen el avance eficiente de nuestra investigación.
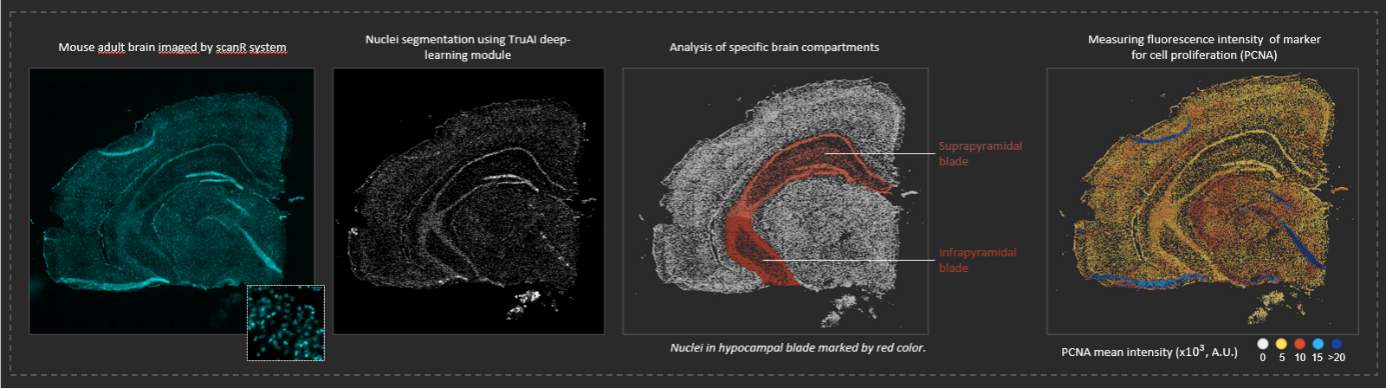
Análisis de imágenes de células individuales en el cerebro de un ratón adulto usando el módulo de aprendizaje profundo TruAI. La preparación de la muestra de cerebro fue llevada a cabo por la Dra. Jana Krejci (Departamento de Epigenética de la Biología Celular en el Instituto de Biofísica).
Solía pasar horas frente al microscopio adquiriendo imágenes manualmente y, después, más horas frente al PC para analizarlas. Se requerirían meses para completar estos experimentos. Pero, ahora, con el scanR, todo puede ejecutarse en pocas horas.
Evident: ¿Cómo se benefician estas aplicaciones de las capacidades del cribado de alto contenido y de la técnica de citometría basada en las imágenes del sistema?
Hana: En el campo de la biología celular, creo que combinar información cuantitativa ecuánime con imágenes es una forma confiable y precisa de representar procesos celulares vitales. El sistema scanR ofrece una solución sofisticada e inteligente para ello. Tal y como mencioné antes, el sistema scanR permite la adquisición y el análisis de muestras de forma totalmente automatizada, por lo tanto, viabiliza la obtención de información cuantitativa e imparcial sobre los procesos celulares. Es importante destacar que con un solo clic, es posible inspeccionar directamente las imágenes que subyacen tras los datos.
Además, es posible crear galerías de imágenes ecuánimes que ilustran la dinámica de las proteínas de interés a lo largo de la vida celular. Gracias a esta conjugación única entre imagen y datos, es posible afinar aún más el análisis de los procesos celulares en compartimentos específicos, como a nivel del núcleo o el citoplasma, así como en compartimentos subnucleares como a nivel de los focos de reparación del ADN, etc. Este complemento visual de los datos cuantitativos ayuda a resolver problemas biológicos de manera más efectiva.
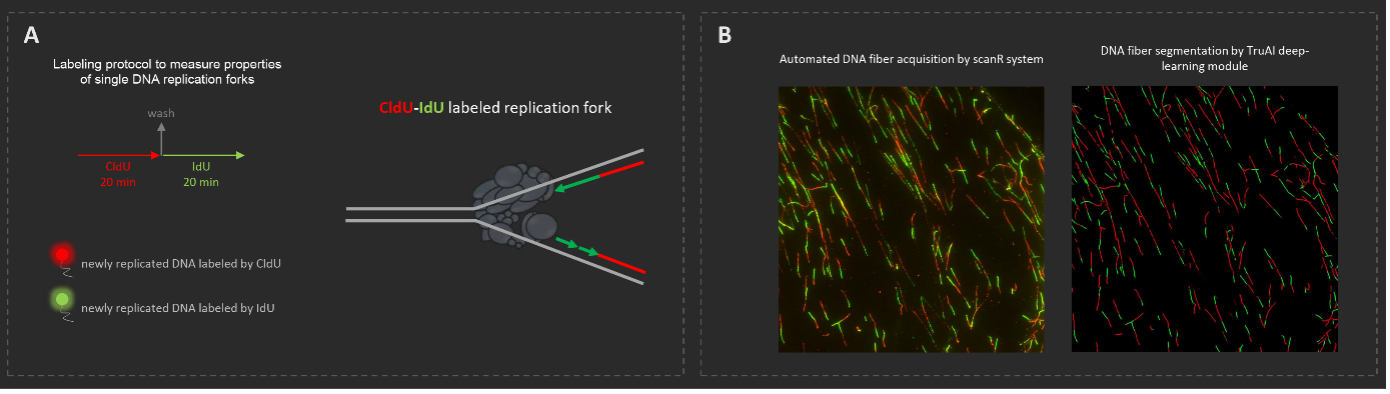
Ejemplo de un trabajo en curso relativo a la adquisición y el análisis automatizados de fibras de ADN al combinar los sistemas scanR y cellSens™. (A) Protocolo de marcado CldU-IdU para visualizar horquillas de replicación únicas. (B) Ejemplos de la adquisición y segmentación automatizadas de la fibra de ADN por parte del sistema scanR y el software cellSens™.
Evident: ¿El sistema, como herramienta de biología celular y biología molecular, ha cambiado la forma en que abordaba la microscopía? ¿Aprovecha el potencial del aprendizaje profundo TruAI™ del sistema?
Hana: Durante mi doctorado, trabajando en el laboratorio del profesor Jiri Lukas, conocí las imágenes de alto contenido y el sistema scanR. Esta tecnología revolucionaria cambió por completo mi perspectiva general sobre la biología celular y la investigación. Me impresionó particularmente su capacidad para proporcionar un análisis ecuánime y totalmente automatizado de los procesos celulares. Dada la actual crisis de reproducibilidad en la investigación científica, creo firmemente que los investigadores deberían buscar herramientas que les permitan obtener y analizar datos de forma totalmente automatizada, a fin de proporcionar una representación ecuánime de los procesos celulares.
En esta era de inteligencia artificial (IA), el análisis de imágenes ha avanzado más allá de las antiguas limitaciones. El sistema scanR ahora ofrece el módulo de aprendizaje profundo TruAI, que ha ampliado el alcance de mi investigación. En el pasado, me centré en analizar células individuales a partir de grandes poblaciones de líneas celulares humanas de cultivo. Sin embargo, con el módulo de IA, es posible ejecutar análisis de elevada precisión a partir de imágenes de células individuales presentes en secciones de órganos.
Otro ejemplo de cómo podemos superar los límites de nuestras capacidades de análisis de imágenes se da al combinar los sistemas scanR y cellSens™. Esto nos permite adquirir y analizar de forma automatizada moléculas individuales, como las horquillas de replicación de ADN que se preparan mediante la técnica de fibra de ADN. Estos son algunos ejemplos de nuevas aplicaciones en las que trabajamos en mi laboratorio; asimismo, esperamos que otros investigadores se beneficien pronto de estas herramientas para hacer grandes descubrimientos en el campo de la biología celular.
Evident: ¿Qué es lo que más le gusta del sistema scanR? ¿Cómo es su aceptación entre sus asociados de investigación?
Hana: Hay muchas características del sistema scanR que aprecio; pero, si debo resaltar solo una, sería la conexión única entre la adquisición y el análisis automatizados, la cual permite llevar a cabo análisis multiparamétricos instantáneos de las imágenes adquiridas. Esta función me impresionó cuando cursaba mi doctorado, como también era el caso de cada nuevo usuario de nuestra pequeña comunidad de usuarios de scanR en nuestro instituto.
Uno de mis colegas lo resumió mejor cuando comentó: «Solía pasar horas frente al microscopio adquiriendo imágenes manualmente y, después, más horas frente al PC para analizarlas. Se requerirían meses para completar estos experimentos. Pero, ahora, con el scanR, todo puede ejecutarse en pocas horas».
Para obtener más información sobre el sistema scanR
Expresamos nuestro sincero agradecimiento a la Dra. Hana Polasek-Sedlackova por compartir su experiencia con respecto a la estación de cribado de alto contenido scanR. Para más información sobre el sistema scanR y nuestras otras soluciones de investigación para las ciencias biológicas, contacte directamente con Evident o con su representante de ventas local.
Dra. Hana Polasek-Sedlackova, investigadora del DNA: Fotografía realizada por Jana Mensatorova.
Referencias
- Polasek-Sedlackova H, Miller TCR, Krejci J, Rask MB, Lukas J. Solving the MCM paradox by visualizing the scaffold of CMG helicase at active replisomes. Nat Commun. 2022 Oct 14;13(1):6090. doi: 10.1038/s41467-022-33887-5. PMID: 36241664; PMCID: PMC9568601. https://www.nature.com/articles/s41467-022-33887-5
- Sedlackova, H., Rask, M.-B., Gupta, R., Choudhary, C., Somyajit, K., Lukas, J. (2020, 21 de octubre). Equilibrium between nascent and parental MCM proteins protects replicating genomes. Nature News. https://www.nature.com/articles/s41586-020-2842-3
Contenido relacionado
Segmentación de instancias simplificada de células y núcleos mediante el aprendizaje profundo
Localización de proteínas de levadura clasificadas con la tecnología de aprendizaje profundo TruAI™

