Previsión de fenotipos de núcleos multiclase para análisis de fármacos mediante el aprendizaje profundo
Introducción
La cuantificación del movimiento dinámico de las proteínas, como los receptores nucleares en respuesta a los fármacos, requiere métodos de segmentación nuclear sólidos que usen colorantes y tinturas. Sin embargo, este proceso presenta limitaciones notables: los procedimientos de tinción de células vivas requieren mucho tiempo, son costosos y pueden provocar fototoxicidad, fotoblanqueo y la inactivación de los compuestos fotosensibles (p. ej., R18811).
Para superar estos desafíos, se ha desarrollado una red neuronal (neural network, NN) con el software de procesamiento de imágenes cellSens™, dotado de la tecnología de aprendizaje profundo TruAI ™, a fin de identificar núcleos celulares sin usar marcadores fluorescentes. Después, se ha formado a la NN para poder distinguir los diferentes fenotipos celulares en respuesta a los fármacos. Aquí, se aplica el proceso de trabajo de la NN para prever la dinámica intracelular del receptor androgénico (RA) en células de cáncer de próstata vivas tratadas con medicamentos dirigidos al RA.
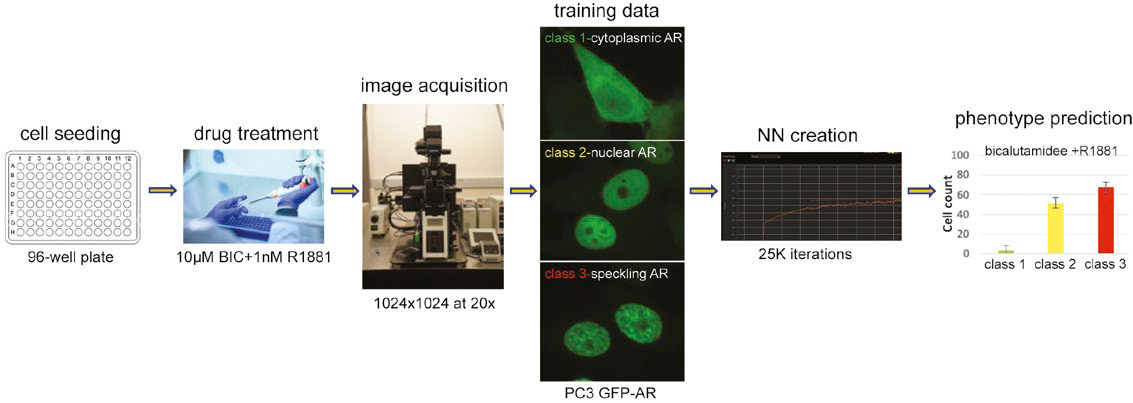
Figura 1. Proceso de trabajo para predecir fenotipos de núcleos multiclase con una NN formada.
Tras la activación, el RA citoplasmático se traslada al núcleo donde se alojará en los sitios de transcripción activos, lo que puede visualizarse como un patrón moteado distinto mediante la microscopía confocal. Para prever la dinámica del RA, primero se formó una NN que detecta los núcleos celulares sin usar tinciones nucleares. A continuación, se clasificaron las células en función de la translocación nuclear del RA con GFP y del estado de transactivación (moteado): ambas lecturas eran fundamentales en el ensayo para comprobar la capacidad de los compuestos al RA diana. Por último, se demostró que el proceso de trabajo de la NN formada predecía con éxito estos fenotipos críticos de RA sin tinción nuclear.
Ventajas
- Identificación de fenotipos de núcleos sin usar marcados fluorescentes.
- Ahorro de tiempo y costos (Esp. costes) con respecto al uso de tintes nucleares (tinción y procesamiento de imágenes).
- Reducción de la fototoxicidad y fotoblanqueo.
- Preservación de la actividad de los compuestos fotosensibles.
Métodos
Se generó un conjunto de datos de formación que consta de tres fenotipos principales de RA, comúnmente observados en las respuestas a los fármacos. Se usó una mitad de las imágenes para generar la NN y la otra mitad para evaluar su desempeño. Finalmente, se aplicó la NN multiclase formada a un conjunto de datos independientes sin tinción nuclear.
Preparación de los datos de la formación a partir de tres fenotipos de RA
Primero, durante la formación de la NN, se generó un conjunto de datos formativos, que consistía en tres fenotipos principales de RA:
- RA citoplasmático inactivo (control sin tratamiento [NTC])
- RA nuclear unido a ligando pero inactivo (10 μM de bicalutamida)
- RA nuclear activo de patrón moteado (R1881 de 1 nM)
Para generar datos reales sobre el terreno, se tiñeron los núcleos con SiR-DNA. Se adquirieron imágenes de 1024 × 1024 píxeles con un objetivo UPLSAPO de 20X mediante el escáner galvanométrico del microscopio confocal láser FV3000 de Olympus. En esta etapa, el conjunto de datos completo (60 imágenes) se dividió en dos partes iguales: una destinada al conjunto de formación, y la otra destinada al conjunto de validación (30 imágenes cada una). Para formar la NN, se usaron 10 imágenes del RA con GFP, proyectadas a máxima intensidad a partir de cada uno de los tres fenotipos de RA (50 % de los datos se basaban en un total de 30 imágenes) con una tinción nuclear.
![Figura 2. Preparación de los datos de formación del fenotipo de RA multiclase basado en la tinción de núcleos. [Izq.] Verde: AR-GFP. Violeta: SiR-DNA. [Derecha] Evaluación de los conjuntos de datos reales en el terreno.](https://static1.olympus-lifescience.com/data/Image/Application/truai-deep-learning/figure-02.jpg?rev=4DB4)
Figura 2. Preparación de los datos de formación del fenotipo de RA multiclase basado en la tinción de núcleos. [Izq.] Verde: AR-GFP. Violeta: SiR-DNA. [Derecha] Evaluación de los conjuntos de datos reales en el terreno.
Optimización de la realidad del terreno
La Figura 2 demuestra los datos reales de terreno evaluados por la NN, con el RA citoplásmico de verde, el RA nuclear de amarillo y el RA moteado de rojo. Al usar este mapa de segmentación con una codificación cromática, se ajusta la realidad del terreno al corregir de forma manual los errores en la detección y segmentación nuclear, como también al reclasificar núcleos mal categorizados.
Creación de redes neutrales con la tecnología de aprendizaje profundo TruAI
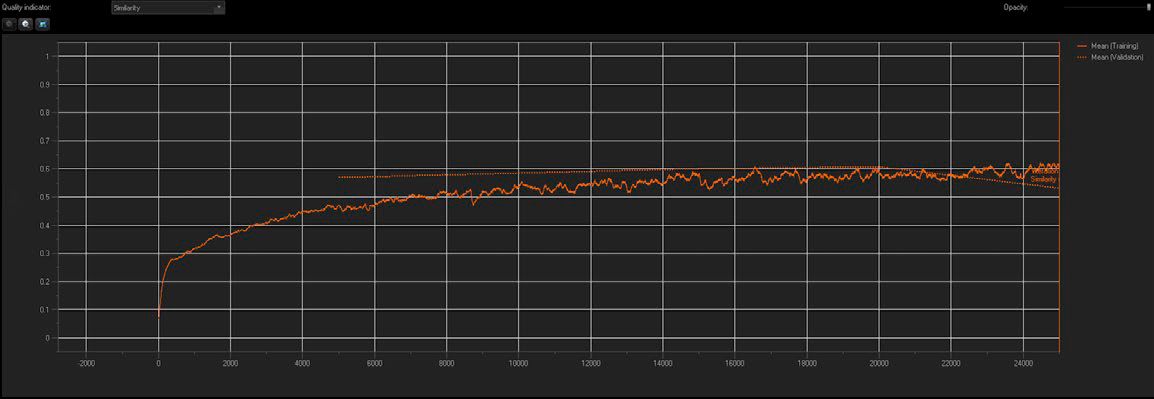
Figura 3. La formación de la NN informa el índice de similitud y los valores de validación promedio en tiempo real.
Se usó la tecnología TruAI para crear una NN estándar con configuraciones de múltiples clases a partir de 10 imágenes de formación, asociadas a cada fenotipo de RA. La calidad y precisión de la NN se mostró en tiempo real. Tal como se muestra en la Figura 3, el índice de similitud aumentó con cada iteración sucesiva durante el proceso de formación hasta que se alcanzó una cota, lo que indica una saturación en la formación (flecha hacia abajo). Se crearon puntos de control cada vez que se alcazaba 5000 iteraciones (20 % de progreso) y el punto de control óptimo se guardó como una NN formada junto con su valor de índice de similitud.
Resultados
Validación de una red neuronal multiclase sobre datos ciegos
Se validó el modelo de NN multiclase creado por medio del 50 % de las imágenes no usadas (datos ciegos), destinadas a la realidad del terreno o a la formación NN (datos ciegos). A continuación, se usaron los resultados de validación para determinar los parámetros del modelo mediante la aplicación de un filtro de área (mín. 49,83 μm - máx. 555,44 μm) a fin de eliminar los restos celulares y las células mal segmentadas. El modelo de la NN formada previó fenotipos de RA multiclase de forma independiente sin ninguna tinción nuclear. La Figura 4 muestra la distribución de las clases de fenotipo a través del uso de los datos provenientes de 10 imágenes.
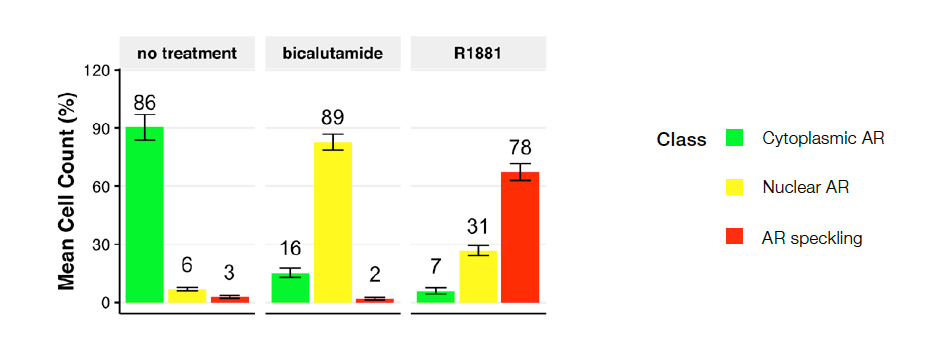
Figura 4. Distribución de clases de células según las siguientes condiciones: control sin tratamiento (izquierda), bicalutamida (centro) o R1881 (derecha). El gráfico muestra el porcentaje de células categorizadas como clase 1 (RA citoplasmático), clase 2 (RA nuclear) y clase 3 (RA moteado). Ningún control de tratamiento dio como resultado en su mayoría células de clase 1. El bicalutamida (antagonista del RA) indujo a una desviación hacia la clase 2, y el R1881 (agonista del RA) dio como resultado la clase 3 del RA activo alojado en los sitios de transcripción activos.
Previsión de fenotipos de RA multiclase en conjuntos de datos independientes
A continuación, para evaluar el modelo de la NN multiclase en imágenes independientes, se preparó un nuevo conjunto de datos con dos antiandrógenos clínicos: bicalutamida y enzalutamida (Figura 5).
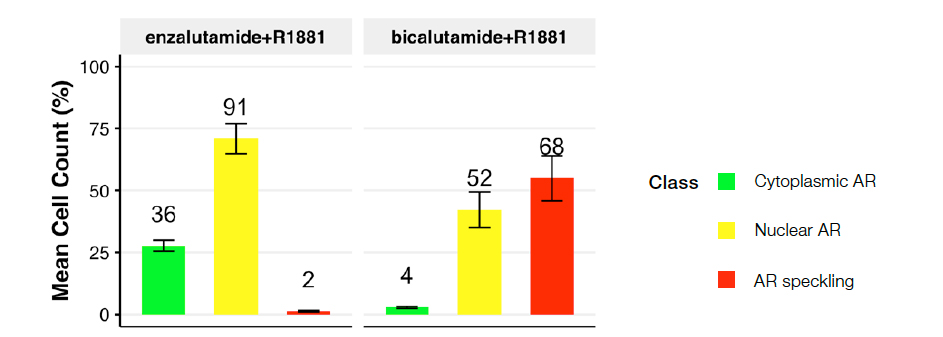
Figura 5. Distribución de las clases de células según las siguientes condiciones: R1881 más enzalutamida (izquierda) o bicalutamida (derecha). El gráfico muestra la distribución de las clases de núcleos prevista a través de las condiciones de tratamiento.
El tratamiento con el enzalutamida (antiandrógeno de segunda generación) dio lugar a una población prominente de RA citoplasmáticos (clase 2) con pocos o ningún RA moteado (clase 3), lo que define un bloqueo eficaz de la activación del RA inducido por ligando. Por el contrario, el bicalutamida (antiandrógeno más antiguo y menos eficaz) resultó en una notable desviación hacia el RA activo (clase 3), lo que sugiere su incapacidad para bloquear eficazmente la activación del RA inducida por R1881 de 1 nM. Estos datos confirman estudios anteriores2.
Conclusión
Al utilizar el microscopio confocal Olympus FV3000 y el software cellSens™ con el módulo de aprendizaje profundo TruAI™, se generó una red neuronal (NN) a fin de detectar y segmentar núcleos no teñidos y subclasificar células según los cambios fenotípicos del RA en respuesta a los fármacos. Este enfoque tiene el potencial de aumentar la eficiencia de los análisis de fármacos al reducir el tiempo de prueba (configuración experimental) y los costos.
Asimismo, las NN pueden aumentar la calidad de los datos debido a la reducción de artefactos, como la fototoxicidad, el fotoblanqueo y la inactivación de compuestos fotosensibles, y a la detección de cambios sutiles que el investigador o los proyectos analíticos tradicionales de imágenes pueden pasar por alto. Este enfoque de aprendizaje profundo puede aplicarse de manera más amplia en la subclasificación de poblaciones de células conforme con los parámetros nucleares o celulares que van desde la localización de proteínas hasta los cambios morfométricos.
Referencias
- Risek B, Bilski P, Rice AB, Schrader WT. Androgen receptor-mediated apoptosis is regulated by photoactivatable androgen receptor ligands [La apoptosis provocada por el receptor androgénico se regula por ligandos fotoactivables del receptor androgénico]. Mol Endocrinol. Septiembre de 2008; 22 (9): 2099-115. doi: 10.1210/me.2007-0426. Epub, 18 de junio de 2008. pmid: 18562628; pmcid: PMC2631375.
- Sugawara T, Lejeune P, K hr S, Neuhaus R, Faus H, Gelato KA, Busemann M, Cleve A, Lücking U, von Nussbaum F, Brands M, Mumberg D, Jung K, Stephan C, Haendler B. BAY 1024767 blocks androgen receptor mutants found in castration-resistant prostate cancer patients [Bloqueo de mutantes del receptor androgénico por BAY 1024767 encontrados en pacientes con cáncer de próstata resistente a la castración. Oncotarget. 2 de febrero de 2016; 7 (5): 6015-28. doi: 10.18632 / oncotarget.6864. pmid: 26760770; pmcid: PMC4868737.
Autores
- Harish Sura, Katherin Patsch y Seungil Kim del Instituto Lawrence J. Ellison en Medicina Transformativa, Universidad del Sur de California, Los Ángeles, CA.
- Shohei Imamura, Gerente de proyectos estratégicos, Olympus Corporation of the Americas
Productos usados para esta aplicación
se ha añadido correctamente a sus marcadores
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
Not Available in Your Country
Sorry, this page is not
available in your country.