Observación 3D de hígado de ratón clarificado usando el microscopio FLUOVIEW FV3000
Observación tridimensional del hígado con alta resolución
La observación 3D de muestras de tejido grueso suele requerir el uso de un microscopio de excitación de dos fotones debido al aumento de la absorción y la dispersión de la luz que se producen al visualizar la muestra de tejido a mayor profundidad. Aunque el hígado es un tejido con una alta dispersión, la utilización de métodos de transparentación combinados con los dispositivos ópticos adecuados permite realizar observaciones en 3D de tejido grueso usando el microscopio confocal FV3000. En este experimento, el objetivo de inmersión en aceite de silicona 30x de Olympus con una apertura numérica (A. N.) de 1,05 y una distancia de trabajo (D. T.) de 0,8 mm nos permitió realizar una observación 3D de alta resolución de la estructura del árbol biliar en muestras de hígado de ratón clarificadas.
Resumen del experimento
Clarificación óptica de muestras de tejido de hígado de ratón
Muestra de tejido de hígado de ratón |
|
El tejido se tiñe para fluorescencia mediante inmunotinción |
|
El tejido del hígado se clarifica usando SeeDB |
Cortesía de: K. Kamimoto, K. Kaneko, CY. Kok, H. Okada, A. Miyajima, and T. Itoh, «Heterogeneity and stochastic growth regulation of biliary epithelial cells dictate dynamic epithelial tissue remodeling» Elife, 2016 Jul. 19;5, pii: e15034, doi: 10.7554/eLife.15034.
Protocolo del experimento para visualizar una red biliar compleja en 3D. Después de recoger el tejido, los tejidos biliares se someten a inmunotinción para visualizar una red biliar compleja en tres dimensiones. Las muestras inmunoteñidas se clarifican posteriormente con SeeDB1).
1) Referencia: Ke MT, S. Fujimoto, and T. Imai, «SeeDB: a simple and morphology-preserving optical clearing agent for neuronal circuit reconstruction» Nat Neurosci, 2013 Aug; 16 (8): 1154–61, doi: 10.1038/nn.3447, Epub 2013 Jun. 23, PMID: 23792946
Observación tridimensional de estructuras del árbol biliar en hígado de ratón dañado mediante un objetivo 20x (UPLSAPO20X (A.N.: 0,75, D.T.: 0,6 mm)
 | El objetivo del experimento era medir cuantitativamente la longitud y el grosor de los ramales de la estructura del árbol biliar en hígados de ratón dañados. Para el análisis inicial de las muestras, se usó un objetivo seco 20x en el FV3000 para obtener imágenes tomográficas consecutivas de campo de visión amplio del tejido biliar (marcador verde de célula epitelial biliar CK19) en muestras de tejido de hígado clarificadas de 200 µm de grosor. Esta configuración permitió llevar una observación cuantitativa efectiva de muchas muestras, y gracias a ella se pudieron encontrar muestras para examinarlas a una resolución más alta. En la imagen anterior, la barra de escala representa 100 µm. |
Observación tridimensional de estructuras del árbol biliar en hígado de ratón dañado con un objetivo 30x (UPLSAPO30XS [A. N.: 1,05; D. T.: 0,8 mm])
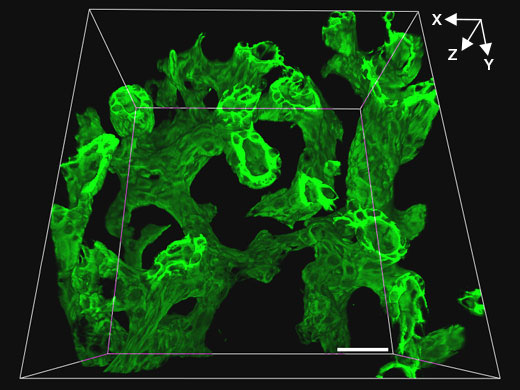 Ratón de control | 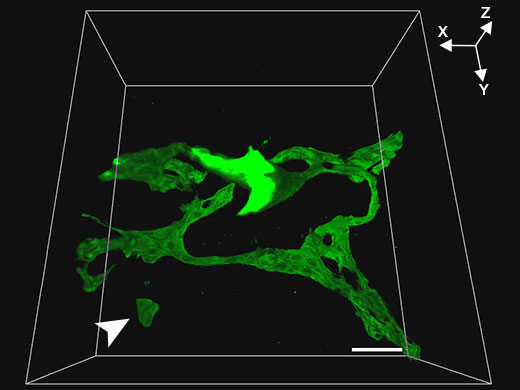 Ratón Klf5-LKO |
Videos asociadosVideo: Estructura del árbol biliar del ratón de control | Con el fin de obtener imágenes tridimensionales de resolución superior, se usó el microscopio FV3000 y un objetivo de inmersión en aceite de silicona 30x de Olympus para obtener imágenes tomográficas consecutivas (intervalo axial en Z de 1 µm) de tejido biliar (marcador celular epitelial biliar CK19, verde) en tejido hepático de 200 µm de grosor clarificado con SeeDB. Esta combinación permitió desarrollar una observación en alta resolución de los árboles biliares del ratón de control y el ratón Klf5-LKO manteniendo un campo de visión amplio. En el ratón Klf5-LKO, los investigadores observaron agrupaciones de células CK19+ (flecha blanca), separadas espacialmente, del árbol biliar. |
Comentario del Dr. Okada:
Visualización tridimensional y análisis estructural detallado de estructuras biliares complejas
Dr. Hajime Okada | En este experimento, se analizaron las estructuras 3D de los árboles biliares relacionadas con el remodelado hepático después de una lesión en el hígado inducida por la dieta. La estructura biliar en tejidos de hígado clarificados en el ratón Klf5-LKO y el ratón de control se compararon usando un microscopio FV3000. El detector altamente sensible del microscopio permitió adquirir imágenes de campo de visión amplio con altos niveles de resolución y brillo, y con la misma rapidez que las observaciones con menos imágenes y promedio. Todo ello, nos permitió conseguir imágenes que podían usarse para efectuar análisis cuantitativos. Además, el objetivo de inmersión en aceite de silicona de Olympus permitió procesar imágenes en profundidad con alta resolución, lo que nos permitió encontrar agrupamientos de células CK19+, separadas espacialmente, del árbol biliar. Estos resultados sugieren que la estructura biliar en el remodelado de tejido bajo diversas condiciones de lesiones hepáticas puede regularse usando algunos mecanismos moleculares. |
Comentario del Dr. Itoh:
Procesamiento de imágenes tridimensionales de hígados clarificados2)
Dr. Tohru Itoh | Hasta el momento, la investigación médica y los estudios bioquímicos centrados en el hígado confiaban en los métodos de observación 2D convencionales usando secciones de tejido en lugar de órganos enteros intactos. No obstante, con estos métodos resulta complicado detectar y entender los «colores reales» del hígado bajo condiciones fisiológicas y las diversas condiciones de las enfermedades hepáticas. Nuestro grupo de investigación desarrolló una nueva tecnología de visualización para marcar y clarificar tejidos del hígado, que nos permitió llevar por primera vez la observación 3D de la estructura biliar en hígados intactos de ratón. Esta combinación de tecnología de tinción y clarificación nos permitió detectar los cambios estructurales biliares dinámicos (remodelado biliar) y estudiar sobre sus mecanismos reguladores y sus funciones fisiológicas. En los experimentos mencionados anteriormente, pudimos establecer un sistema experimental para visualizar la estructura biliar en 3D de forma efectiva usando el reactivo de clarificación SeeDB y un microscopio confocal. La utilización de un microscopio FV3000 permitió clarificar algunos de los mecanismos moleculares para el remodelado biliar. Con las técnicas descritas en este documento, el microscopio confocal resulta útil para desarrollar análisis en 3D relativos a los cambios dinámicos de los diversos tejidos y las células en el hígado, como la estructura biliar. Esperamos que nuestra nueva tecnología de visualización pueda contribuir al desarrollo de dispositivos de diagnóstico y terapias para las afecciones hepáticas y la medicina regenerativa. Esto se conseguirá gracias a la comprensión del proceso de desarrollo y regeneración desde el punto de vista del remodelado de tejido y las interacciones entre las células. 2) Referencias: K. Kamimoto, K. Kaneko, CY. Kok, H. Okada, A. Miyajima, and T. Itoh, «Heterogeneity and stochastic growth regulation of biliary epithelial cells dictate dynamic epithelial tissue remodeling». Elife, 2016 Jul. 19;5, pii: e15034, doi: 10.7554/eLife.15034. K. Kaneko, K. Kamimoto, A. Miyajima, and T. Itoh, «Adaptive remodeling of the biliary architecture underlies liver homeostasis» Hepatology, 2015 Jun.;61(6):2056-66, doi: 10.1002/hep.27685, Epub 2015 Apr. 22. |
¿Cómo el microscopio confocal FV3000 favoreció a nuestro experimento?
Sistema completamente espectral que proporciona una alta sensibilidad
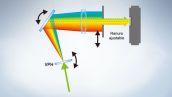
La serie FV3000 emplea la tecnología TruSpectral de Olympus que difracta la luz por medio de la transmisión a través de la unidad de holograma de fase de volumen. Esta tecnología proporciona una emisión de luz mucho mayor que la de las unidades de detección espectral convencionales con rejillas de tipo reflexión, y minimiza la potencia láser necesaria para llevar una observación de tejido profundo.
Objetivos de inmersión en silicona para una observación en profundidad con alta resolución de imágenes de células vivas

El índice de refracción del aceite de silicona (ne≈1,40) es muy similar al del tejido vivo (ne≈1,38), lo que permite llevar observaciones de alta resolución en profundidad dentro del tejido vivo, minimizando la aberración esférica debida a las diferencias entre los índices de refracción. El aceite de silicona no se seca ni se endurece, por lo que no es necesario volver a rellenarlo. Esto lo hace ideal para observaciones de intervalo de tiempo prolongado.
Reconocimientos
Esta nota de aplicación has sido preparada con la ayuda de los siguientes investigadores.
División de desarrollo de mamíferos, Centro de investigación de cepas genéticas, Instituto Nacional de Genética, Dr. Hajime Okada
Laboratorio de terapia con células madre, Instituto de Biociencia Cuantitativa, Universidad de Tokio, Profesor asociado del proyecto, Dr. Tohru Itoh
Productos usados para esta aplicación
se ha añadido correctamente a sus marcadores
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
Not Available in Your Country
Sorry, this page is not
available in your country.





