Die Zellteilung ist bei allen Lebewesen ein zentraler Vorgang. Vor jeder Teilung muss die genetische Information der Zelle, die in der DNA gespeichert ist, exakt kopiert werden. Dieser Kopiervorgang wird als DNA-Replikation bezeichnet und seine ordnungsgemäße Regulierung spielt eine entscheidende Rolle für die Gesundheit und das Überleben eines Individuums. Wie Zellen die DNA-Replikation regulieren, um eine hohe Genauigkeit bei der Genomverdoppelung zu erreichen, ist eine der grundlegenden Fragen in der biomedizinischen Forschung. Fehler während der DNA-Replikation können zu einer Instabilität des Genoms führen, die mit schweren Krankheiten wie Krebs in Verbindung gebracht wird. Schätzungen zufolge könnten bis zu zwei Drittel aller Krebserkrankungen auf eine Ansammlung von Fehlern bei der DNA-Replikation zurückzuführen sein.
Lernen Sie eine Wegbereiterin auf dem Gebiet der DNA-Replikation kennen
Wie Zellen ihre DNA verdoppeln, ist auch der Forschungsschwerpunkt von Dr. Hana Polasek-Sedlackova. Im Rahmen eines wissenschaftlichen Forschungspraktikums an der Masaryk-Universität erhielt sie bereits während der Schulzeit erste Einblicke in die Labor- und DNA-Forschung. Seitdem ist sie ein aufsteigender Stern auf ihrem Gebiet. Für ihre Bachelorarbeit erhielt sie den renommierten weltweiten Undergraduate Award in Life Sciences, eine Art „Junior-Nobelpreis“. Nach ihrem Abschluss erhielt sie ein Stipendium der Novo Nordisk Foundation für die Aufnahme in das Copenhagen Bioscience PhD-Programm. Mit diesem Stipendium begann sie als Doktorandin im Labor von Professor Jiri Lukas am Novo Nordisk Foundation Center for Protein Research an der Universität Kopenhagen und setzte dort ihre Postdoc-Ausbildung fort. Ihre Forschungsarbeiten wurden in führenden wissenschaftlichen Zeitschriften veröffentlicht, zum Beispiel in Science and Nature. Seit kurzem leitet sie ihre eigene unabhängige Forschungsgruppe am Institute of Biophysics (IBP) of the Czech Academy of Sciences im tschechischen Brünn.
Einer ihrer Beiträge zur Wissenschaft war die Lösung des MCM-Paradoxons, das seit den 90er Jahren unter Fachleuten diskutiert wird. Es beschrieb scheinbar widersprüchliche Ergebnisse zwischen der Mikroskopie und anderen wissenschaftlichen Methoden, die zur Aufklärung der Vorgänge während der Replikation eingesetzt werden. Diese Widersprüche konnten nun mit Hilfe unserer High-Content-Screening-Station scanR, die Hana Polasek-Sedlackova häufig nutzt, erfolgreich geklärt werden. Wir trafen Hana Polasek-Sedlackova während eines scanR-Anwendertreffens am Institut für Molekulargenetik in Prag und nutzten die Gelegenheit, sie nach ihren Erfahrungen mit dem System und dessen Bedeutung für ihre Forschung zu fragen.
Interview mit Hana Polasek-Sedlackova
Evident: Als Sie Ihre eigene Forschungsgruppe am IBP gründeten, war die scanR High-Content-Screening-Station das erste Mikroskop, das Sie angeschafft haben. Warum haben Sie sich für dieses System entschieden?
Hana Polasek-Sedlackova: Im Lauf der Jahre hatte ich Gelegenheit, mit verschiedenen Mikroskopen zu arbeiten. Meine Arbeit hat mir gezeigt, dass es drei wichtige Aspekte gibt, die es bei der Auswahl eines Mikroskops zu berücksichtigen gibt. Diese Aspekte sind Hardware, Software und Kundenservice. Nach sorgfältiger Abwägung dieser Kriterien bin ich zu dem Schluss gekommen, dass scanR das beste High-Content-Screening-System ist.
Erstens verfügt das scanR System über ein einfaches und anpassbares Hardwaresystem, das an die spezifischen experimentellen Anforderungen des Bedieners angepasst werden kann. Es verfügt über Bildgebungsfunktionen von Weitfeld bis Superauflösung und ermöglicht fortschrittliche Fluoreszenzmikroskopietechniken, die Bildgebung von Lebendzellen und vieles mehr.
Zweitens verfügt das scanR System über eine intuitive und bedienerfreundliche Erfassungs- und Analyseplattform, mit der man in kurzer Zeit qualitativ hochwertige und quantitative Daten erhalten kann. Die Echtzeit-Multiparameteranalyse der erfassten Bilddaten ermöglicht eine sofortige Datenüberprüfung und eine weitere Optimierung der Aufnahmeparameter, zum Beispiel mit der hilfreichen Rescan-Funktion. (Mit der Rescan-Funktion wird ein Schnellscan der Probe mit einer niedrigeren Auflösung durchgeführt, um zunächst die Objekte von Interesse zu identifizieren und diese dann mit einer höheren Auflösung erneut zu scannen).
Drittens bietet Evident einen hervorragenden Kundenservice. Ihre Anwendungsspezialisten für die Mikroskop-Wartung und für Analysesupport sind ausgewiesene Fachleute, die jederzeit bereit sind, einem bei speziellen Anforderungen, Wünschen oder Problemen zu helfen. Dank der Czech Science Foundation und internen Mitteln unseres Instituts waren wir in der Lage, die scanR Technologie für mein neues Institut anzuschaffen.
Messung der DNA-Replikationsdynamik mit dem scanR System. (A) Schematische Darstellung einer menschlichen Zelle mit Vergrößerung einer Replikationsgabel (einer Grundeinheit der DNA-Replikation). Spezifische Fluoreszenzsonden ermöglichen die Untersuchung der MCM-Bindung an die DNA (Halo), der Assemblierung der aktiven CMG-Helikase (GFP) und der DNA-Synthese (EdU). (B) scanR-basierte Multiparameteranalyse von Schlüsselereignissen der DNA-Replikation in menschlichen U2OS-Osteosarkomzellen. (C) Bildergalerien, die die Dynamik von DNA-Replikationsproteinen im Lauf des Zellzyklus veranschaulichen.
Evident: Wie hat das scanR System Ihre Forschungsarbeiten vorangebracht? Können Sie ein Beispiel nennen, bei dem Ihnen das scanR System zu neuen Erkenntnissen verholfen hat?
Hana Polasek-Sedlackova: Das scanR System hat uns dabei unterstützt, grundlegende Entdeckungen auf dem Gebiet der DNA-Replikation zu machen. In meiner Forschung spielte es eine wichtige Rolle bei der Lösung des MCM-Paradoxons, eines lange bestehenden Rätsels über das Verhalten des MCM2-7-Proteinkomplexes. Der „Minichromosome Maintenance”- bzw. MCM-Komplex bildet den Kern der replikativen Helikase, die für die Entwindung der DNA-Doppelhelix verantwortlich ist, um eine genaue Verdoppelung zu gewährleisten. Es gibt zwei allgemein bekannte Fakten, die paradox sind und Wissenschaftler seit Jahren vor ein Rätsel stellen: (1) Warum enthalten Zellen einen enormen Überschuss an inaktiven MCM-Komplexen, wenn nur 5–10 % als aktive replikative Helikasen verwendet werden? (2) Warum ist es bisher nie gelungen, eukaryotische MCM-Komplexe an Replikationsstellen innerhalb einer Zelle mit mikroskopischen Verfahren darzustellen?
Wir haben uns kürzlich erneut mit dieser konzeptionellen Sackgasse befasst und das MCM-Paradoxon schließlich geklärt. Mit Hilfe von CRISPR-Cas9-Genomediting und ScanR High-Content-Imaging waren wir in der Lage, langjährige Herausforderungen bei der Darstellung von DNA-Replikationsgabeln in Lebendzellen aus dem Weg zu räumen, und haben den neuen Homöostase-Weg von DNA-Replikationsursprüngen aufgezeigt, der zum Schutz der Genomintegrität notwendig ist.1-2
Wir fanden heraus, dass der Überschuss an inaktiven MCM-Komplexen als natürliche Pausenstellen für die Replikation (NRPSs) dient, um die Geschwindigkeit der Bewegung der Replikationsgabel zu regulieren und Fehler bei der DNA-Replikation zu minimieren. Mit dieser Eigenschaft stellen NRPSs eine weitere Ebene der Kontrolle der Geschwindigkeit der Bewegung der Replikationsgabel dar, die häufig von Krebszellen genutzt wird, was sie zu einem neuen attraktiven Ziel für die Krebstherapie macht. Derzeit untersucht mein Forschungsteam die Spitze der Regulationswege der DNA-Replikationsursprünge und deren Beitrag zu den pathophysiologischen Zuständen der Entwicklung menschlichen Gewebes.

Die Entdeckung von Proteinvarianten von MCM-Komplexen mittels scanR wirft Licht auf das MCM-Paradoxon. (A) Ein HaloTag-Markierungsprotokoll zur Unterscheidung von MCM-Proteinpools. Die scanR-basierte High-Content-Analyse ergab unterschiedliche Dynamiken der parentalen und naszierenden MCM-Proteinkomplexe während des DNA-Replikationsprogramms. (B) Ein Modell, das die unterschiedliche Funktion von parentalen und naszierenden MCM-Komplexe während der DNA-Replikation beschreibt. Während die parentalen MCM-Komplexe in erster Linie in aktive CMG-Helikasen umgewandelt werden, bleiben die naszierenden MCM-Komplexe weitgehend inaktiv, fungieren aber als natürliche Pausenstellen des Replisoms, die für die Regulierung einer angemessenen Bewegung der Replikationsgabel wichtig sind.
Evident: Für welche Anwendungen und mit welcher Art von Proben verwenden Sie das scanR System?
Hana Polasek-Sedlackova: In unserer Forschung verwenden wir verschiedene Gewebekulturmodelle, die meisten davon von menschlichen Proben. Dazu gehören Krebszelllinien aus verschiedenen Geweben wie Knochen-, Brust-, Eierstock-, Gebärmutterhals- oder Nierengewebe, aber auch ihre nicht krebsartigen Pendants, Stammzellen und Fibroblasten. Gelegentlich verwenden wir in unseren Untersuchungen auch Tierproben. Wir verwenden diese Proben zusammen mit verschiedenen zellbiologischen, biochemischen und genetischen Ansätzen, um neue Proteine oder ganze Regulationswege zu identifizieren, die bei der fehlerfreien Genomverdopplung eine wichtige Rolle spielen. Zudem schauen wir uns an, wie diese neuen Proteine oder Signalwege in Krebszellen reguliert werden und ob dies die Tumorentwicklung begünstigt. Wenn das der Fall ist, können wir unser Wissen nutzen, um neue Ansätze für die Krebstherapie zu entwickeln.
Unsere typische experimentelle Pipeline für die Identifizierung neuer Proteinfaktoren bei der DNA-Replikation beinhaltet die Erstellung von Profilen einer großen Population von Einzelzellen, und das scanR System war in dieser Hinsicht sehr hilfreich. Wenn wir beispielsweise einen neuen Proteinkandidaten identifizieren, regulieren wir ihn zunächst mit verschiedenen Methoden in den Zellen herunter. Dann überprüfen wir schrittweise mithilfe von High-Content-Imaging die Genauigkeit der DNA-Replikation. Der Vorteil der Verwendung von Zellen in unseren Experimenten besteht darin, dass wir den Prozess der Genomduplikation in seiner natürlichen Umgebung beobachten können. Die Komplexität dieses Prozesses kann jedoch auch zu Herausforderungen bei der Dateninterpretation führen.
Daher müssen wir bei der Untersuchung der DNA-Replikation sorgfältig prüfen, wie sich die Deletion eines Gens von Interesse, auf andere zelluläre Prozesse auswirkt, z. B. auf den Zellzyklus, die Reaktion auf DNA-Schäden, die Transkription, die Erhaltung des Chromatins, die Erhaltung bestimmter Chromosomenregionen, die Gesamtstabilität des Genoms usw. Solche unverfälschten und quantitativen Profile großer Populationen von Einzelzellen, die innerhalb eines sehr kurzen Zeitraums gewonnen werden, helfen uns enorm, unsere Daten schnell zu interpretieren und die nächsten Schritte in unsere Forschung zu planen.
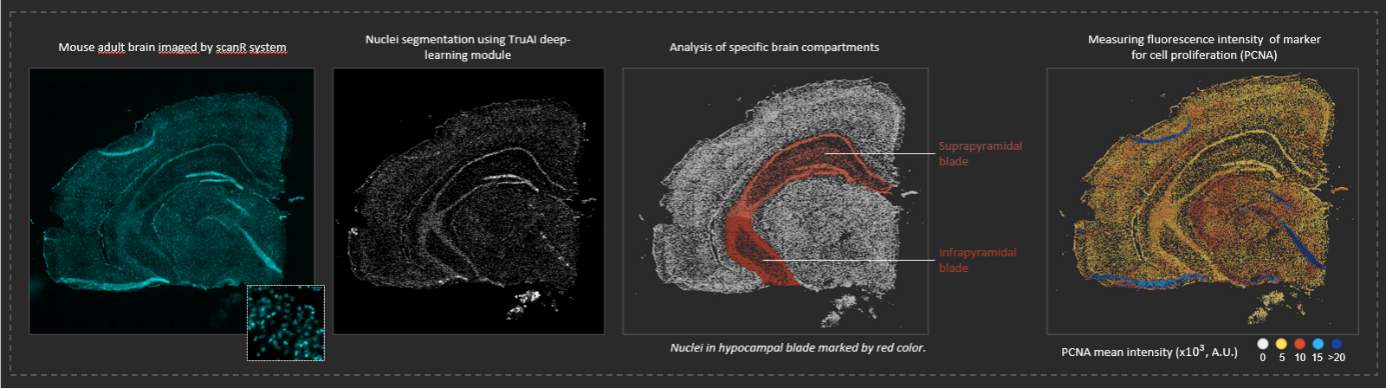
Bildanalyse einzelner Zellen im Gehirn einer erwachsenen Maus mithilfe des TruAI Deep-Learning-Moduls. Die Gehirnprobe wurde von Dr. Jana Krejci (Abteilung für Zellbiologie und Epigenetik am Institut für Biophysik) präpariert.
Früher habe ich Stunden am Mikroskop verbracht, um Bilder manuell aufzunehmen, und danach noch mehr Stunden am Computer, um sie zu analysieren.. Solche Experimente dauerten Monate. Mit dem scanR System ist nun alles in wenigen Stunden erledigt.
Evident: Wie profitieren diese Anwendungen von den Möglichkeiten der High-Content-Analyse und dem bildbasierten Zytometrieansatz des Systems?
Hana Polasek-Sedlackova: Ich bin überzeugt, dass im Bereich der Zellbiologie die Kombination unvoreingenommener quantitativer Informationen mit Bildern eine zuverlässige und präzise Möglichkeit ist, lebenswichtige Zellprozesse darzustellen. Das scanR System bietet hierfür eine schlichte und intelligente Lösung. Wie bereits erwähnt, ermöglicht das scanR System die vollautomatische Erfassung und Analyse von Proben, sodass wir objektive und quantitative Informationen über die zellulären Prozesse erhalten. Wichtig ist, dass man mit nur einem Klick die Bilder zu den jeweiligen Daten direkt aufrufen kann.
Zudem lassen sich Galerien unverfälschter Bilder erstellen, die die Dynamik der betreffenden Proteine während der gesamten Lebensdauer der Zelle veranschaulichen. Durch diese einzigartige Verknüpfung von Bildern und Daten können zelluläre Prozesse in spezifischen Kompartimenten wie dem Zellkern oder dem Zytoplasma sowie in subnukleären Kompartimenten, zum Beispiel an Stellen mit DNA-Reparatur usw., noch genauer untersucht werden. Diese visuelle Ergänzung quantitativer Daten hilft dabei, biologische Fragestellungen zu beantworten.
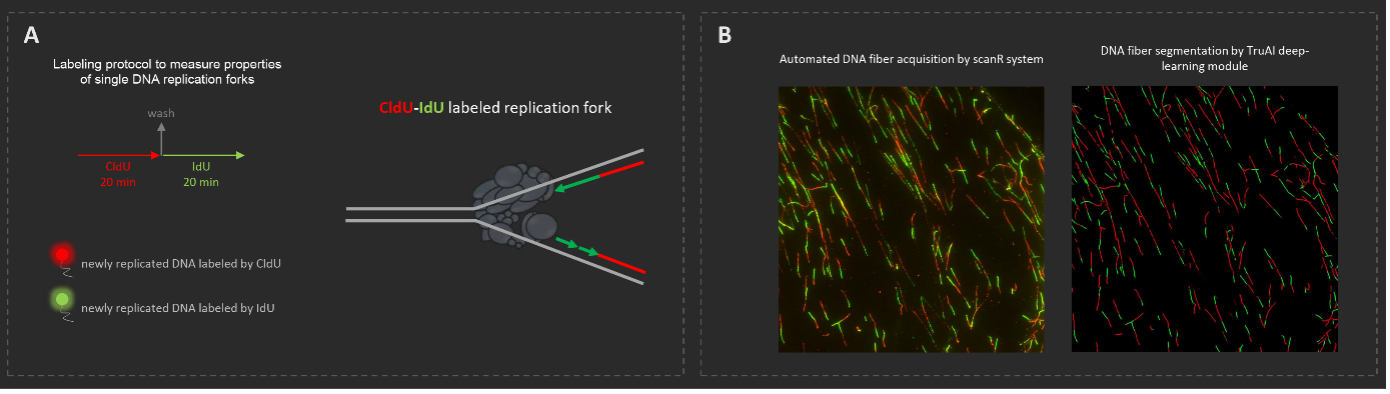
Automatische Erfassung und Analyse von DNA-Fasern durch die Kombination der Systeme scanR und cellSens (Beispiel) (A) Ein CldU-IdU-Markierungsprotokoll zur Visualisierung einzelner Replikationsgabeln. (B) Beispiele für die automatische Erfassung und Segmentierung von DNA-Fasern mit dem scanR System und der cellSens Software.
Evident: Hat das System Ihre Herangehensweise an die Mikroskopie als Werkzeug in der Zellbiologie und Molekularbiologie verändert? Nutzen Sie die TruAI Deep-Learning-Funktionen des Systems?
Hana Polasek-Sedlackova: Ich konnte bereits während meiner Doktorarbeit im Labor von Professor Jiri Lukas erste Erfahrungen mit der High-Content-Bildgebung und dem scanR System machen. Diese revolutionäre Technologie hat meine Sicht auf die Zellbiologie und die Forschung im Allgemeinen völlig verändert. Besonders beeindruckt hat mich die Fähigkeit, eine unvoreingenommene und vollautomatische Analyse zellulärer Prozesse durchzuführen. In Anbetracht der aktuellen Reproduzierbarkeitskrise in der wissenschaftlichen Forschung bin ich der festen Überzeugung, dass man als Forschender Werkzeuge einsetzen sollten, die es einem ermöglichen, Daten auf automatische Weise zu erhalten und zu analysieren und zelluläre Prozesse unverfälscht wiederzugeben.
In der heutigen Ära der künstlichen Intelligenz (KI) hat die Bildanalyse ihre bisherigen Grenzen gesprengt. Das scanR System verfügt nun auch über das TruAI Deep-Learning-Modul, das den Umfang meiner Forschung erweitert hat. Früher habe ich mich hauptsächlich auf die Analyse einzelner Zellen in großen Zellpopulationen kultivierter menschlicher Zelllinien konzentriert. Mit dem KI-Modul können wir aber zum Beispiel sehr präzise Bildanalysen von einzelnen Zellen in Organschnitten durchführen.
Ein weiteres Beispiel dafür, wie wir die Grenzen unserer Bildanalysefunktionen erweitern können, ist die Kombination der scanR und cellSens Systeme. Dies ermöglicht uns die automatische Erfassung und Analyse einzelner Moleküle, wie z. B. von DNA-Replikationsgabeln, die mit der DNA-Faser-Technik hergestellt wurden. Dies sind einige Beispiele für neue Anwendungen, an denen wir derzeit in meinem Labor arbeiten, und wir hoffen, dass diese Werkzeuge bald auch von anderen Forschenden genutzt werden können, um große Entdeckungen im Bereich der Zellbiologie zu machen.
Evident: Was gefällt Ihnen am besten an diesem scanR System? Wie ist die Resonanz unter den wissenschaftlichen Mitarbeitern?
Hana Polasek-Sedlackova: Ich schätze viele Eigenschaften des scanR Systems, aber wenn ich nur eine hervorheben sollte, dann wäre es die einzigartige Verbindung zwischen automatischer Erfassung und Analyse, die es uns ermöglicht, eine Multiparameteranalyse der erfassten Bilder in Echtzeit durchzuführen. Ich war schon als Doktorandin von dieser Funktion beeindruckt, und jedem neuem Benutzer in unserer kleinen scanR-Benutzergemeinschaft an unserem Institut geht es ebenso.
Einer meiner Kollegen brachte es am besten auf den Punkt, als er sagte: „Früher habe ich Stunden am Mikroskop verbracht, um Bilder manuell aufzunehmen, und danach noch mehr Stunden am Computer, um sie zu analysieren. Solche Experimente dauerten Monate. Mit dem scanR System ist jetzt alles in wenigen Stunden erledigt.”
Hier finden Sie weitere Informationen zum scanR System
Unser Dank gilt Dr. Hana Polasek-Sedlackova für den Einblick in ihre Erfahrungen mit der scanR High-Content-Screening-Station. Weitere Informationen zum scanR System und zu unseren anderen Lösungen für die Forschung in den Biowissenschaften erhalten Sie von Evident oder dem zuständigen Evident Vertriebsmitarbeiter.
Foto von Dr. Hana Polasek-Sedlackova von Jana Mensatorova.
Literatur
- Polasek-Sedlackova H, Miller TCR, Krejci J, Rask MB, Lukas J. Solving the MCM paradox by visualizing the scaffold of CMG helicase at active replisomes. Nat Commun. 2022 Oct 14;13(1):6090. doi: 10.1038/s41467-022-33887-5. PMID: 36241664; PMCID: PMC9568601. https://www.nature.com/articles/s41467-022-33887-5
- Sedlackova, H., Rask, M.-B., Gupta, R., Choudhary, C., Somyajit, K., Lukas, J. (2020, October 21). Equilibrium between nascent and parental MCM proteins protects replicating genomes. Nature News. https://www.nature.com/articles/s41586-020-2842-3
Ähnliche Artikel
Vereinfachung der Instanzsegmentierung von Zellen und Zellkernen mithilfe von Deep Learning
Lokalisierung von Hefeproteinen – klassifiziert mit der TruAI Deep-Learning Technologie

