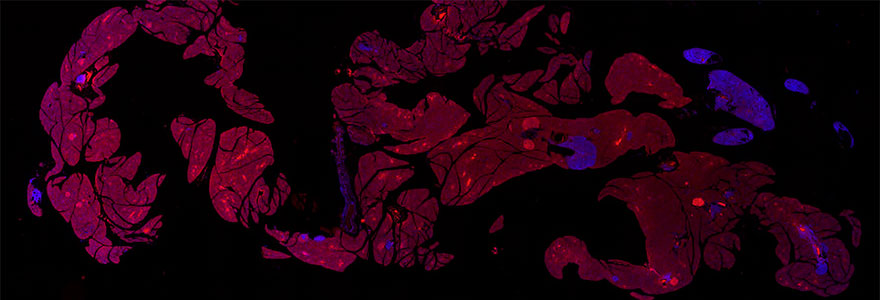
Beschleunigung und Optimierung der Segmentierung und Analyse von Pankreasinseln mit dem VS200 Objektträgerscanner für die Forschung und der Deep-Learning-Lösung TruAI
Um in der Forschung relevante Datenmengen zu erhalten, muss eine große Anzahl von Bildern analysiert werden. Dabei kann es sich um eine quantitative oder qualitative Analyse handeln, die in jedem Fall zuverlässig und unverfälscht sein muss. Je nach morphologischem Untersuchungsbereich kann diese Aufgabe recht aufwändig sein. Um diesen Prozess zu beschleunigen, bietet Olympus eine Deep-Learning-Lösung in Kombination mit der SLIDEVIEW VS200 Objektträgerscannereinheit für die Forschung an. Für den Test dieser Lösung verwendeten wir sie für die Segmentierung und Analyse von Pankreasinseln.
Erkennung und Segmentierung von Pankreasinseln
Die experimentelle Diabetes-Forschung zielt darauf ab, den komplexen Prozess dieser Krankheit besser zu verstehen, um bessere und wirksamere Therapien zu entwickeln. Die Analyse einer großen Anzahl von Pankreasschnitten ist für die Forschung unerlässlich.
Die Bauchspeicheldrüse (Pankreas) hat zwei Hauptfunktionen: Sie produziert Enzyme, um Proteine, Lipide, Kohlenhydrate und Nukleinsäuren in der Nahrung aufzuspalten (exokrine Funktion), und sie produziert die Hormone Insulin und Glukagon, um den Blutzuckerspiegel zu kontrollieren (endokrine Funktion). Bei einer Diabeteserkrankung ist die letztere Funktion gestört. Die sogenannten Langerhans-Inseln – das sind Gruppen von Betazellen in der Bauchspeicheldrüse – spielen bei der Insulinsekretion eine wesentliche Rolle. Insulin hilft bei der Senkung des Blutzuckerspiegels, indem es die Aufnahme des Blutzuckers in die Zellen anderer Gewebe zur Energiegewinnung anregt.
Bei der Untersuchung von Diabetes und anderen Stoffwechselkrankheiten werden der prozentuale Anteil und die relative Position dieser insulinhaltigen Betazellen analysiert. Für die Bestimmung der Morphologie und die Quantifizierung der Langerhans-Inseln der Bauchspeicheldrüse ist die mikroskopische Analyse einer großen Anzahl von Bildern entscheidend.
Experiment mit der SLIDEVIEW VS200 Objektträgerscannereinheit für die Forschung und der Deep-Learning Solution TruAI
Um zu demonstrieren, wie die Olympus Deep-Learning-Lösung TruAI diese Forschungsanwendung verbessern kann, haben wir die Alterungsprozesse in den Langerhans-Inseln von CB57BL/6NTac-Mäusen untersucht, um die Veränderungen in der Morphologie und Funktion der Inseln zu verstehen.
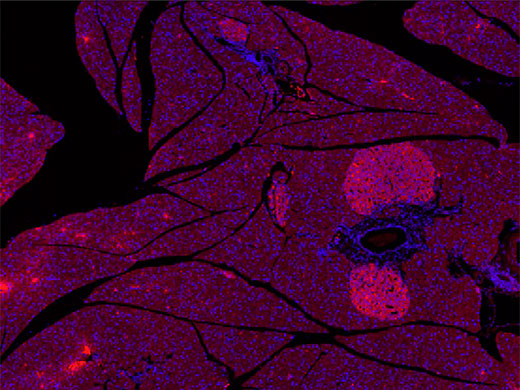
Beispiel für einen Objektträger, der mit der VS200 Objektträgerscannereinheit von Olympus für unser Experiment gescannt wurde. Mit Alexa 594 gefärbter Pankreasschnitt und DAPI-Gewebeschnitte mit freundlicher Genehmigung von Univ.-Prof. Dr. rer. nat. Simone E. Baltrusch vom Institut für Medizinische Biochemie und Molekularbiologie der Universitätsmedizin Rostock in Rostock, Deutschland
Die mit freundlicher Genehmigung von Univ.-Prof. Simone E. Baltrusch und Dr. Cindy Zehm vom Institut für Medizinische Biochemie und Molekularbiologie der Universitätsmedizin Rostock, Deutschland, verwendeten Proben sind Schnitte der Bauchspeicheldrüse von Mäusen, die mit einem Insulin-Antikörper-Färbeprotokoll präpariert wurden. Diese Färbung ermöglicht die Identifizierung von Betazellen innerhalb der Langerhans-Inseln. Diese Zellen gehören zum endokrinen Teil der Bauchspeicheldrüse, der sich in der Zellarchitektur vom exokrinen Teil unterscheidet.
Digitale Objektträger der Mauspankreasproben wurden mit dem SLIDEVIEW VS200 Objektträgerscanner bei 10-facher Vergrößerung in kürzester Zeit aufgenommen. Das Ergebnis waren 40 Bilder, auf denen die Langerhans-Inseln mit dem menschlichen Auge leicht zu identifizieren waren. Die Bilder zeigen deutlich den roten, mit Alexa 594 gefärbten Sekundärantikörper, der an den primären Antikörper für insulinproduzierende Betazellen gebunden ist, im Kontrast zur (blauen) DAPI-Gegenfärbung der Zellkerne.
Herausforderungen bei der automatischen Erkennung und Analyse von Pankreasinseln
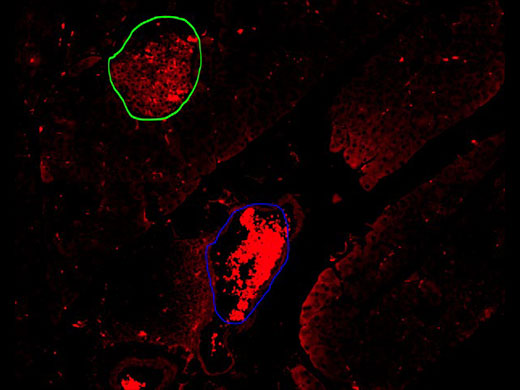
Für die Visualisierung von Pankreasinseln in einer Mauspankreasschnittprobe können spezifische Fluoreszenzantikörper verwendet werden. Für die Analyse dieser Strukturen in gescannten Bildern werden die Inseln normalerweise manuell ausgewählt, was sehr zeitaufwändig ist. Mit herkömmlichen automatischen Segmentierungsmethoden, beispielsweise schwellenwertbasierten Algorithmen, ist es nicht möglich, ausschließlich Pankreasinseln zu erkennen (siehe folgende Abbildung).
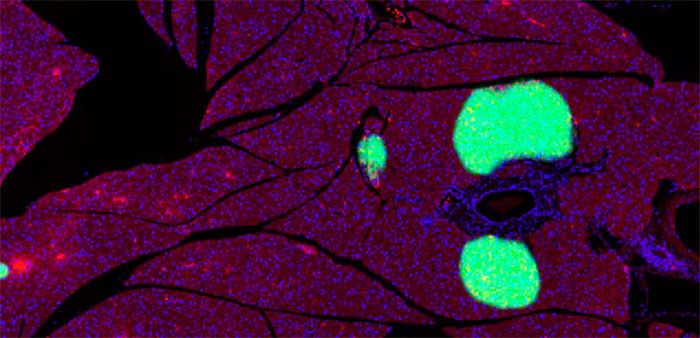
Aufnahme mit 10-facher Vergrößerung; zeigt die Detektion der Inseln mithilfe der konventionellen Schwellenwertmethode in gelb, bei der die Inseln (grüner Kreis) nicht von roten Blutkörperchen (blauer Kreis) unterschieden werden können.
Bei der Betrachtung des Alexa 594-Kanals wurde auch die Autofluoreszenz der Erythrozyten erfasst, was die automatische Unterscheidung des markierten Antikörpers, mit dem die Betazellen auf einer Pankreasinsel (grüner Kreis) gefärbt wurden, von einem Blutgefäß voller Erythrozyten (blauer Kreis) erschwerte. Der VS200 Objektträgerscanner für die Forschung bietet als Zusatz zur VS-Desktop-Software für die Bildanalyse das TruAI-Modul, das eine Deep-Learning-Lösung auf der Basis von konvolutionalen neuronalen Netzen bereitstellt. Diese Netze sind eine Form der selbstlernenden Mikroskopie und eine äußerst leistungsfähige Technologie zur Objektsegmentierung. Dank dieser Technologie lässt sich die Erkennung der Mauspankreasinseln automatisieren. Der Trainingsprozess des neuronalen Netzes wird im nächsten Abschnitt erläutert. |
Pankreasinseln (grüner Kreis); Blutzellen (blauer Kreis) |
Einfacher Workflow zur automatischen Segmentierung und Analyse von Proben mit der Deep-Learning-Lösung TruAI
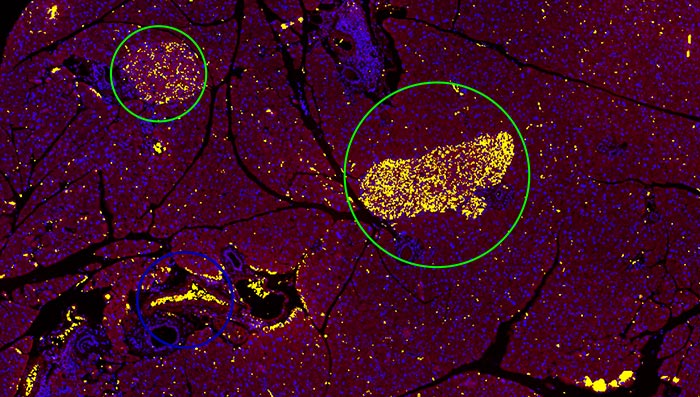
Der erste Schritt hin zur automatischen Analyse besteht darin, der Software annotierte Probenbilder oder Daten des „Grundwissens“ zur Verfügung zu stellen. Diese erfolgte durch manuelle Beschriftung der Inseln von zwölf verschiedenen Mauspankreasproben (grüner Kreis).
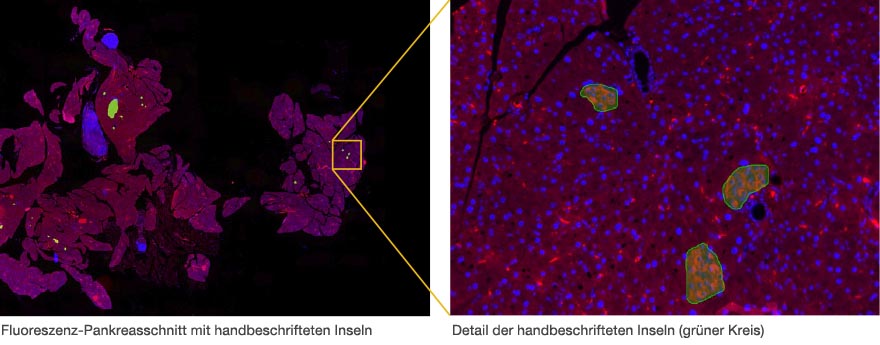
Je mehr Objekte markiert werden, desto besser. Eine höhere Anzahl von Objekten mit unterschiedlicher Intensität, Farbe, Größe und Form macht das neuronale Netz robuster. Das einfach zu bedienende Software-Add-on verfügt über verschiedene Werkzeuge, die die manuelle Beschriftung erleichtern.
Nach der Generierung der Beschriftungsdaten wird im nächsten Schritt das tiefe neuronale Netz (DNN) trainiert. In dieser Phase vergleicht das Netz die Daten des Grundwissens mit seinen eigenen berechneten Daten, bis ein hoher Wahrscheinlichkeitswert erreicht ist. Bei diesen berechneten Daten handelt es sich um eine Form der künstlichen Intelligenz (KI), die das menschliche Gehirn imitiert. Dieses sogenannte DNN lernt, Strukturen zu erkennen und intelligente Entscheidungen zu treffen.
Im letzten Schritt wird das berechnete DNN in diesem Fall auf die restlichen Pankreasbilder angewendet, um die Langerhans-Inseln automatisch zu erkennen und zu trennen.
Ein trainiertes tiefes neuronales Netz der Olympus TruAI Technologie kann auf jede VS-Desktop Station sowie an andere kompatible Olympus Produkte übermittelt und dort verwendet werden.
Dieser automatisierte Prozess zur Segmentierung und Analyse von Pankreasinseln umfasst die folgenden drei einfachen Schritte:
1. Mit der SLIDEVIEW VS200 Objektträgerscannereinheit für die Forschung wird ein neues Bild gescannt.
|
|
2. Das trainierte DNN erkennt und trennt Pankreasinseln.
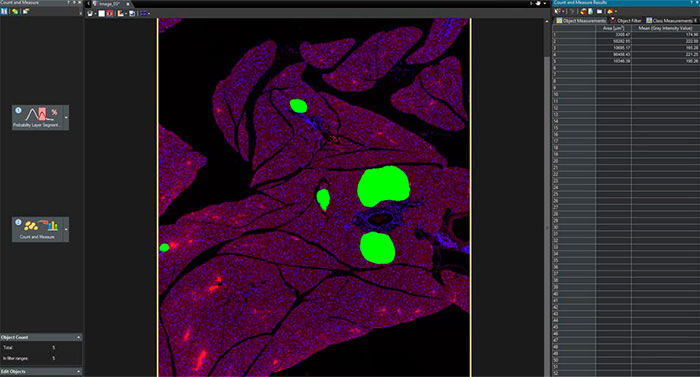
3. Die erkannten Pankreasinseln können segmentiert und zur weiteren Zähl- und Messanalyse des Ausgangsbildes verwendet werden.
Zusammenfassung der Vorteile der SLIDEVIEW VS200 Objektträgerscannereinheit mit der Deep-Learning-Lösung TruAI bei der Erkennung und Segmentierung von Pankreasinseln
Das TruAI-Modul ermöglichte eine einfache Objekterkennung und Segmentierung von Pankreasinseln aus komplexen Bildern mit einer viel höheren Zuverlässigkeit und Genauigkeit als andere bestehende automatische Methoden. Basierend auf diesen Segmentierungsergebnissen können auch weitere Analysen, z. B. Zählungen und Messungen, durchgeführt werden.
Die SLIDEVIEW VS200 Objektträgerscannereinheit kann in Kombination mit der Deep-Learning-Lösung TruAI einen kompletten Arbeitsablauf von der Probenerfassung bis zur präzisen quantitativen Datenanalyse in einer Vielzahl von biologischen Anwendungen auf einer Vielzahl von Bildern, z. B. Zellen und Gewebeproben in Hellfeld und Fluoreszenz, übernehmen.
Die präzise Automatisierung der Bildanalyse nimmt viel mühsame Handarbeit ab und sorgt für mehr Effizienz in der Forschung.
Danksagungen
Dieses Anwendungsbeispiel wurde mit Hilfe von Forschern des Instituts für Medizinische Biochemie und Molekularbiologie, Universitätsmedizin, Universität Rostock, Rostock, Deutschland und Sara Quinones Gonzalez, Product Manager, Olympus Soft Imaging Solutions, in Münster, Deutschland, erstellt:
- Univ.-Prof. Dr. rer. nat. Simone E. Baltrusch, Institut für Medizinische Biochemie und Molekularbiologie, Universitätsmedizin, Universität Rostock, Deutschland
- Dr. Cindy Zehm, Institut für Medizinische Biochemie und Molekularbiologie, Universitätsmedizin, Universität Rostock, Deutschland
- Sara Quinones Gonzalez, Product Manager, Olympus Soft Imaging Solutions GmbH, Münster, Deutschland
Verwendete Produkte
wurde erfolgreich zu Ihren Lesezeichen hinzugefügt
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
Not Available in Your Country
Sorry, this page is not
available in your country.