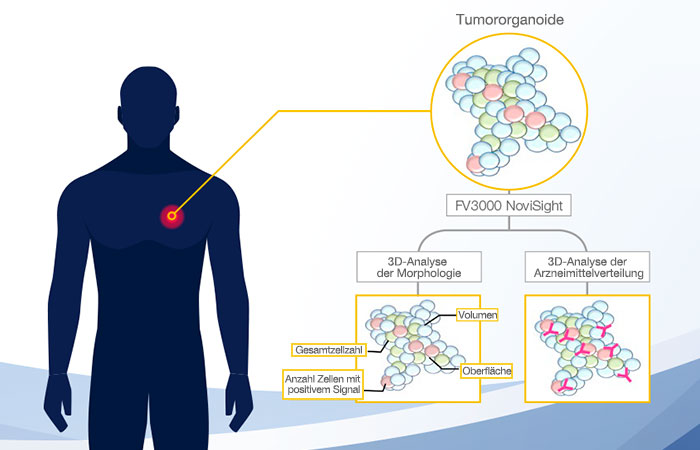
3D-Analyse der von Patienten stammenden Tumororganoide
Die konfokalen Bilder der von Patienten stammenden Organoide (patient-derived tumor organoids, PDO) wurden mit der NoviSight 3D-Software quantitativ analysiert, um ihre morphologischen Eigenschaften und die Antikörperbindung von gegen Molekülziele gerichteten Arzneimitteln zu beurteilen.
Einleitung
3D-Gewebemodelle werden in der Arzneimittelforschung gerne eingesetzt, da sie die In-vivo-Mikroumgebung nachbilden. PDO zählen zu den nützlichsten Tools, die zur Simulation des Ansprechens von Patienten auf Arzneimittel eingesetzt werden können. Tatsächlich hat eine aktuelle Studie ergeben, dass die phänotypischen und genotypischen Profile von PDO eine hohe Ähnlichkeit mit denen des ursprünglichen Patiententumors aufweisen. Noch wichtiger ist, dass sich anhand von PDO die Arzneimittelreaktivität mit einer Genauigkeit von mehr als 80 % vorhersagen ließ.
Mit den Tumororganoiden von Fukushima-Patienten (F-PDO), dem konfokalen FLUOVIEW FV3000 Laser-Scanning-Mikroskop von Olympus und der NoviSight 3D-Analysesoftware haben wir einen neuartigen Arbeitsablauf für die Untersuchung der PDO-Morphologie und die Medikamentenverteilung entwickelt, den wir nun anbieten können.
Grafische Zusammenfassung
Vorteile
- Imaging und Analyse von PDO unter Beibehaltung der in ihrer 3D-Struktur enthaltenen Informationen.
- NoviSight Software erkennt Objekte in 3D-Bildern, klassifiziert diese und liefert statistische Daten.
Methoden
Vorbereitung der Zellen
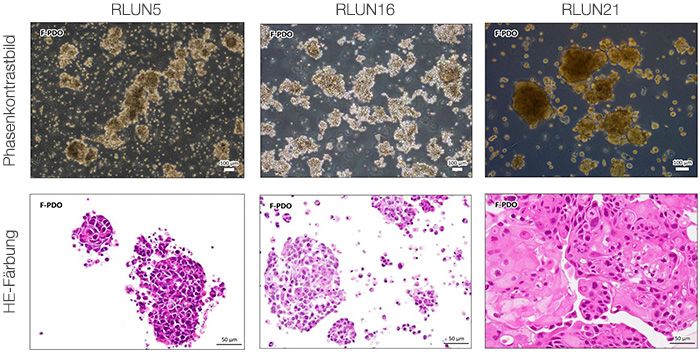
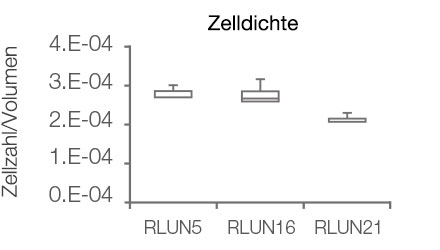
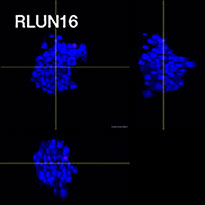
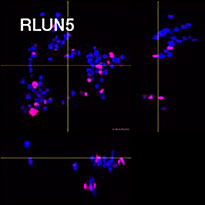
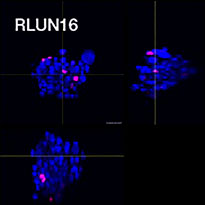
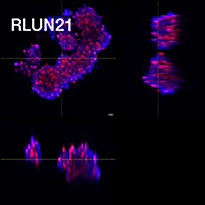
Zur Gewinnung der Zellen wurden drei Typen von Lungen-F-PDO verwendet (RLUN5, RLUN16, and RLUN21). RLUN5 wurde aus einem adenosquamösen Karzinom, RLUN16 und RLUN21 aus einem squamösen Karzinom gezüchtet. RLUN21 bildet große Zellaggregate und weist ein größeres Zytoplasma auf als die anderen beiden (Abb. 1).
Die F-PDO wurden zentrifugiert, aufgenommen und über Nacht mit 4 % Paraformaldehyd fixiert. Erbitux, ein Inhibitor des Rezeptors für den epidermalen Wachstumsfaktor (EGFR), und Herceptin, ein Inhibitor des Rezeptors für den humanen epidermalen Wachstumsfaktor 2 (HER2), wurden mit HiLyte Fluor 555 (Chemical Dojin) markiert und drei Stunden lang mit den F-PDO inkubiert.
Immunfärbung und Klärung
Es wurde das Protokoll für die Immunfärbung nach Hama et al. mit einigen Modifikationen verwendet (Abb. 2). Jedes F-PDO wurde über Nacht mit einem Ki67-Antikörper inkubiert (Abcam, 16667) und mit AbScale Lösung (0,33 M Urea und 0,2 % Triton-X100) verdünnt. Nach Waschen mit AbScale wurden die F-PDO drei Stunden lang mit einem zweiten Antikörper inkubiert und mit AbScale verdünnt. Nach Waschen mit AbScale Spüllösung für eine Stunde wurden die F-PDO erneut mit 4 % Paraformaldehyd fixiert. Anschließend wurden sie mit phosphatgepufferter Salzlösung (PBS) gewaschen und über Nacht mit dem Klärungsreagenz ScaleS4 und DAPI inkubiert. Die geklärten F-PDO wurden dann mit dem konfokalen FV3000 Laser-Scanning-Mikroskop von Olympus aufgenommen.
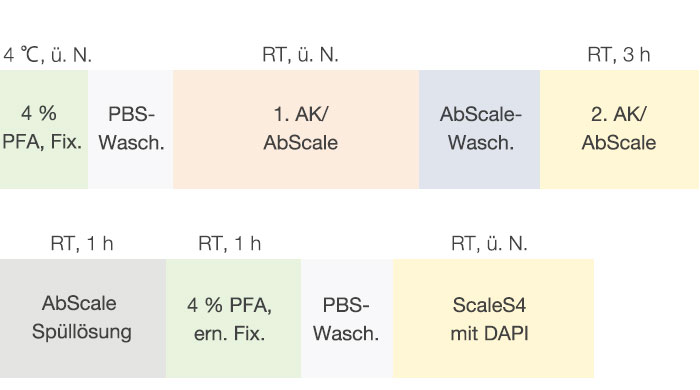
Abbildung 2 Ablauf der Immunfärbung und Klärung von F-PDO
Bildaufnahme und Analyse
Wir verwendeten 10x oder 30x Vergrößerung und 2 µm Z-Abstand für die Aufnahme von Bildern der geklärten F-PDO für die 3D-Analyse.
Ein geeigneter Z-Abstand ist wichtig für eine genaue 3D-Analyse der Zellen. Nach Importieren der Bilder in die NoviSight Software wurde eine 3D-Rekonstruktion vorgenommen. Die NoviSight Software kann 3D-Objekte beispielsweise Zellkerne erkennen und sie in quantitative Daten konvertieren.
Ergebnisse
3D-Quantifizierung der F-PDO-Morphologie
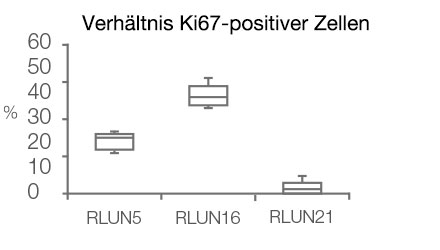
Die NoviSight Software ermöglichte die Quantifizierung von PDO mithilfe mehrerer Z-Ebenen-Bilder. Die Anfärbung der Zellkerne ermöglichte die Erfassung der gesamten Struktur durch die Software und die Erkennung einzelner Zellen (Abb. 3A). Außerdem wurden die verschiedenen erkannten Objekte auf der Kurve als Einzel-Plots hinzugefügt. Wenn die Software alle Zellkerne als Objekt erkannte, wurden Ki67-positive Zellen sowohl grafisch als auch dreidimensional durch Gating von Plots mit hoher Ki67-Identität extrahiert (Abb. 3B). Anschließend wurden unter Verwendung dieser Methoden Zellzahl, Volumen, Zelldichte und das Verhältnis Ki67-positiver Zellen der F-PDO quantifiziert (Abb. 3C). Es stellte sich heraus, dass das Verhältnis Ki67-positver Zellen bei RLUN21 niedriger war als bei den beiden anderen F-PDO. Darüber hinaus könnte das größere Zytoplasma der Grund für die geringen Zelldichte gewesen sein.
A | |||
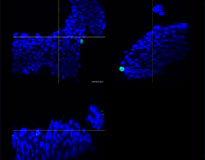 Ursprüngliches RLUN21 | 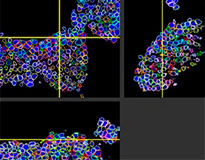 Zellkernerkennung | 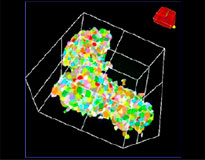 Zellkernerkennung, Volumenansicht | 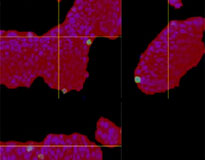 RLUN21 |
B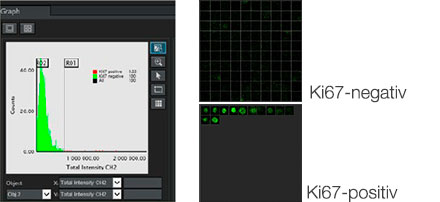 | C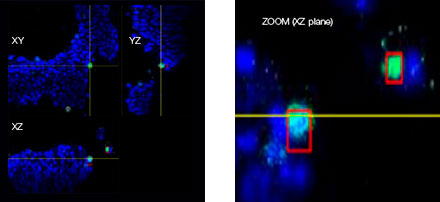 |
D
| RLUN5 | RLUN16 | RLUN21 | |
| Zellzahl | 926 (±719) | 294 (±162) | 2595 (±1480) |
| Volumen (Voxel) | 3.35.E+06 (±3.26.E+06) | 1.08.E+06 (±6.85.E+05) | 1.13.E+07 (±9.12.E+06) |
 |  |
| Abbildung 3 3D-Quantifizierung der Morphologie von F-PDO |
A. Ursprüngliche 3 Seitenansichten von RLUN21, Zellkernerkennung mit NoviSight 3D (3 Seitenansichten und Volumenansicht).
B. Ki67-positive/-negative Zellen wurden nach Ki67-Intensität sortiert. Die NoviSight Software kann sortierte Zellen in einer „Galerieansicht“ anzeigen.
C. Klassifizierte Ki67-positive Zellen können in 3D mit einem roten Kreis angezeigt werden.
D. Quantitative Daten zu den F-PDO.
Analyse der 3D-Verteilung von Arzneimitteln
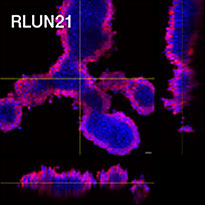
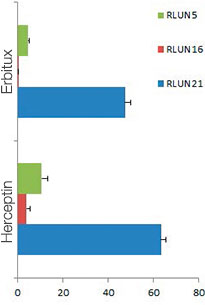
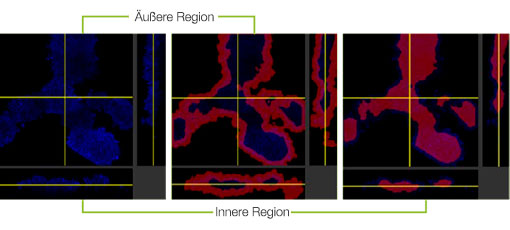
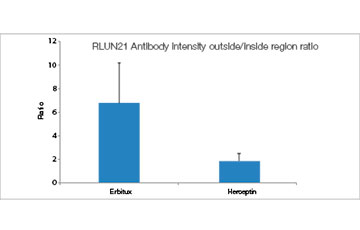
Wurde mit HyLight-555 markiertes Erbitux oder Herceptin zu den einzelnen F-PDO gegeben, band Erbitux homogen an die Oberfläche von RLUN21, Herceptin dagegen band inhomogen an RLUN21. Mit dem Volumenerkennungsmodul der NoviSight Software wurde das Antikörperarzneimittel-positive Volumenverhältnis quantifiziert (Abb. 4A). Anschließend wurde mit der NoviSight Software die Zielregion der Analyse modifiziert und der Unterschied zwischen den Bindungsmustern quantifiziert. Zu diesem Zweck wurden zwei Zielregionen erstellt: eine Volumenregion im Bereich der Oberfläche mit auf einen bestimmten Prozentsatz reduziertem Volumen und eine umrissene Volumenregion beliebiger Breite. Die Färbeintensität der Antikörpermedikamente innerhalb der Volumina wurde berechnet. Zwischen dem reduzierten und dem umrissenen Volumen ließ sich für Erbitux ein großer quantitativer Unterschied, für Herceptin dagegen nur ein geringer Unterschied feststellen. Dies bedeutet, das Erbitux stark an die umrissene Region von RLUN21 band, Herceptin dagegen band generell an RLUN21. Diese Methode erlaubte die quantitative Darstellung der Verteilung der beiden Antikörpermedikamente.
A
 |
 |
 | Erkennung der Antikörpermedikamente 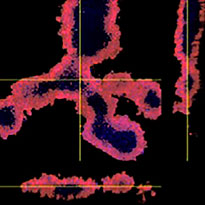 |
 |
 |
 | Erkennung der Antikörpermedikamente 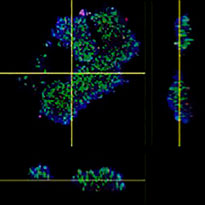 |
Antikörper-positives Volumen (%)
B
 |  |
| Abbildung 4 3D-Quantifizierung der Antikörpermedikament-Verteilung |
A. 3 Seitenansichten von mit Erbitux oder Herceptin angefärbten F-PDO und Volumenerkennung der Bindung der Antikörpermedikamente mit NoviSight. Die Abbildung zeigt Antikörperbindung im gesamten Volumen.
B. NoviSight Modul zur Modifizierung von Zielregionen. Mit NoviSight kann die Zielregion der Analyse modifiziert werden, sodass quantitative Daten aus der eingegrenzten Region erfasst werden können.
Schlussfolgerung
In dieser Studie wurde gezeigt, dass mit der NoviSight Software anhand von Bildern, die mit dem konfokalen FV3000 Laser-Scanning-Mikroskop von Olympus aufgenommen wurden, eine quantitative morphologische und pharmakologische Analyse von PDO durchgeführt werden kann. Durch Kombination der hochauflösenden Bilder des FV3000 Mikroskops mit der hohen Erkennungsgenauigkeit der NoviSight Software lassen sich zuverlässigere quantitative Resultate erhalten.
Literaturnachweis
- Vlachogiannis G et al., (2018) Patient-derived organoids model treatment response of metastatic gastrointestinal cancers. Science. 359(6378):920-926. doi: 10.1126/science.aao2774
- Takahashi et al., (2019) An In Vitro System for Evaluating Molecular Targeted Drugs Using Lung Patient-Derived Tumor Organoids. Cells. 8(5), 481. doi:/10.3390
- Tamura H, et al. Evaluation of anticancer agents using patient-derived tumor organoids characteristically similar to source tissues. Oncol Rep.,40, 635-646, 2018
- Hama et al., (2016) Deep Imaging of Cleared Brain by Confocal Laser-Scanning Microscopy. Nature Protocol Exchange. doi:/10.1038 Mayu
Autoren
Mayu Ogawa (Olympus)
Danksagung
Diese Studie wurde unterstützt durch Professor Takagi von der Medizinischen Universität Fukushima. Weitere Informationen über F-PDO (von Fukushima-Patienten stammende Organoide) finden sich unter https://www.fmu.ac.jp/home/trc/en/contract-research-provision/f-pdo/
Verwendete Produkte
wurde erfolgreich zu Ihren Lesezeichen hinzugefügt
Maximum Compare Limit of 5 Items
Please adjust your selection to be no more than 5 items to compare at once
Not Available in Your Country
Sorry, this page is not
available in your country.